Обработка данных секвенирования по Сэнгеру
1. Сборка консенсуса по хроматограммам
Данные
Все файлы были получены с помощью программы UGENE. Для сборки референса была взята прямая последовательность и последовательность, обратно комплементарная обратной. По ним автоматически был построен референс, после чего в обе последовательности вручную были внесены исправления в соответствии с хроматограммой. По исправленным последовательностям был построен консенсус. Выравнивание в UGENE осуществлялось тоже автоматически.
Исходные файлы: 36_F.ab1, 36_R.ab1.
Выравнивание прямой, обратной последовательностей и референса: ref_aln.fasta.
Исправленные последовательности: 36_F_verif.fasta, 36_R_verif.fasta.
Выравнивание на консенсусную последовательность: cons_aln.fasta.
Консенсусная последовательность: cons.fasta.
Проблемные нуклеотиды
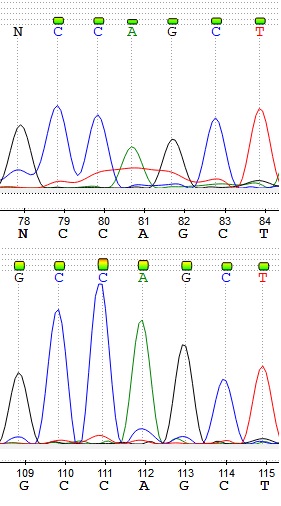
В тексте ниже нуклеотиды нумеруются в соответствии с их номерами в консенсусе, они же равны номерам в обратной последовательности. На картинках хроматограмма для прямой последовательности находится сверху, для обратной – снизу.
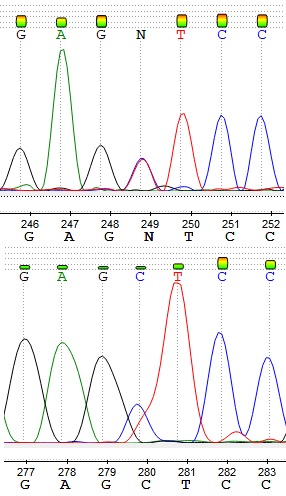
Примером неправильного определения нуклеотида программой может служить 280 нуклеотид: в обратной последовательности программа определяет его как цитозин. Но на хроматограмме прямой последовательности можно увидеть явный полиморфизм: возможен как цитозин, так и тимин. N и C в прямой и обратной последовательностях соответственно заменены на y.
Ещё один пример полиморфизма - 100 нуклеотид, здесь программа в обеих последовательностях не различила M и определила нуклеотид как N. Исправлено на m.
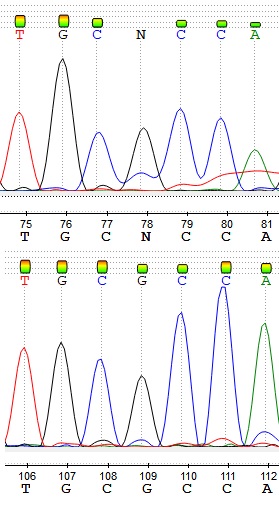
В качестве примера влияния высокого шума на работу программы можно привести 109 нуклеотид: незначительный пик цитозина помешал программе определить гуанин в прямой последовательности. Визуально даже в прямой цепи полиморизм не просматривается, а в обратной шум ещё меньше. N исправлено на g.
В прямой последовательности можно ошибочно определить 112 аденин как полиморфизм W. Однако видно, что высокий уровень сигнала T обусловлен не отдельным пиком, а пятном. Программа в данном случае тоже не ошибается.
Характеристика хроматограммы
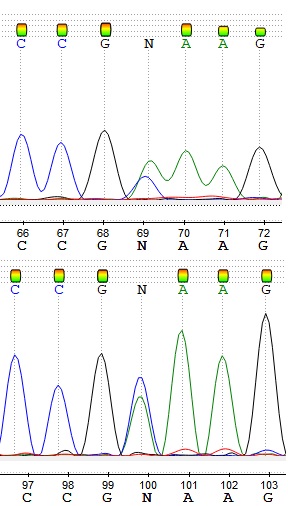
В целом, хроматограмма достаточно качественная, большинство проблем с определением нуклеотидов вызваны полиморфизмами. Расстояния между соседними пиками практически не различаются. Несовпадений нуклеотидов в прямой и обратно комплементарной обратной последователности, построенных по хроматограммам, тоже нет (вышесказанное справедливо для всей последовательности кроме нечитаемых концов). Интересно, что в обоих последовательностх нечитаемые участки есть только на одном конце: на 5'-конце прямой и 3'-конце обратной по 18 нечитаемых нуклеотидов определяются как визуально, так и автоматически. На 5'-конце обратной последовательности все нуклеотиды читаются и автоматически, и визуально. На 3'-конце прямой последовательности программа не может определить 4 нуклеотида, однако визуально это можно сделать. Однако ухудшение качества хроматограммы заметно на обоих концах последовательностей.
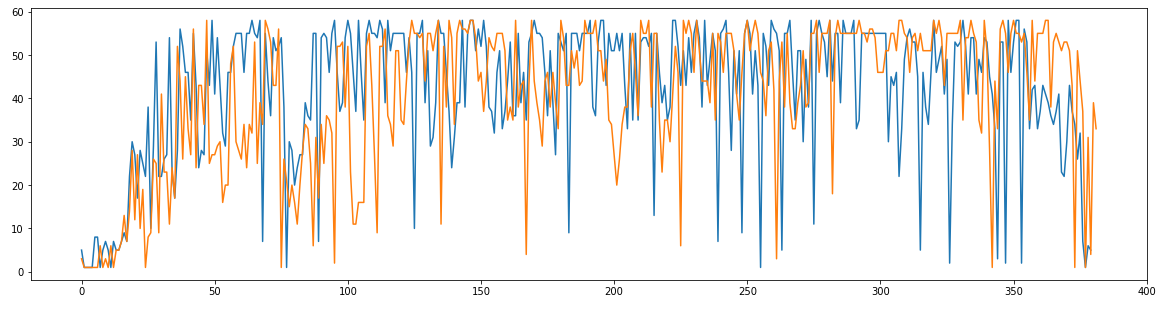
В обоих последовательностях сигнал в среднем в 10 раз выше шума. Примерно за 40 нуклеотидов до нечитаемых концов шум начинает постепенно возрастать, достигая в нечитаемых концах 80%-ой силы от силы сигнала. Сила сигнала вдоль последовательности не изменяется, однако стоит отметить, что на хроматограмме обратной последовательности силы сигнала и шума приблизительно в 2 раза выше, чем на хроматограмме прямой. Качество прочтения отражено на графике ниже. Он получен с помощью следующего кода: code.txt.
2. Пример нечитаемого фрагмента хроматограммы
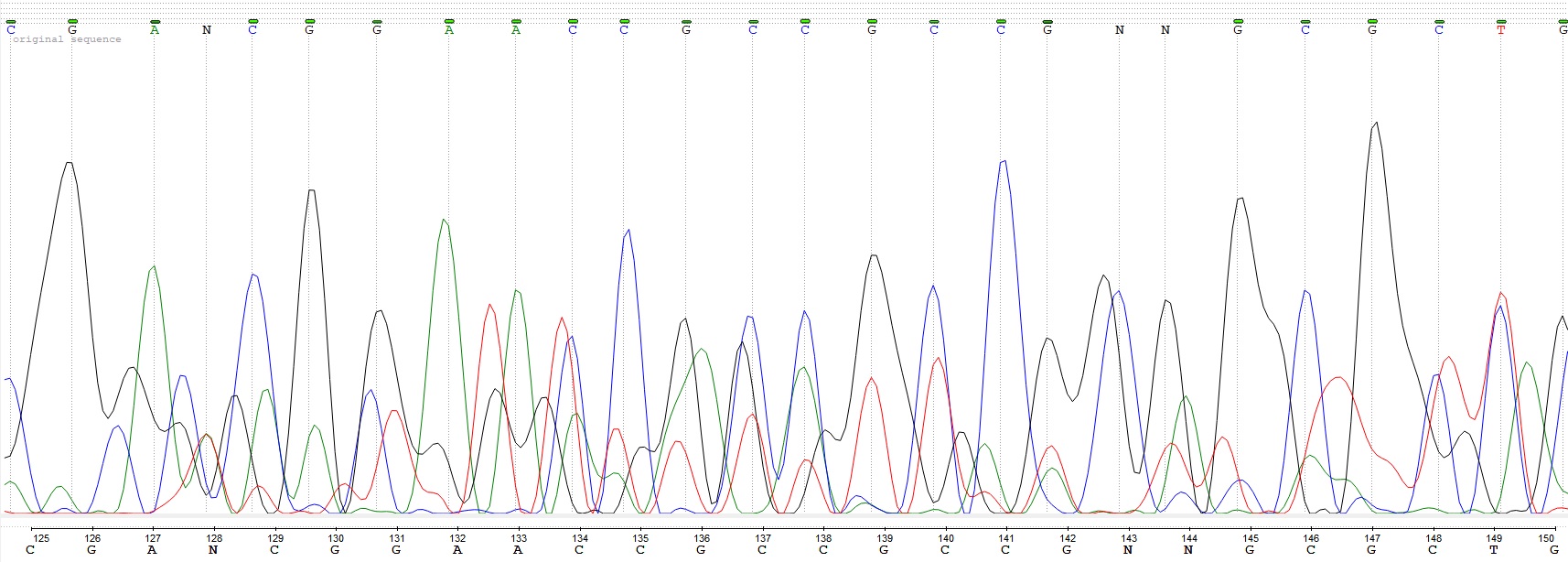
Достаточно содержательным и бессмысленным для описания оказался файл kamp3_18SIII_R_F04_WSBS-Seq-1-08-15.ab1 из директории bad на диске P. Здесь рассмотрен его участок со 125 по 150 нуклеотид. Невозможность прочтения обусловлена наличием нескольких пиков на каждом нуклеотиде, а также тем, что расстояния между соседими пиками достаточно различаются (максимальное различие - примерно в 2 раза).