Нуклеотидный BLAST
1. Определение функции и таксономии нуклеотидной последовательности
В результате расшифровки хроматограммы был получен консенсус для исследуемой нуклеотидной последовательности. Для определения её функции и таксономического положения организма, геному которого она принадлежит, был выбран алгоритм blastn, так как последовательность неизвестная, и нужно найти хоть сколько-нибудь на неё похожие последовательности. По этой же причине поиск производился по самой крупной из баз данных: nr/nt. Для большей точности параметр word size брался равным 7 (минимальное возможное значение), все остальные параметры брались по умолчанию.
blastn выдал много находок с высоким процентом сходства, поэтому были рассмотрены все выравнивания.
Исследуемая последовательность почти наверняка принадлежит гену гистона H3. На это указывает как то, что большинство находок представлены этим геном, так и то, что они принадлежат разным организмам, для которых наименьшим общим рангом таксона является лишь отряд. Последний факт указывает на медленную эволюцию последовательности, что согласуется с предположением о её принадлежности к гену гистона H3, который является достаточно консервативным.
Как было описано выше, 18 находок с наибольшими процентами идентичности принадлежат разным представителям отряда Dendrochirotida из класса Holothuroidea (Голотурий), поэтому таксономическую принадлежность организма, геному которого принадлежит исследуемая последовательность можно предположить лишь с точностью до отряда: Dendrochirotida.
2. Поиск генов белков в неаннотированной нуклеотидной последовательности
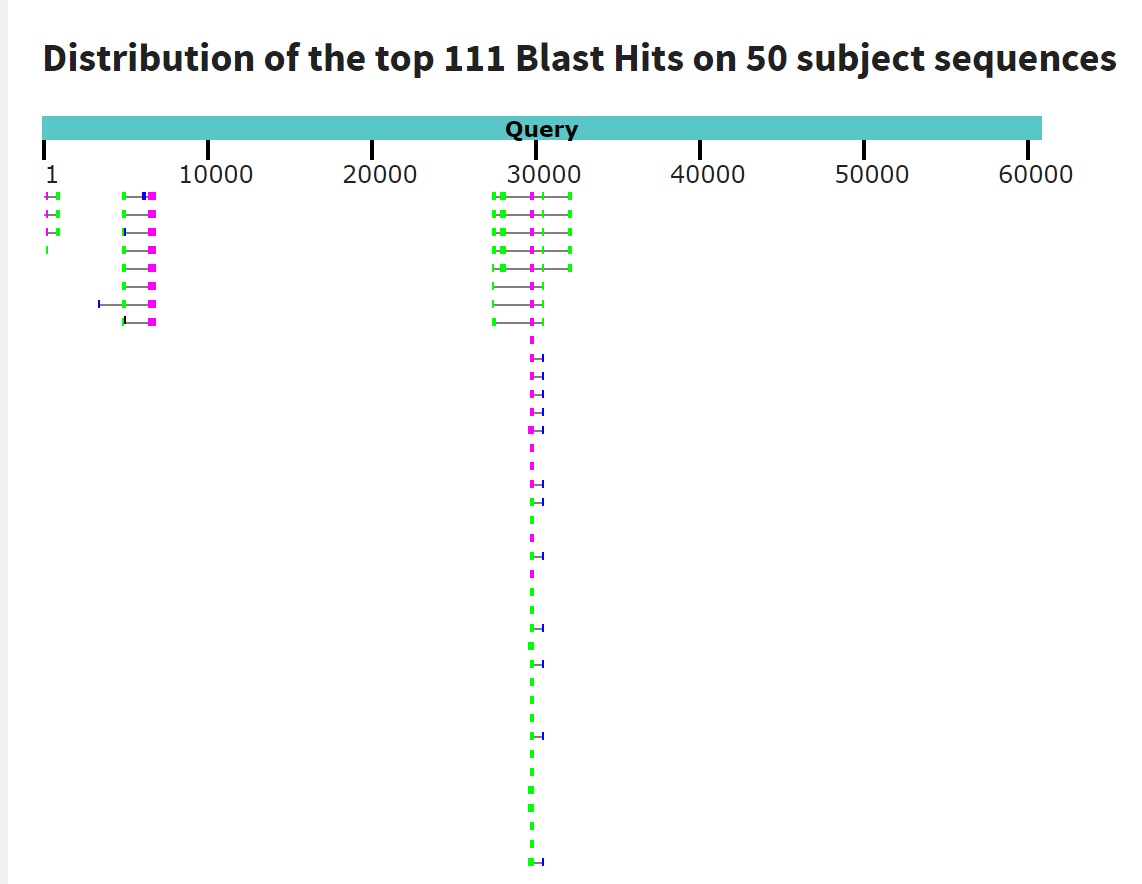
В качестве контига для исследования был выбран контиг FO081457.1 из сборки генома WBcel235 Caenorhabditis elegans. Для определения содержащихся в контиге белок-кодирующих генов был выбран blastx, так как функцию белка, кодируемого исследуемым геном, можно определить по найденным с помощью blastx белкам. Так как необходимо было добыть достоверную информацию, в качестве базы данных был выбран swissprot, для точности word size брался равным 2, остальные параметры - по умолчанию. Была получена следующая выдача: contig_blast.txt Среди находок оказалось несколько белков, все с низким процентом покрытия (это объясняется тем, что размер контига намного превышает размер среднего гена). Судя по распределению находок по контигу (Рис.1), в нём содержится 9 экзонов белок-кодирующих генов. В частности, на 29461-29658 участке контига, вероятно, находится экзон гена тирозинкиназы CSK, которая фосфорилирует src-киназы, таким образом, регулируя рост, дифференциацию, миграцию клеток, а также иммунный ответ.
3. Интепретация карты локального сходства гомологичных хромосом двух бактерий
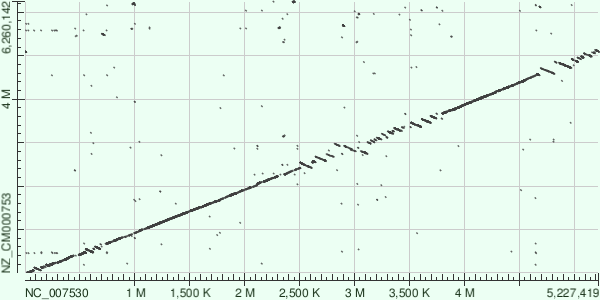
В качестве сравниваемых бактерий были выбраны Bacillus anthracis (NC_007530) и Bacillus thuringiensis (NZ_CM000753). Так как бактерии близкородственны, то был для построения карты локального сходства был выбран megablast. Для уменьшения шума порог на E-value (Expect threshold) был взят равным 10e-95 (заметно меньше шума, чем, например, с 10e-40). На карте локального сходства (Рис.2) видно множество инверсий на участках от 0.55 до 0.75 Mb, от 2.5 до 3.9 Mb и от 4.2 Mb до конца генома. Также можно обнаружить наличие малозаметных инсерций или делеций, проведя прямую y=x: график отклоняется от неё к горизонтальной оси, что свидетельствует о делециях в геноме B. thuringiensis или инсерциях в геноме B. anthracis (по нажатии на изображение перестройки выделяются).