Нуклеотидный пангеном
Выбор бактерий для построения нуклеотидного пангенома
Для построения нуклеотидного пангенома (НПГ) изначально были выбраны геномы 4 штамма Bacillus subtilis. Была выбран именно эта бактерия, так как она достаточно изучена, чтобы среди её штаммов можно было найти несколько представителей с полностью собранной хромосомой. После построения НПГ и его проверки командой CheckPangenome были обнаружены 7 находок, удовлетворяющих критериям мажорных блоков, полученные в результате blastn поиска консенсусов мажорных блоков все-против-всех, а большое количество блоков (почти полностью покрывающее собой геном) признано некачественными, то есть полученная сборка не удовлетворяла критериям НПГ. Поэтому для успешного построения пангенома были взяты 5 более родственных штаммов – из подвида Bacillus subtilis subtilis: таблица со штаммами (Табл.1, входной файл). В полученном НПГ была найдена лишь одна находка blastn, удовлетворяющая критериям мажорных блоков, несколько десятков блоков также были признаны некачественными, но дальнейшую работу с НПГ всё же возможно было провести, поэтому ниже рассмотрен именно этот пангеном.
| Ссылки на геномы | Краткое название генома | Название хромосомы | Тип хромосомы | Полное название штамма | ||
|---|---|---|---|---|---|---|
| all:embl:CP076731.1 | MIZ8 | chr1 | c | Bacillus subtilis subsp. subtilis str. Miz-8 | ||
| all:embl:CP016852.1 | 168G | chr1 | c | Bacillus subtilis subsp. subtilis str. 168G | ||
| all:embl:CP014166.1 | CU1050 | chr1 | c | Bacillus subtilis subsp. subtilis str. CU1050 | ||
| all:embl:CP015975.1 | DELTA6 | chr1 | c | Bacillus subtilis subsp. subtilis str. delta6 | ||
| all:embl:CP051860.2 | 168 | chr1 | c | Bacillus subtilis subsp. subtilis str. 168 | ||
Построение нуклеотидного пангенома с помощью NPG-explorer
Нуклеотидный пангеном строился путём последовательного запуска следующих программ (по ссылкам доступны log-файлы с их выходными потоками для исследуемого (второго) НПГ): Prepare, Examine (пустая выдача), MakePangenome, CheckPangenome, PostProcessing (пустая выдача). Перед запуком MakePangenome в файле npge.conf значение параметра Workers было изменено с -1 на 1, а значение параметра MIN_IDENTITY – с 0.9 на рекомендуемые Examine 0.886.
Стабильное ядро нуклеотидного пангенома
| Число блоков (s-blocks) | 238 |
| Размер нуклеотидного ядра (процент нуклеотидов в ядре от числа нуклеотидов во всех геномах) | 83.63% |
| Процент консервативных колонок в объединённом выравнивании s-блоков | 74.61% |
| Идентичность блоков | 0.9841 |
| Средняя длина фрагментов | 14394 bp (от 99 до 241428) |
Описание крупнейших делеций
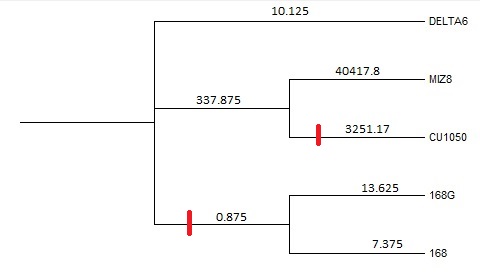
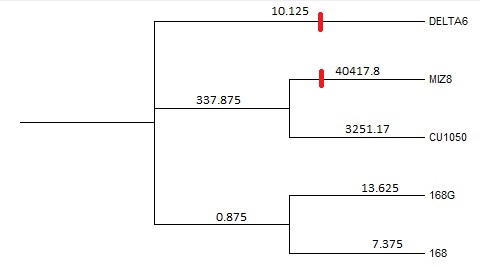
Крупнейшие делеции была найдены путём сортировки содержимого файла pangenome.bi в Excel по длине блока в выравнивании и фильтрации h-блоков (так как именно они являются объектами инсерций и делеций): таблица h-блоков. Крупнейший h-блок: h3x55391 – отсутствовал у штаммов delta6 и Miz-8, а следующий по размеру за ним h2x19724 – delta6, Miz-8 и CU1050. Заметно (Табл.3), что у штамма CU1050 намного больше делеций, чем у остальных, а у штаммов 168 и 168G их почти нет, что также подтверждается расстоянием между этими штаммами и предполагаемым общим предком на филогенетическом дереве (Рис.1 и Рис.2): Miz-8 удалён на порядок дальше, чем CU1050, а 168 и 168G – на порядок меньше. Загадкой остаётся штамм delta6, удалённый так же, как 168G, но при этом имеющий делеций даже больше, чем у CU1050, на порядок более удалённого.
| Штамм | Число делеций |
|---|---|
| 168 | 3 |
| 168G | 0 |
| CU1050 | 65 |
| delta6 | 93 |
| Miz-8 | 200 |
| Название гена (для штамма 168) | Размер (bp) |
|---|---|
| HIR76_18145 polyketide synthase PksJ | 15132 (в блоке только последняя треть) |
| HIR76_18140 polyketide synthase PksL | 13617 |
| HIR76_18135 polyketide synthase PksM | 12789 |
| HIR76_18130 amino acid adenylation domain-containing protein | 16467 |
| HIR76_18125 polyketide synthase dehydratase domain-containing | 7632 |
Блок h3x55391 содержит гены, кодирующие белки, большая часть которых отвечает за синтез поликетидного антибиотика бациллаена (ссылки на записи Uniprot в таблице). Расстояния между генами в блоке малы вплоть до 1 нуклеотида, из чего следует, что блок является частью одного оперона, вероятно, полностью отвечающего за синтез бациллаена. Так как блок отсутствует у штаммов delta6 и Miz-8, то они, судя по всему, не производят этот антибиотик. Может возникнуть вопрос, пропал ли этот блок в результате делеции или появился в результате инсерции: для обоих вариантов достаточно 2 эволюционных события (Рис.1 и Рис.2). В пользу делеций говорит расстояние до общего предка штамма Miz-8: делеция блока у такого далёкого штамма, несущего множество других делеций, более вероятна, чем инсерция у предка маломутировавших 168 и 168G.
Также делецию можно подтвердить, рассмотрев весь удалённый участок: это несколько h-блоков по обе стороны от рассматриваемого, отделённые от него повторами (тоже кодирующими). Видно, что границы удалённых фрагментов у двух штаммов разные: первый (h4x470) и последний (h4x1902) h-блоки в удалённом участке присутствует не у 3, а у 4 штаммов (Рис.3), что тоже говорит о делеции (u-блок у delta6 – видимо независимая инсерция из встроившейся в геном плазмиды – там находится ген устойчивости к хлорамфениколу). Окончательным доказательством делеции является то, что начало первого h-блока и конец предшествующего s-блока кодируют один и тот же белок.
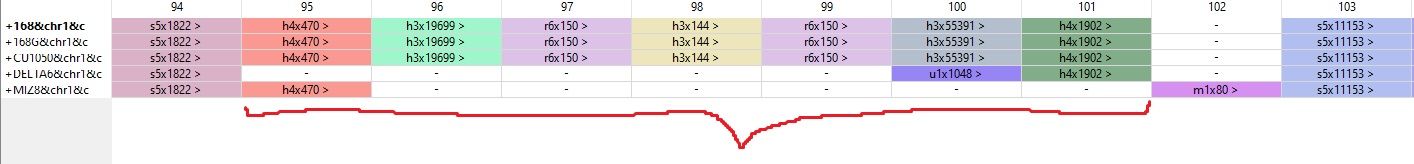
К вышесказанному, вместе с потерей исследуемого фрагмента бактерии лишаются всех генов синтеза бациллаена: начало и конец оперона находятся внутри этого участка (Рис.4).

Блок h2x19724 менее интересен: он содержит около двух десятков генов гипотетических белков. Однако помимо них была найден ген некой сайт-специфичной рекомбиназы. BLASTx показал её наибольшее сходство с интегразой фага phi3T. Это наблюдение явно указывает на появление этого участка у 168 и 168G в результате инсерции у их общего предка после заражения данным фагом.
Стабильное ядро нуклеотидного пангенома
В качестве примера перестановки синтений можно привести перестановку с инверсией небольшого g-блока g5x151 (Рис.5) у Miz-8.

Пример ошибки аннотации гена
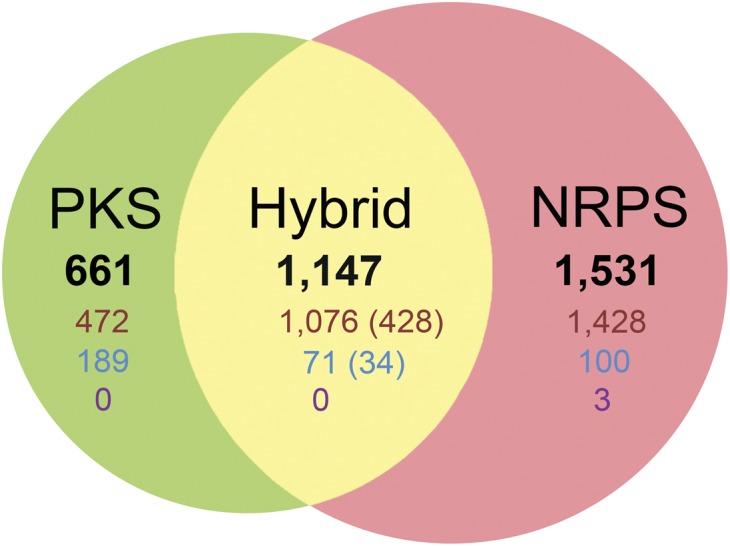
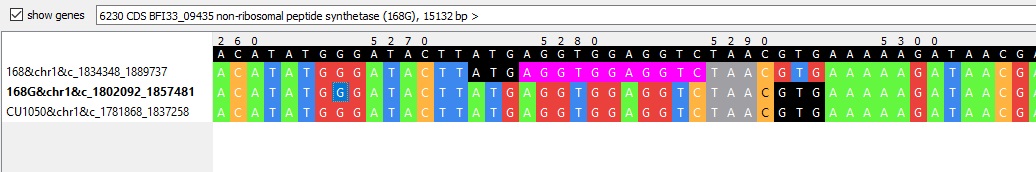
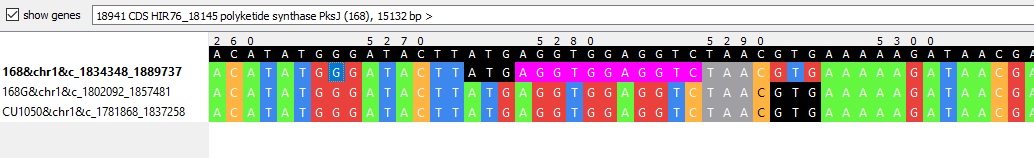
Сразу две ошибки аннотации гена удалось найти в пределах одного гена блока h3x55391: у CU1050 и 168G первый ген блока принимается за синтетазу нерибосомных белков, а у 168 – за синтазу поликетидов PksJ (Рис.7 и Рис.8). Проблема в том, что у бактерий обе активности часто берёт на себя один и тот же фермент (Рис.6) [1], а BLASTx исследуемого гена (точнее, последней его трети, вошедшей в блок) находит оба типа ферментов. Поэтому гены могли назвать по первой из обнаруженных активностей их белка, и фактической ошибки нет. Но формально единообразие названий установить нужно.
Следующая ошибка – неправильное определение конца следующего за вышеописанным гена у штамма 168: NPG отмечает старт-кодон гена до стоп-кодона предыдущего и старт-кодонов его гомологов у других двух штаммов (Рис.7 и Рис.8). Трансляция с этого кодона не может идти в принципе: во-первых, потому что рибосома, идущая со стороны промотора оперона, будет транслировать этот кодон, как продолжение первого белка блока, и перейдёт к синтезу нового белка только после стоп-кодона; и во-вторых, даже если рибосома как-то начнёт транслировать мРНК не с первого гена, а прямо с данного кодона, то в результате сдвига рамки считывания (стоп-кодон в другой рамке) получится белок, совершенно отличный от белков других двух штаммов. Поэтому старт-кодон – GTG после стоп-кодона. Ошибка, видимо, не связана со слабостью старт-кодона GTG: такие же ошибки встречаются в том же блоке с ATG у всех штаммов
Список литературы
- Hao Wang, David P. Fewer, Liisa Holm, Leo Rouhiainen, and Kaarina Sivonen. Atlas of nonribosomal peptide and polyketide biosynthetic pathways reveals common occurrence of nonmodular enzymes. PNAS June 24, 2014 111 (25) 9259-9264. DOI: 10.1073/pnas.1401734111.