Трансмембранные белки
Знакомство с базой данных OPM
В качестве белка с β-листами в трансмембранной части выбран транспортер жирных кислот FadL. Этот белок пронизывает внешнюю мембрану E. coli и переносит длинноцепочечные жирные кислоты из внешней среды в периплазму.
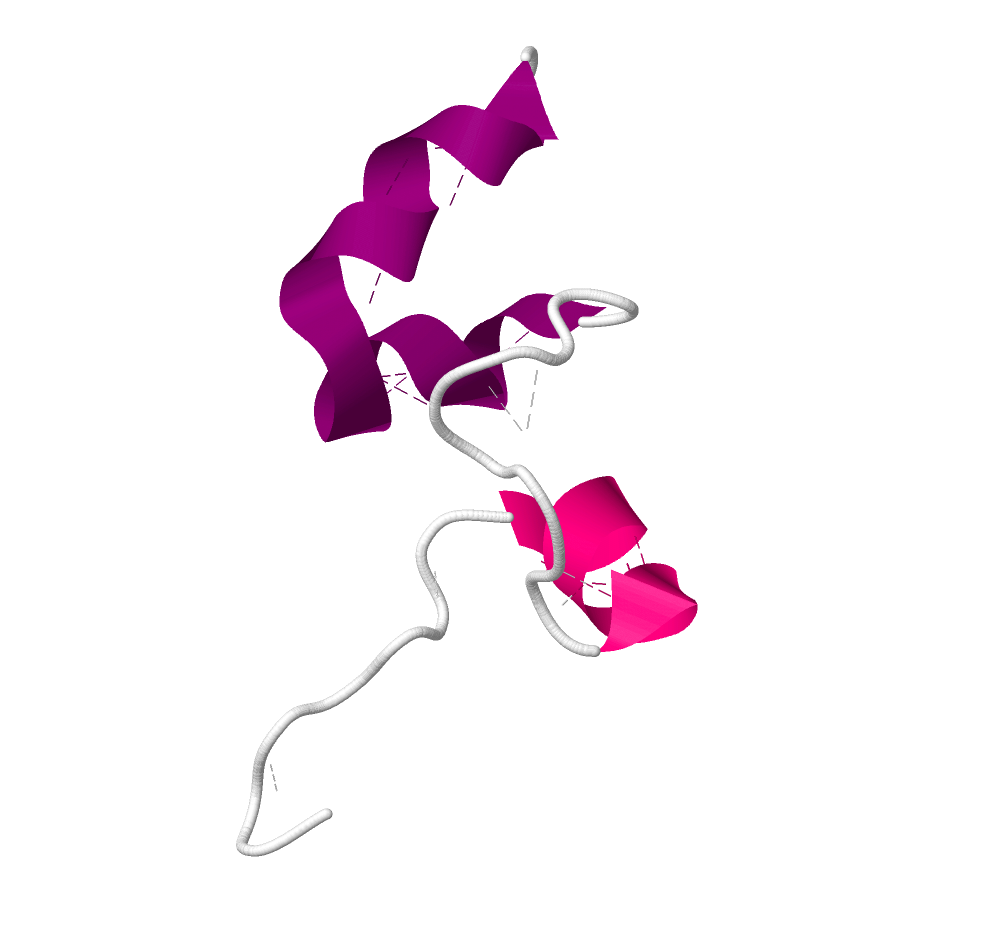
Структура FadL интересна тем, что внутри β-бочонка из 14 β-тяжей находится ещё один трансмембранный участок (4-40 остатки, Рис. 1). Jmol выделяет в нём одну α- и две 310 спирали, неперпендикулярные (благодаря чему в нём умещается 37 остатков, в 4,5 раза больше, чем в одном β-тяже) поверхности мембраны, 2 участка в нём показаны неструктурированными, однако в них тоже можно найти водородные связи, что обеспечивает такому участку возможность встроиться в мембрану (все водородные связи образованы). Интересно, что описанная структура в OPM не включается в число трансмембранных участков
.При измерении толщины гидрофобной части FadL в мембране как расстояния между сторонами мембраны в Jmol (Рис. 2) получилась величина на 1 Å больше заявленной в OPM. Истинное её значение скорее соответствует величине из OPM, так как в Jmol мы получили только верхнюю оценку для неё.
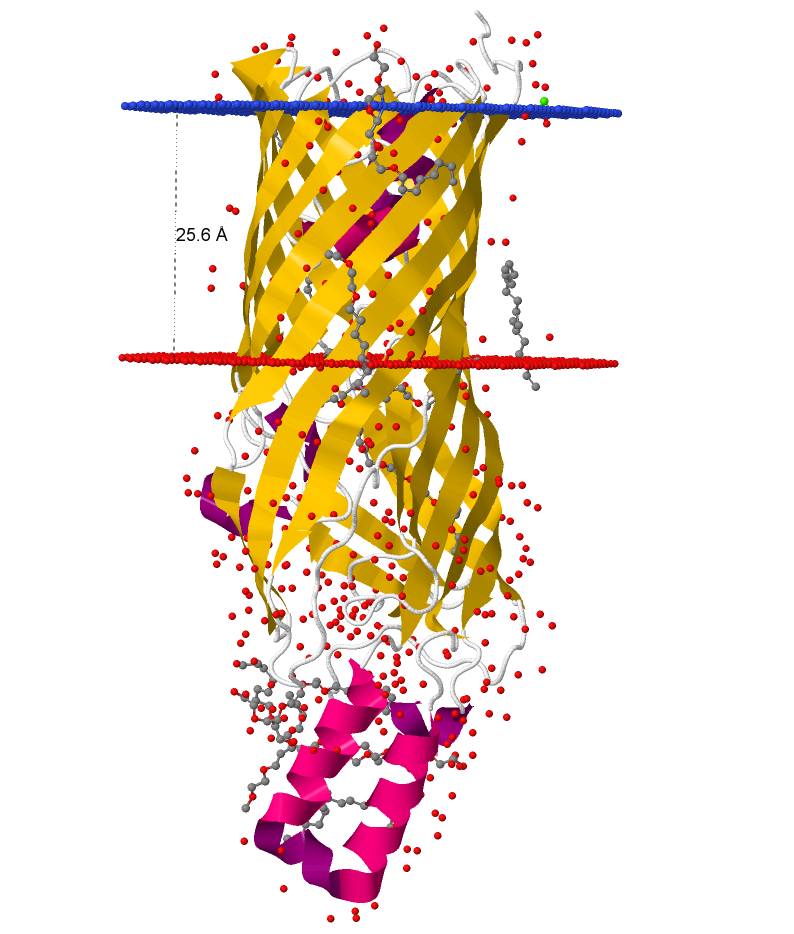
Рисунок 2. Полная структура FadL. p-сторона мембраны снизу, указано расстояние между сторонами мембраны.
| Имя белка | Fatty Acid Transporter FadL (транспортер жирных кислот FadL) |
| PDB ID | 3pgs |
| Uniprot ID | FADL_ECOLI |
| Толщина гидрофобной части белка в мембране | 24,6 Å |
| Координаты трансмембранных участков в OPM | 43-53, 78-87, 94-101, 126-135, 142-148, 208-216, 223-229, 274-281, 289-295, 325-334, 341-348, 367-376, 383-390, 411-421 |
| Среднее количество остатков в одном β-тяже белка | 8 |
| Мембрана | Внешняя мембрана бактерии |
Предсказание положения в мембране с помощью DeepTMHMM
Выданный белок, AMIC_STRR6, – пермеаза транспортной системы олигопептидов AmiC грам-положительной бактерии Streptococcus pneumoniae (SwissProt ID: AMIC_STRR6).
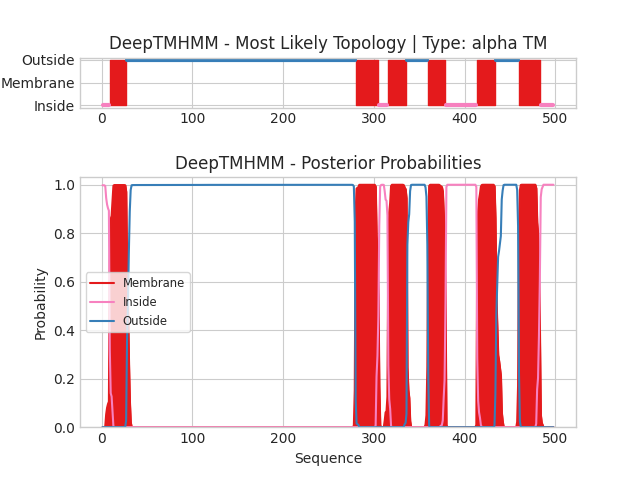
Для неё TMHMM предсказал 6 трансмембранных α-спиралей ("Membrane" на Рис. 3), 4 участка в цитоплазме ("Inside") и 3 снаружи ("Outdide"), N- и C-концы в цитоплазме. Первый участок снаружи мембраны особенно длинный – видимо, является пептид-распознающим доменом.
Входная последовательность: amic.fasta. Текстовая выдача программы: gff3, 3line.
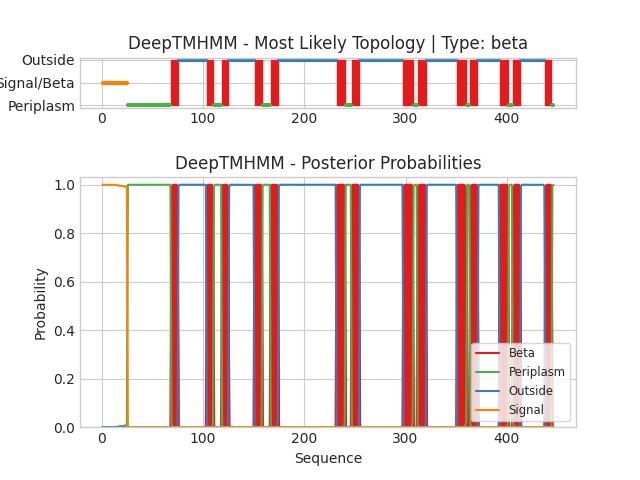
Для FadL TMHMM предсказал 14 трансмембранных β-тяжей ("Beta" на Рис. 4) – однако их координаты сдвинуты вперёд на длину сигнального пептида ("Signal") периплазматической локализации, которой нет в структуре pdb, так как этот пептид отрезается, как только белок попадает в периплазму. Также TMHMM не включает в состав трансмембранных участков 1-2 крайних остатка, включённые в OPM, с обоих концов для каждого участка. Ориентация белка относительно сторон мембраны определена так же, как и в OPM: N- и C-концы в периплазме ("Periplasm"). Однако правильно определить локализацию первого трансмембранного участка также не удалось: TMHMM считает его периплазматическим.
Входная последовательность: FadL.fasta. Текстовая выдача программы: gff3, 3line.
Предсказание положения в мембране с помощью PPM
Предскажем положение в мембране AmiC с помощью другого алгоритма. Входная структура: AmiC.pdb (предсказание AlphaFold).
| Параметр | Значение | Обоснование выбора |
|---|---|---|
| Number of Membranes | 1 | S. pneumoniae – грам-положительная. |
| Type of membrane | Gram-positive bacteria inner membrane | S. pneumoniae – грам-положительная. |
| Allow curvature | no | Вряд ли система транспорта в клетку будет находиться в изогнутых углублениях мембраны (туда будут реже попадать транспортируемые вещества), наличие же выпуклых ворсинок у стрептококков не наблюдалась. Поэтому можно считать мембрану близкой к идеально ровной. |
| Topology (N-ter) | in | Уже предсказали в TMHMM. |
PPM определил те же 6 трансмембранных α-спиралей, что и TMHMM (Рис. 5, Табл. 4). Так же, как и в случае сравнения координат β-тяжей FadL в OPM и предсказании TMHMM, для α-спиралей AmiC TMHMM выдаёт более узкие координаты, однако различие меньше чем было у FadL. Никаких структурных артефактов по сравнению с TMHMM выявлено не было.
| Толщина гидрофобной части белка в мембране | 27.2 ± 1.2 Å |
| Координаты трансмембранных участков | 5-31, 280-302, 316-340, 360-379, 418-440, 460-482 |
| Среднее количество остатков в одной α-спирали белка | 22,5 |
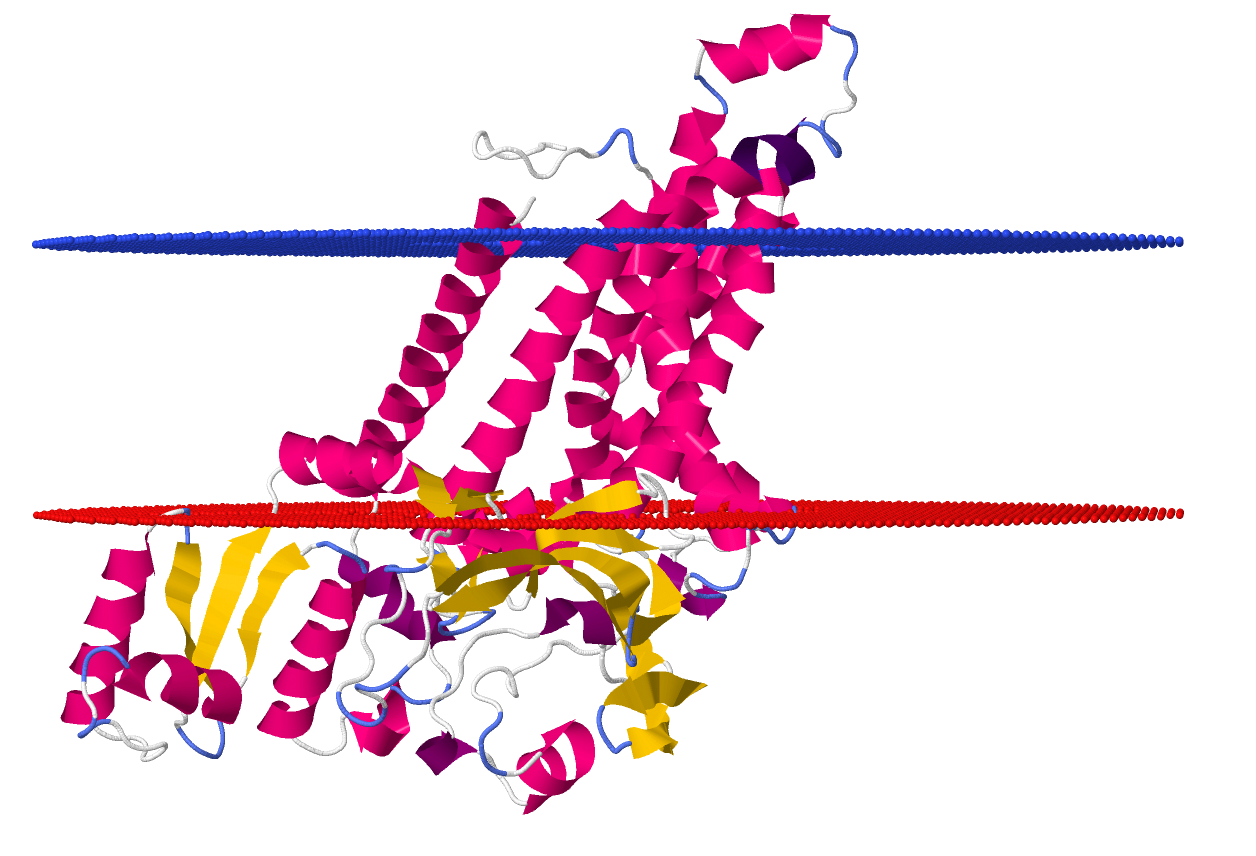
Оценка качества предсказания Alphafold
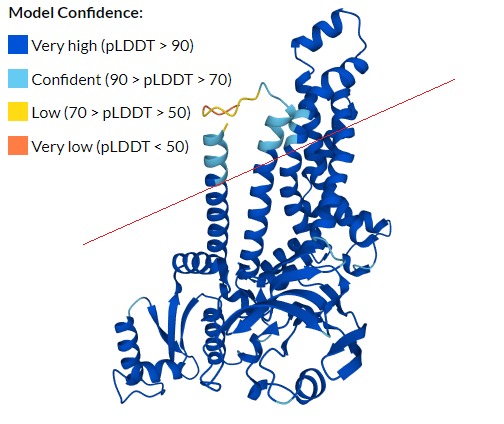
Рисунок 6. Достоверность предсказания структуры AmiC с помощью Alphafold. p-сторона предполагаемой мембраны снизу (не показана).
(алгоритмы предсказания трансмембранных спиралей сравнили в предыдущих разделах)
Предсказание AlphaFold (Рис. 6) в целом оказалось качественным, однако некачественные участки (N- и C-концы и маленькая область в середине) оказались рядом на внутренней границе предсказанной мембраны. Если на самом деле эти участки неструктурированы (т.е. не образовали всех возможных водородных связей и не могут находиться внутри мембраны), то реальная граница мембраны (красная линия на рисунке) пройдёт под углом к предсказанной, что повлияет на все предсказанные параметры (в частности, уменьшится толщина гидрофобной части белка в мембране, увеличится среднее кол-во остатков в одной трансмембранном участке и т.д.). Соответственно, если структура обсуждаемых участков действительно предсказана неправильно, то при повышении точности предсказания структуры изменится предсказание PPM трансмембранного положения.
Работа с базой данных TCDB
Выданный белок оказался не найден в TCDB. На вход базе был подан AC из UniProt (P0A4M8), результат: "Sorry! p0a4m8 is not included in TCDB".
Для FadL в TCDB нашлась информация (в основном ссылки на другие базы данных, из интересного – указаны транспортируемые липиды, почему-то указан 1 трансмембранный участок вместо 14 (15 с учётом первого)). TC-код FadL – 1.B.9.1.1. На соответствующей странице было определено, что он означает: 5 позиций кода – это класс, подкласс, семейство, подсемейство и номер самого транспортера внутри подсемейства соответственно. FadL в этой классификации занял следующее положение: класс "Каналы/поры", подкласс "β-бочонковые порины", семейство FadL (белка наружной мембраны FadL), подсемейство без названия.