Поиск ортологов и паралогов
Поиск гомологичных белков
Сначала был получен список гомологов АТФ-связывающей субъединицы протеазы Clp Escherichia coli:
cat *.fasta > /home/students/y20/artem.kays/term4/pr4/proteomes.fasta
makeblastdb -dbtype prot -in proteomes.fasta -out prot_db
blastp -query CLPX_ECOLI.fasta -db prot_db -evalue 0.001 -out blast_output.txt
Были найдены следующие белки:
Score E
Sequences producing significant alignments: (Bits) Value
sp|A1SME0|CLPX_NOCSJ ATP-dependent Clp protease ATP-binding subun... 528 0.0
tr|Q1AVT0|Q1AVT0_RUBXD ATP-dependent Clp protease ATP-binding sub... 524 0.0
sp|A1TCB3|CLPX_MYCVP ATP-dependent Clp protease ATP-binding subun... 523 0.0
sp|Q9CBY6|CLPX_MYCLE ATP-dependent Clp protease ATP-binding subun... 519 0.0
sp|Q8FN57|CLPX_COREF ATP-dependent Clp protease ATP-binding subun... 518 0.0
sp|A0JXL2|CLPX_ARTS2 ATP-dependent Clp protease ATP-binding subun... 516 0.0
tr|Q47MU4|Q47MU4_THEFY ATP-dependent Clp protease ATP-binding sub... 516 0.0
sp|B0RAS4|CLPX_CLAMS ATP-dependent Clp protease ATP-binding subun... 491 2e-173
tr|A0K1M3|A0K1M3_ARTS2 ATPase AAA-2 domain protein OS=Arthrobacte... 54.3 6e-08
tr|Q1AU05|Q1AU05_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus (... 52.0 3e-07
tr|Q8FMH5|Q8FMH5_COREF Putative endopeptidase Clp ATP-binding cha... 47.0 1e-05
tr|Q47MZ2|Q47MZ2_THEFY ATPase OS=Thermobifida fusca (strain YX) O... 43.9 1e-04
tr|Q1AY82|Q1AY82_RUBXD ATPase AAA-2 OS=Rubrobacter xylanophilus (... 43.5 2e-04
tr|A1TG29|A1TG29_MYCVP ATPase AAA-2 domain protein OS=Mycolicibac... 43.1 2e-04
sp|A0JXB1|RUVB_ARTS2 Holliday junction ATP-dependent DNA helicase... 42.4 3e-04
tr|A1SDV1|A1SDV1_NOCSJ ATP-dependent zinc metalloprotease FtsH OS... 42.0 4e-04
tr|Q47KU4|Q47KU4_THEFY ATP-dependent zinc metalloprotease FtsH OS... 42.0 5e-04
tr|A0JR82|A0JR82_ARTS2 ATP-dependent zinc metalloprotease FtsH OS... 41.6 5e-04
tr|A0K236|A0K236_ARTS2 AAA ATPase, central domain protein OS=Arth... 41.6 6e-04
tr|A1TG43|A1TG43_MYCVP ATP-dependent zinc metalloprotease FtsH OS... 41.6 6e-04
tr|B0RHW4|B0RHW4_CLAMS ATP-dependent zinc metalloprotease FtsH OS... 41.2 8e-04
Все найденные белки оказались АТФазами, при этом 15 из 21 являются протеазами.
Далее были изъяты последовательности этих белков и выровнены программой muscle:
for line in $(grep '^[s,t][p,r]|' blast_output.txt | cut -d\| -f2);
do echo 'proteomes.fasta:'$line >> ACs.txt;
done
seqret @ACs.txt seqs.fasta
muscle -in seqs.fasta -out seqs_al.fasta
Реконструкция филогении и визуализация
По полученному выравниванию в программе MEGA11 было реконструирвано филогенетическое дерево (скобочная формула) методом UPGMA:
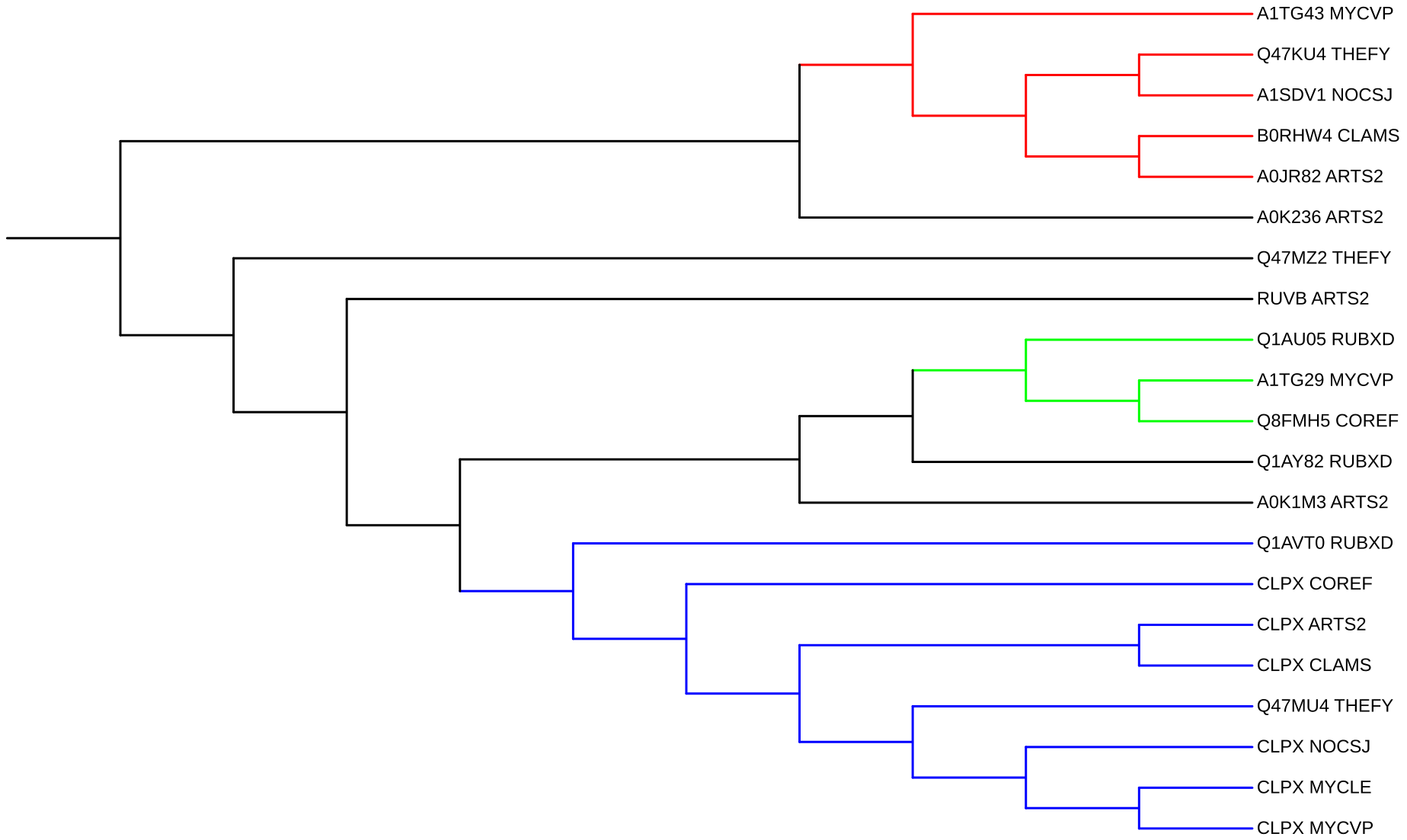
Рисунок 1. Полная реконструкция филогенетического дерева. Красным цветом обозначена ортологическая группа FtsH, зелёным – ClpC, синим – ClpX
Ортологи: A1TG43_MYCVP и A0JR82_ARTS2 (FtsH), Q1AU05_RUBXD и Q8FMH5_COREF (ClpC), Q1AVT0_RUBXD и CLPX_COREF (группа ClpX).
Паралоги: A0JR82_ARTS2 и A0K236_ARTS2, Q1AVT0_RUBXD и Q1AU05_RUBXD, Q47MU4_THEFY и Q47MZ2_THEFY.
На дереве выделяется явно выделяются 2 ортологические группы белков FtsH и ClpX и ещё одна группа из предсказанных белков. Из близости этой группы к ClpX и аннотации её белков можно предположить, что она содержит субъединицы C Clp протеаз (один из белков аннотирован как ClpC, другие 2 как просто АТФ-зависимые шапероны).
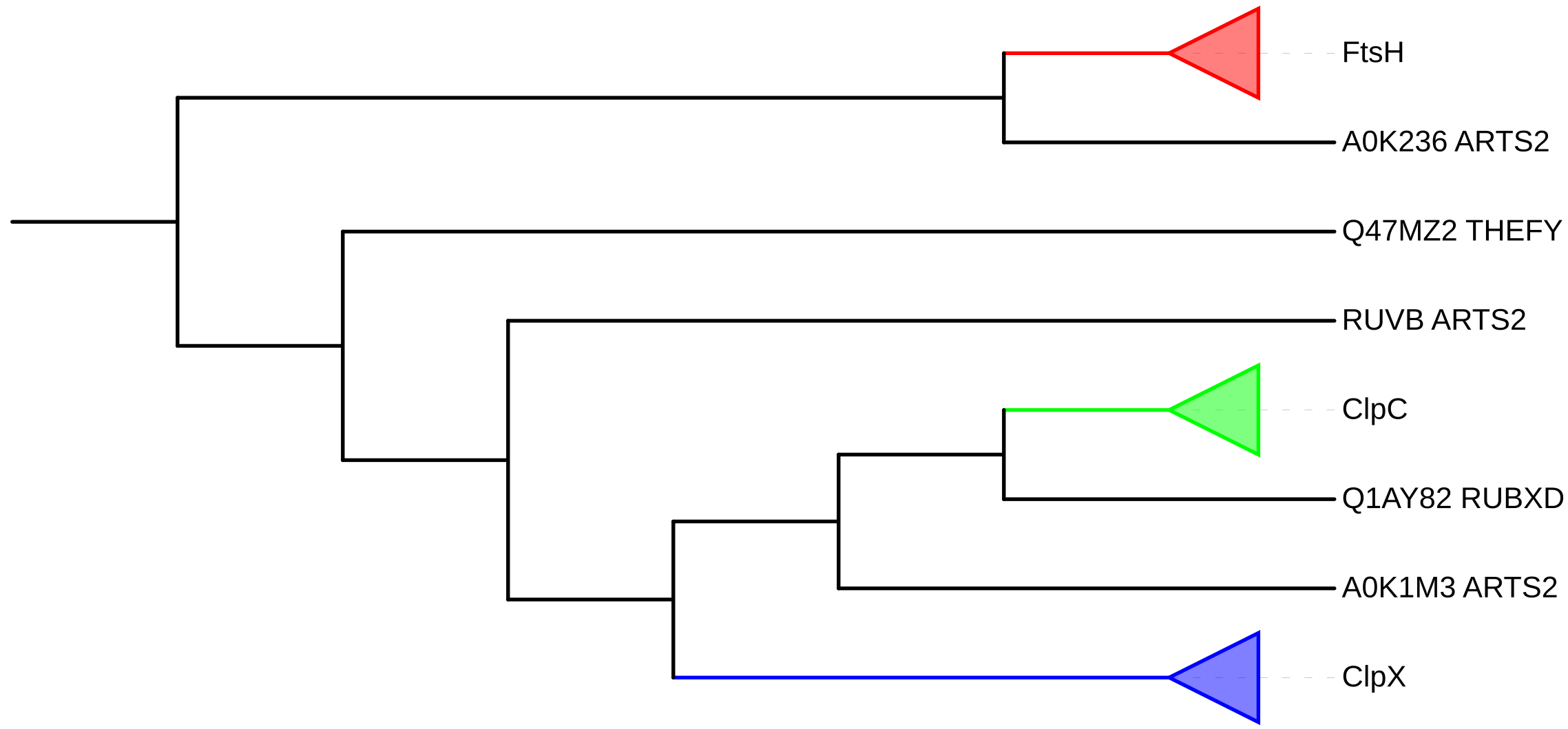
Рисунок 2. Дерево с объединёнными ортологичными группами.
Сравним реконструированную филогению каждой ортологической группы с филогенией бактерий:
Группа FtsH (АТФ-зависимые цинковые металлопротеазы) содержит 5 белков (недостаёт белков видов RUBXD, COREF и MYCLE), но при этом не противоречит филогенетическому дереву бактерий (добавляя недостающие листья, можно получить исходное дерево).
Группа ClpC (АТФ-связывающие субъединицы Clp протеаз) содержит всего 3 белка (видов RUBXD, COREF и MYCVP) и тоже не противоречит исходному дереву.
Группа ClpX (АТФ-связывающие субъединицы Clp протеаз) представлена белками всех 8 видов, но содержит только одну из пяти нетривиальных ветвей филогенетического дерева бактерий – белок RUBXD против всех остальных белков. На дереве выделяются клады (ARTS2, CLAMS), (MYCVP, MYCLE) и (MYCVP, MYCLE, COREF, THEFY, NOCSJ, ARTS2, CLAMS), сходные с исходным деревом, однако больше никаких общих с ним клад нет.