Сигналы и мотивы – 1
Cигнал вставки селеноцистеина SECIS
SECIS (selenocysteine insertion sequence) представляет собой последовательность длиной около 60 нуклеотидов (Рис. 1), находящуюся либо в рамке считывания между UGA и следующим стоп-кодоном (у прокариот), либо в 3'-нетранслируемой области (у эукариот).
В составе мРНК последовательность SECIS формирует шпильку, которая связывается с фактором элонгации (SelB у про- и eEFsec у эукариот соответственно), привлекающим селеноцистеиновую тРНК в A сайт рибосомы, когда там находится стоп-кодон UGA (Рис. 2). В результате вместо терминации трансляции происходит вставка селеноцистеина в полипептидную цепь.
SECIS высокоэффективен, но только в присутствии селеноцистеина в клетке. В его отсутствие может произойти как вставка цистеина, связанного с селеноцистеиновой тРНК, так и терминация трансляции.
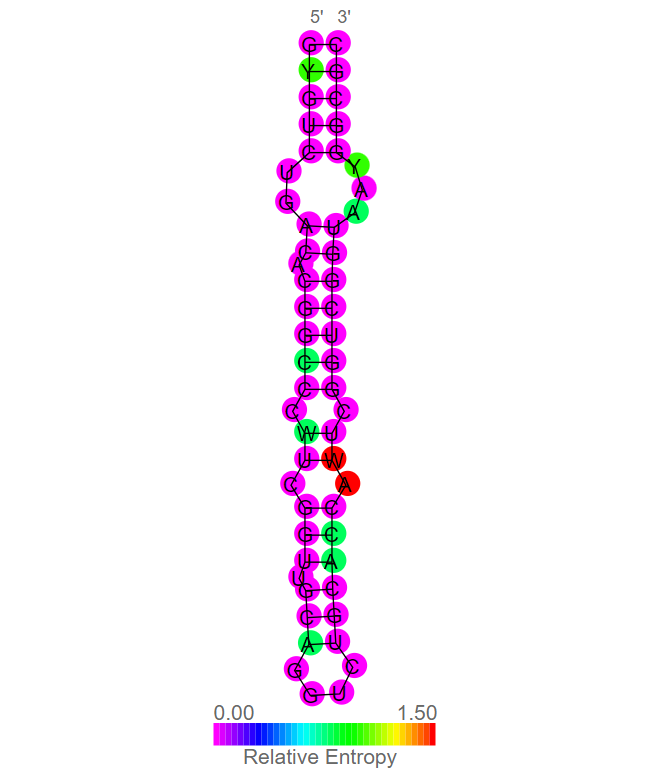
Рисунок 1. Вторичная структура одного из прокариотических типов SECIS (источник – база данных Rfam: RF01988).
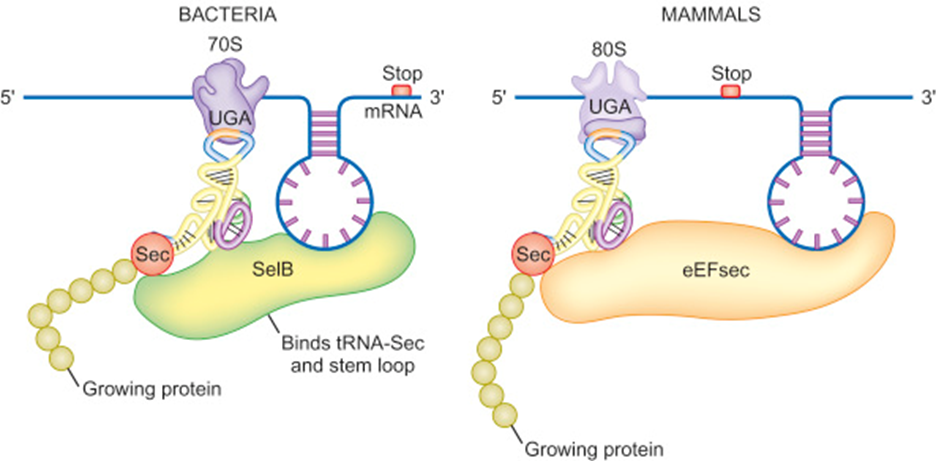
Рисунок 2. Схема действия SECIS (источник – лекции по молекулярной биологии для 3 курса ФББ МГУ).
Источники:
- Hanna Engelberg-Kulka, Zesheng Liu, Chuang Li, Myriam Reches (2001). An extended Escherichia coli “Selenocysteine Insertion Sequence” (SECIS) as a multifunctional RNA structure. Biofactors, 14(1-4), 61–68. DOI: 10.1002/biof.5520140109;
- Labunskyy, V. M., Hatfield, D. L., Gladyshev, V. N. (2014). Selenoproteins: Molecular Pathways and Physiological Roles. Physiological Reviews, 94(3), 739–777. DOI: 10.1152/physrev.00039.2013.
Построение позиционной весовой матрицы (PWM) для последовательности Козак
Для начала из предложенной в задании таблицы человеческих генов были отобраны включающие стартовый кодон ATG участки (lrn.fasta) 40 генов для построения PWM и ещё 60 (tst.fasta) для положительного контроля с помощью следующего скрипта (включает код из подсказок к практикуму): lrn+pos_ctrl.py.
Для отрицательного контроля надо было отобрать 60 участков с ATG, не являющимися старт-кодонами. Вероятность того, что случайно взятый кодон является ATG равна p(A)*p(T)*p(G) = 0.27952*0.2205 = 0.0172. Тогда можно приближённо оценить, что в геноме содержится 3088269832*0.0172 = 53197038 ATG, что на 3 порядка больше числа генов (около 20 тысяч), поэтому будем считать, что в случайной выборке ATG стартовых окажется ничтожно мало, и возьмём в качестве отрицательного контроля случайные ATG (GC-состав и длины хромосом брались из NCBI).
Чтобы эффективно выбрать случайные ATG, будем брать случайные участки в геноме и искать в них ATG. Оценим, какую минимальную длину должны иметь такие участки, чтобы в них гарантированно (будем считать вероятность 0.99 достаточной) встретился хотя бы один ATG. Будем считать, что число ATG распределено биномиально с вероятностью p(ATG) = 0.0172. Тогда 0.99 = P(k(ATG)>0) = 1 - P(k(ATG)=0) = 1 - (1-p(ATG))n → n = ln(0.01) / ln(1 - p(ATG)) = 265.
ATG для отрицательного контроля (neg_ctrl.fasta) были получены следующим скриптом: neg_ctrl.py.
По 40 участкам для обучения была построена PWM (pwm.csv) и матрица информационного содержания сигнала (ic.csv). Также была проверена гипотеза о равенстве средних весов положительного и отрицательного контроля (res.txt). Она была отвергнута с p-value = 0.0008, веса значимо различаются: у положительного контроля – 4.24, у отрицательного – 2.75 (скрипт для матриц и проверки гипотезы: res.txt). В первом случае наблюдается наличие последовательности Козак.
LOGO
Итоговое информационное содержание сигнала оказалось равным 8.21. По визуализации матрицы IC (Рис. 3) видно, что информационное содержание сигнала достаточно низкое: для 2, 3 и -1 позиции оно практически равно нулю, то есть по этим позициям распознавание сигнала рибосомой, судя по всему, не идёт
.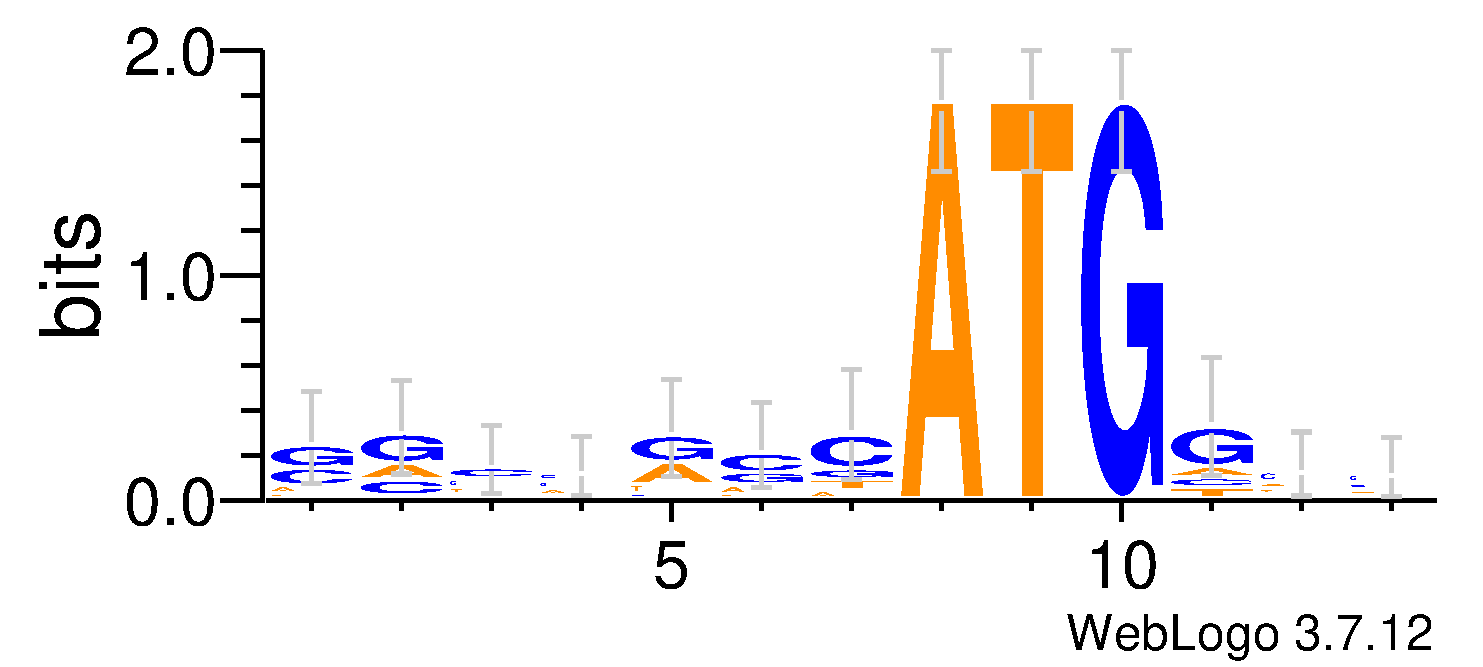
Рисунок 3. Визуализация информационного содержания Козак.