BLAST
Часть 1
Для последовательности белка Цианат-гидратазы S. proteamaculans был произведен поиск гомологичных последовательностей с помощью инструмента BLAST.
Из таблицы взяты имена последовательностей из начала, середины и конца. Для них с помощью muscle было построено выравнивание с помощью muscle.
| Параметр | Его значение |
|---|---|
| Database | База данных |
| Organism | Какие организмы включить/исключить |
| Exclude | Исключить группу последовательностей из рассмотрения |
| Algorithm | Алгоритм поиска |
| Max target sequences | Максимальное число последовательностей для поиска |
| Short queries | Производить ли автоматическую замену параметров для маленьких последовательностей |
| Expect threshold | Ожидаемое число случайных совпадений в произвольной последовательности |
| Word size | Длина инициализирующего зерна (слова с которого начинается выравнивание) |
| Max matches in a query range | Ограничение для числа возможных связей с последовательностью. (Используется в случае сильного связывания) |
| Matrix | Тип матрицы |
| Gap Costs | Цена гэпа |
| Compositional adjustments | Тип поправок для матрицы, связанных с характеристиками последовательности. |
| Filter | Не учитывать фрагменты с низкой сложностью (повторы) |
| Mask | Не использовать последовательность для генерации зерна. Скрыть строчные символы. |

Выравнивание можно скачать по ссылке.
Изображение получено с помощью программы Jalview.
Взяли 15 имен последовательностей из результатов BLAST с начала и с низа списка, так чтобы параметр E-value был разным. Для них построено выравнивание. PKM04286.1 сразу же удалили, так как он оказался фрагментом белка. Остальные 8 также были исключены, на основе того, что их начальные или конечные позиции не были консервативны. Оставшиеся последовательности выровняли заново.
На полученном выравнивании (Рисунок 1) чётко видна гомология последовательностей. Они все начинаются на MTQS(L/Q) (за небольшим исключением) и кончаются на GKYLPTKPF. В них нет гэпов. С 35 по 51 и с 74 по 114 они полностью гомологичны (и в некоторых других позициях).
Часть 2
На странице BLASTp нужно отметить опцию Align two or more sequences и запустить выравнивание.
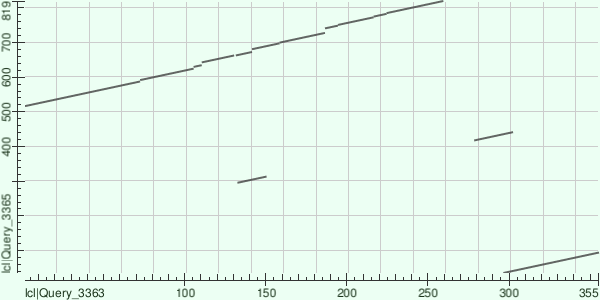
Изображение получено с помощью BLAST.
На C конце первой последовательности (расположенной по вертикали) и C конце второй (расположенной по горизонтали) лежит почти полностью гомологичный участок длинной 250 аминокислот. На N конце первой лежит фрагмент гомологичный фрагменту C конца второй, также в середине первой лежит фрагмент гомологичный другому фрагменту этой последовательности (дупликация), его также можно найти и у второй последовательности. В середине первой и на C конце второй лежат гомологичные фрагменты. Инверсий нигде нет.
Часть 3
Для игры с BLAST я вспомнил строку известного стихотворения Роберта Бёрнса "my heart is in the highlands". Её я стал искать. Я пробовал разные варианты Expect threshold (0-5-10), Gap cost, варианта матрицы и алгоритма (blastp, Quick BLASTP) – результат не менялся, я получал один и тот же набор пептидов (с очень большим E).
Для второго раунда я использовал последовательность своего белка. Я сделал несколько сеансов поиска, каждый раз изменяя какой-нибудь один параметр.
Изначально было найдено 6253 последовательностей, когда я увеличил цену гэпа () было найдено 6248.
Я искал белки только по базе данных SwissProt, и тогда было найдено всего 104 штуки.
Low complexity regions – параметр не изменил ничего.
Выбор PAM30 привел к тому, что было найдено 6248 последовательностей.
"My heart is in the highlands"
Robert Burns
©Бакулин Артемий, 2018