Ресеквенирование. Поиск полиморфизмов у человека.
Индексирование референса
- Создали директорию mkdir artemisbakulin
- Скопировали референс cp chr3.fasta artemisbakliin
- Проиндексировали его: hisat2-build chr3.fasta chr3
Анализ качества прочтений
- Скопировали прочтения к себе в директорию: cp /nfs/srv/databases/ngs/Human/reads/chr3.fastq ./
- Проели анализ качества: fastqc chr3.fastq
- Обрезали с конца каждого рида нуклеотиды с качеством меньше 20, и оставили только те последоватеьности длина которых была больше 50:
java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr3.fastq chr3.trimmed.fastq TRAILING:20 MINLEN:50 - Провели повторный анализ качества: fastqc chr3.trimmed.fastq
Результаты анализа:
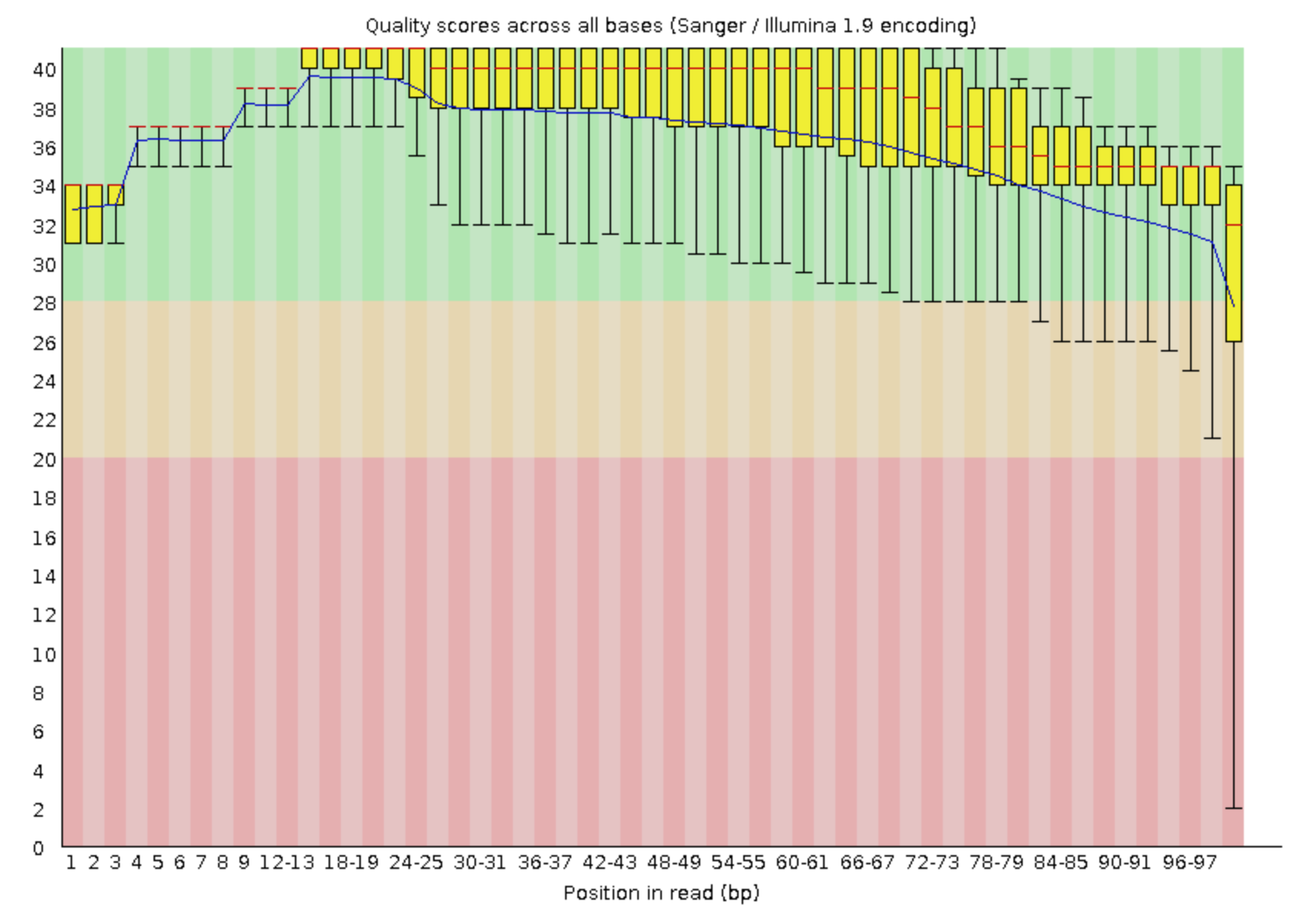
Видно, что качество хорошее, есть только незначительное количество выбросов с качеством меньше 20.
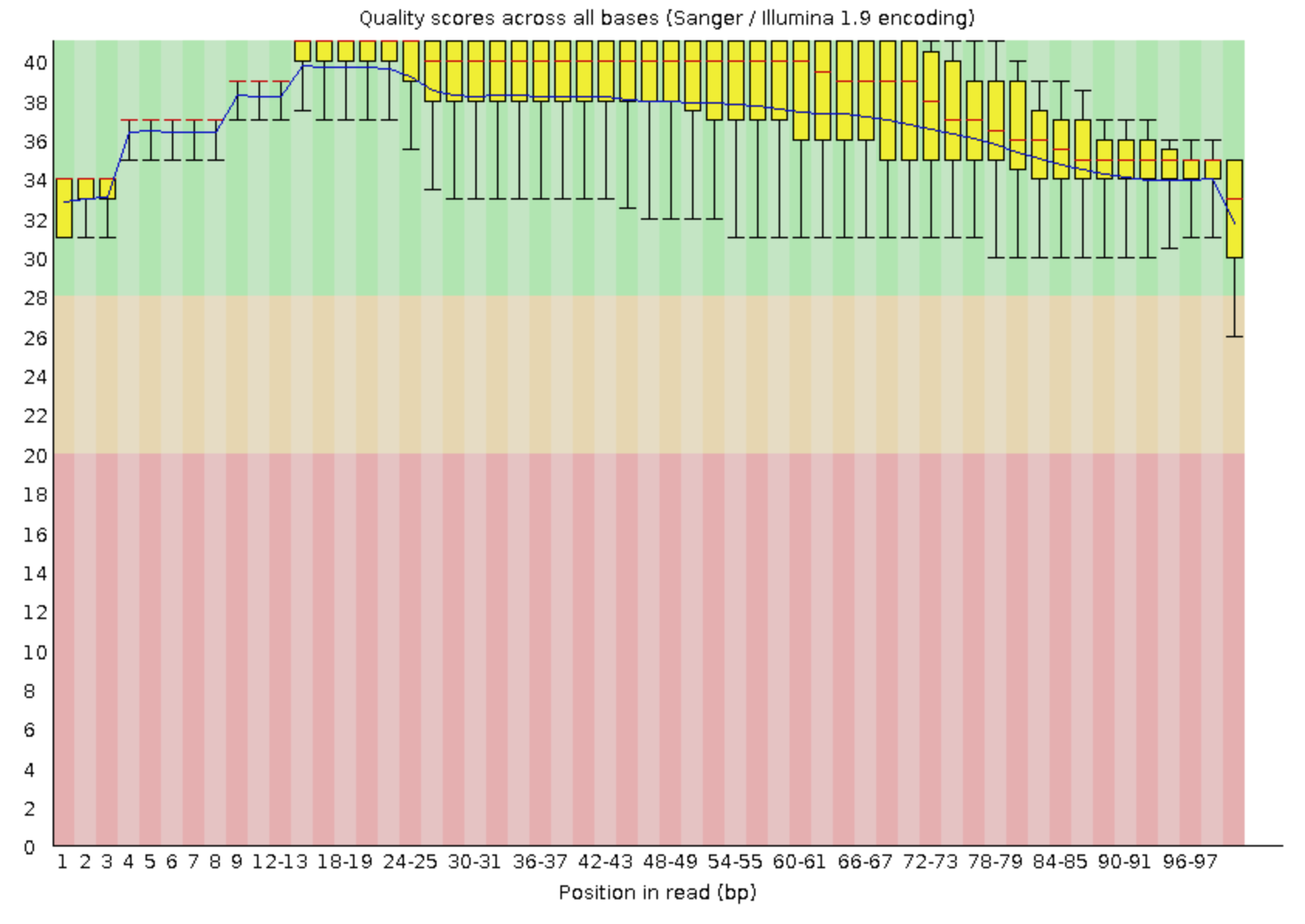
Видно, что trimmomatic убрал все выбросы.
Всего было удалено 362 прочтения, то есть около 2%. В принципе это нужно было делать, но не критично.
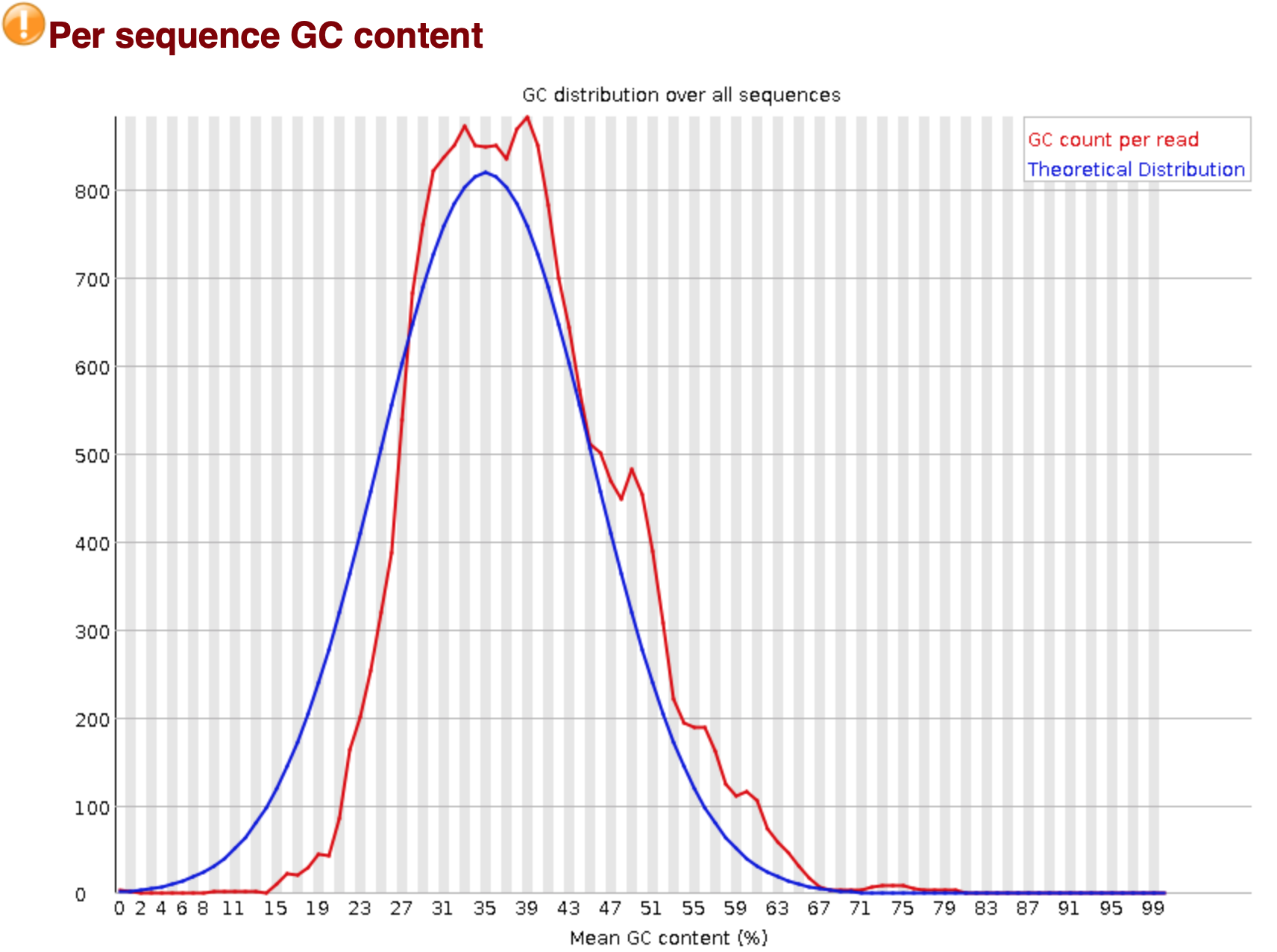
Гипотетически содержание GC в прочтениях должно соответствовать нормальному распределению (синяя линия), мы видим отклонения от него (красная линия), но в целом наше распределение близко к нормлаьному.
Картирование
Затем эти прочтения были картированы на геном.
- Картирование: hisat2 -x chr3 -U chr3.trimmed.fastq --no-spliced-alignment --no-softclip > chr3.sam
- Переход в бинарный формат: samtools view chr3.sam -bo aligned.bam
- Сортировка: samtools sort chr3.bam -T aligned -o chr3.sorted.bam
- Статистика по картированию: samtools flagstat chr3.sorted.bam > flagstat.out
Оказалось, что накартировались 99.62% ридов, это хорошо, только 79 прочтений из 20589 не смогли пройти эту операцию.
Поиск SNP
- Коллинг полиморфизмов: samtools mpileup -uf chr3.fasta -o chr3.bcf aligned.sorted.bam
- Переход из бинарного формата в текстовый: bcftools call -cv chr3.bcf -o chr3.vcf
- Получение статистики по полиморфизмам: bcftools stats chr3.vcf > stats.out
- Построение графика распределение глубины картирования полиморфизмов: plot-vcfstats -p plot_vcf stats.out
Итак всего было найдено 218 SNPs и 12 инделей.
Примеры полиморфизмов:
| Позиция | Тип полиморфизма | Референс | Вариант | Качество | Глубина |
|---|---|---|---|---|---|
| 41356306 | Инсерция | ctgtgtgtgtgtgtgtgtgt | cTGtgtgtgtgtgtgtgtgtgt,ctgtgtgtgtgtgtgtgt | 14.6233 | 2 |
| 41607450 | Транзиция | C | T | 222.009 | 52 |
| 41939993 | Трансверсия | T | A | 225.009 | 67 |
Базы данных snp
- dbsnp:
annotate_variation.pl -filter -out snp138_filtered -build hg19 -dbtype snp138 snp.avinput /nfs/srv/databases/annovar/humandb.old/ - refgene:
annotate_variation.pl -out refgen -build hg19 snp.avinput /nfs/srv/databases/annovar/humandb.old/ - 1000 genomes:
annotate_variation.pl -filter -out 1000g -build hg19 -dbtype 1000g2014oct_all snp.avinput /nfs/srv/databases/annovar/humandb.old/ - Gwas:
annotate_variation.pl -regionanno -out gwas -build hg19 -dbtype gwasCatalog snp.avinput /nfs/srv/databases/annovar/humandb.old/ - Clinvar:
annotate_variation.pl -filter -out clinvar -build hg19 -dbtype clinvar_20150629 snp.avinput /nfs/srv/databases/annovar/humandb.old/
Итоги анализа snp по базам данных:
Из файла refgen.variant_function можно извлечь следующие данные:
| Категория | Количество snp |
| exonic | 13 |
| intronic | 198 |
| UTR3 | 4 |
| intergenic | 1 |
| splicing | 0 |
| ncRNA | 0 |
| UTR5 | 2 |
| upstream | 0 |
| downstream | 0 |
SNP попали в гены ULK4, FNDC3B
Из файла refgen.exonic_variant_function можно извлечь следующие данные о нуклеотидных заменах. Префиксом c. обозначены нуклеотидные замены, p. аминокислотные.
nonsynonymous SNV ULK4:NM_017886:exon33:c.A3292G:p.K1098E
nonsynonymous SNV ULK4:NM_017886:exon24:c.G2551A:p.V851I
nonsynonymous SNV ULK4:NM_017886:exon24:c.T2530A:p.L844M
nonsynonymous SNV ULK4:NM_017886:exon21:c.G2143A:p.A715T
nonsynonymous SNV ULK4:NM_017886:exon20:c.T1918G:p.S640A
nonsynonymous SNV ULK4:NM_017886:exon19:c.T1808C:p.L603S
nonsynonymous SNV ULK4:NM_017886:exon18:c.A1706G:p.K569R
synonymous SNV ULK4:NM_017886:exon17:c.A1599G:p.V533V
synonymous SNV ULK4:NM_017886:exon16:c.A1536G:p.Q512Q
nonsynonymous SNV ULK4:NM_017886:exon11:c.A1042G:p.S348G
nonsynonymous SNV FNDC3B:NM_001135095:exon6:c.C536G:p.T179S,FNDC3B:NM_022763:exon6:c.C536G:p.T179S
synonymous SNV FNDC3B:NM_001135095:exon6:c.T687C:p.H229H,FNDC3B:NM_022763:exon6:c.T687C:p.H229H
synonymous SNV FNDC3B:NM_001135095:exon12:c.T1374C:p.G458G,FNDC3B:NM_022763:exon12:c.T1374C:p.G458G
В выдаче dbsnp было найдено 177 snp, имеющих rs.
О частотах можно узнать из выдачи 1000g. Частоты лежат в диапазоне между 1 и 95%. Скачать
В выдаче GWAS написано, что найденные полиморфизмы ассоциированы с высоким ростом, с высоким кровяным давлением и риском развития диабета второго типа (ссылка на статью).
©Бакулин Артемий, 2018