Секвенирование по Сэнгеру
Взяли хроматограммы прямой и обратной цепи какой-то последовательности и посмотрели на их качество. Для этого их открыли в программе 4Peaks. И выровняли относительно друг друга.
И в целом на первый взгляд они оказываются неплохими. В среднем уровень сигнала оказывается в четыре-пять раз выше шума. Впрочем высота пиков оказывается не одинаковой, раз в тридцать нуклеотидов появляются высокие пики. В центральной части пики достаточно узкие, не накладываются друг на друга. На прямой цепи в центральной части было обнаружено 40 гэпов, на обратной 60. Однако в основном позиции гэпов не пересекались и можно было вполне установить, какой это нуклеотид.
Конечно, качество концов оставляет желать лучшего, но ничего иного ожидать никогда и не приходится.
Из бинарного файла можно было извлечь следующие сведения о качестве секвенирования:
3' и 5' концы было достаточно трудно разобрать, поэтому я их обрезал в обоих последовательностях. С 3' конца прямой 47 (встроенная утилита предлагала 18), с обратной 52 (программа предлагала 9), 5' конец прямой был отрезан с 689 позиции (программа предлагала 758), а у обратной с 694 (программа предлагала 770).
Далее будут представлены примеры сделанных замен в прямой цепи на основе выравнивания с обратной и оценки качества пиков. Сверху прямая цепь, снизу обратная.
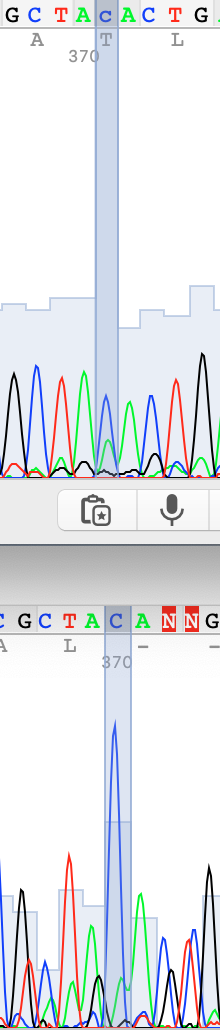
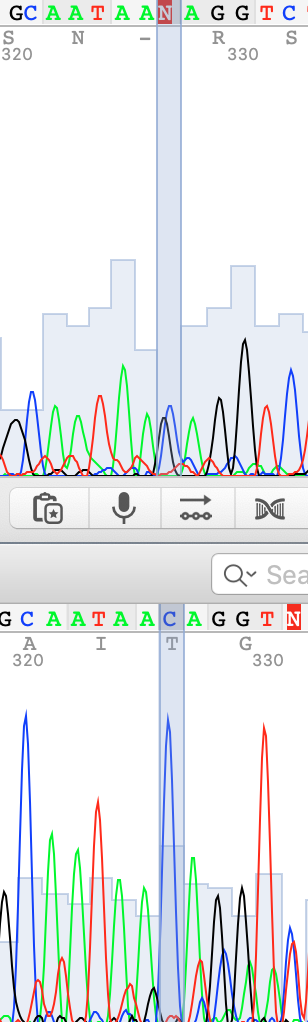
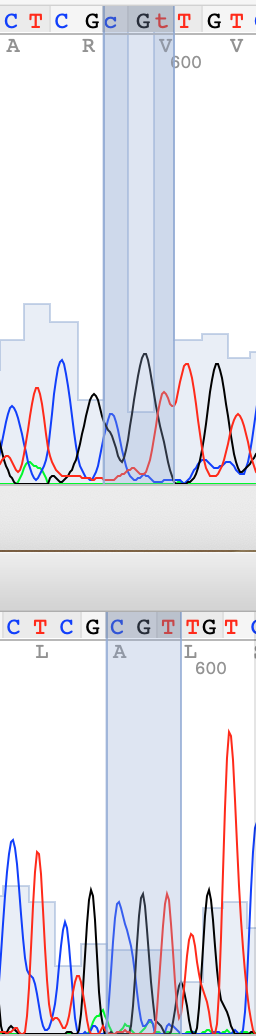
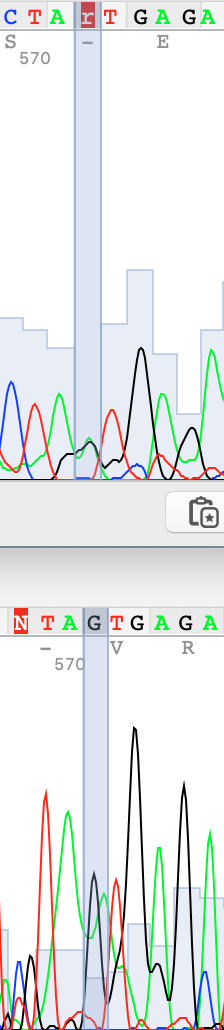
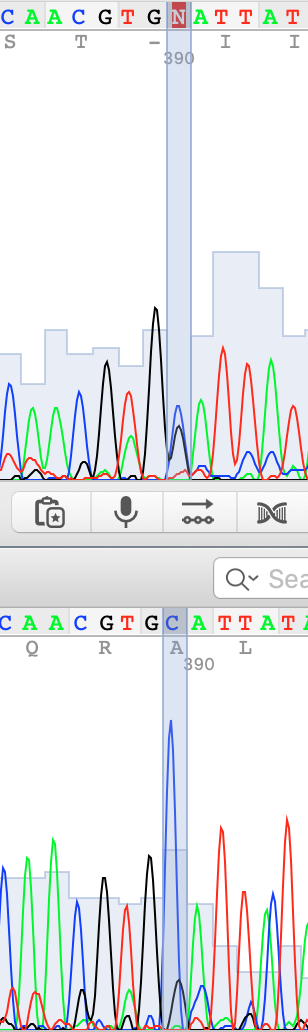
Сами хроматограммы можно скачать здесь: прямая и обратная .
Отредактированная прямая последовательность
Выравнивание отредактированной прямой последовательности и изначальной обратной (перевернутой комплементарной к обратной).
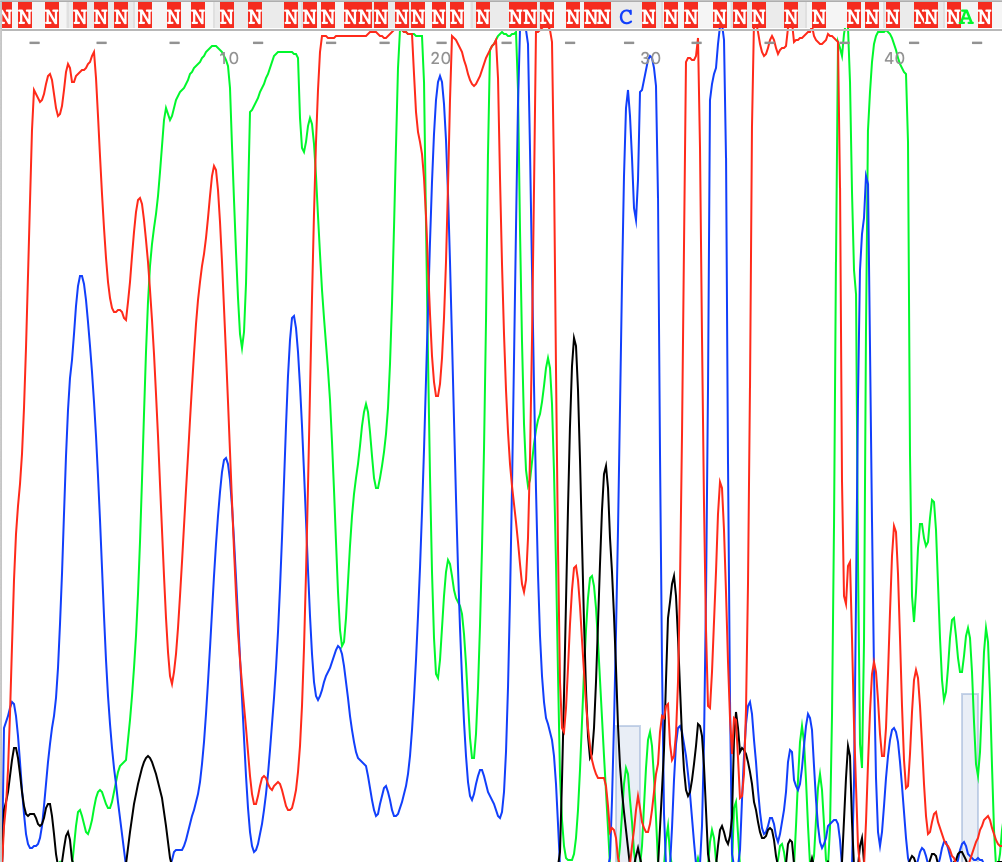
Плохая хроматограмма
В качестве примера плохой хроматограммы был взят образец WS2943_SP6R.ab1.
Здесь видны множественные наслоения пиков друг на друга, кроме того они неадекватно большого размера. Это можно связать либо с техническим загрязнением самого секвенатора или же образца другой ДНК. По такой хроматограмме ничего установить нельзя.
©Бакулин Артемий, 2018