Структура нуклеиновых кислот
Задание 1
В задании предлагалось построить трехмерные структуры a, b, z форм ДНК в формате pdb.
Пример команды: fiber -z -seq=gatcgatcgatcgatcgatc gatc-z.pdb
В частности были построены a и b формы для последовательности (GATC)5 и z форма для (GC)20.
Скачать a форму
Скачать b форму
Скачать z форму
Задание 2
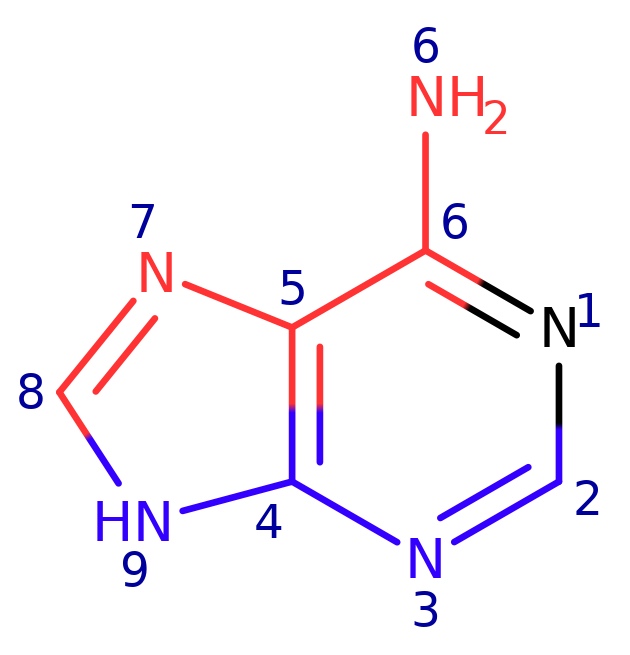
Из банка структур PDB была структура последовательности с ID 3V9D. В ней я рассмотрел расположение атомов 8 аденина.
В сторону большой бороздки обращены атомы: C8, N7, C5, C6, N6.
В сторону малой бороздки обращены атомы: N9, C4, N3, C2.
Затем в программе pymol были проанализированы a,b, z структуры ДНК.
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | Правозакрученная | Правозакрученная | Левозакрученная |
| Шаг спирали | 28.6 Å | 35.7 Å | 45.6 Å |
| Среднее число оснований на виток | 11 | 10.5 | 12 |
| Ширина большой бороздки | 11.0 Å (/3v9d//B/DG`5/P - /3v9d//A/DC`4/P) | 17.4 Å (/1bna/B/B/DT`19/P - /1bna//A/DG`2/P) | 17.2 Å (/1tne//A/DC`5/P - /1tne//B/DC`11/P) |
| Ширина малой бороздки | 15.6 Å (/3v9d//A/DC`4/P - /3v9d//B/DG`13/P) | 8.9 Å (/1bna//A/DG`10/P - /1bna//B/DT`19/P) | 8.2 Å (/1tne/A/A/DG`6/P - /1tne/B/B/8MG`10/P) |
Задание 3
Для выполнения следующего задания из банка PDB была скачана структура 2CV1.
Предварительно потребовалось перевести файл со структурой в старый формат, потому что только с ним может работать программа.
Это было сделано командой: remediator --old 2cv1_clean.pdb > 2cv1_old.pdb
Этот результат был перенаправлен утилите find_pair:
find_pair -t 2cv1_old.pdb 2cv1_old.out
Торсионные углы
Были получены значения торсионных углов для нуклеотидов каждой цепи
Первая цепь
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | ||
|---|---|---|---|---|---|---|---|---|---|
| 1 | G | --- | 173.7 | 38.6 | 90.4 | -139.2 | -80.3 | -175.6 | |
| 2 | G | -73.3 | -179.4 | 52.4 | 85.3 | -139.2 | -73.7 | -165.0 | |
| 3 | C | -69.1 | 167.7 | 55.5 | 81.7 | -148.9 | -70.9 | -160.5 | |
| 4 | C | -59.8 | 170.9 | 50.6 | 79.5 | -156.3 | -76.7 | -161.6 | |
| 5 | C | -55.8 | 170.3 | 50.8 | 79.4 | -153.3 | -68.4 | -157.9 | |
| 6 | C | -67.8 | 171.8 | 62.8 | 85.7 | -159.2 | -80.4 | -165.2 | |
| 7 | A | -44.9 | -177.4 | 50.2 | 112.4 | 27.8 | 159.0 | -124.6 | |
| 8 | G | 47.8 | 147.2 | 43.5 | 81.8 | -141.2 | -73.1 | -176.4 | |
| 9 | G | -64.3 | 179.0 | 50.3 | 80.0 | -153.9 | -72.9 | -165.2 | |
| 10 | G | -56.4 | 173.0 | 47.0 | 82.1 | -163.9 | -89.5 | -162.6 |
Вторая цепь
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | ||
|---|---|---|---|---|---|---|---|---|---|
| base | alpha | beta | gamma | delta | epsilon | zeta | chi | ||
| 1 | C | -84.2 | 175.8 | 61.8 | 86.4 | -147.3 | 151.1 | -146.4 | |
| 2 | U | -77.9 | 173.6 | 64.2 | 85.5 | -157.4 | -72.1 | -164.0 | |
| 3 | G | -53.2 | 169.6 | 51.7 | 84.1 | -152.3 | -65.8 | -168.7 | |
| 4 | G | -56.7 | 173.1 | 49.4 | 82.2 | -154.9 | -75.7 | -168.1 | |
| 5 | G | -69.5 | 178.4 | 51.2 | 80.6 | -156.6 | -69.8 | -164.3 | |
| 6 | G | -55.7 | 171.1 | 45.2 | 83.5 | -154.2 | -72.0 | -159.7 | |
| 7 | U | -65.6 | 179.2 | 49.3 | 84.2 | -152.1 | -70.7 | -153.3 | |
| 8 | C | -56.3 | 157.5 | 57.2 | 86.0 | -157.6 | -68.9 | -159.4 | |
| 9 | C | -66.2 | 171.7 | 54.7 | 83.6 | -138.8 | -81.4 | -165.3 | |
| 10 | C | -69.3 | -178.7 | 50.7 | 83.6 | -156.1 | -77.9 | -161.5 |
Структура водородных связей
В том же файле были найдены координаты оснований, образующих пары. Исходя из этих данных можно предсказать координаты стеблей данной молекулы. Соответсвенно: 501-7 и 566-572, 510-513 и 522-525, 549-553 и 561-565, 538-544 и 526-532
1 (0.008) ....>C:.501_:[..G]G-----C[..C]:.572_:C<.... (0.004) |
2 (0.010) ....>C:.502_:[..G]G-*---U[..U]:.571_:C<.... (0.009) |
3 (0.007) ....>C:.503_:[..C]C-----G[..G]:.570_:C<.... (0.010) |
4 (0.005) ....>C:.504_:[..C]C-----G[..G]:.569_:C<.... (0.004) |
5 (0.007) ....>C:.505_:[..C]C-----G[..G]:.568_:C<.... (0.007) |
6 (0.010) ....>C:.506_:[..C]C-----G[..G]:.567_:C<.... (0.006) |
7 (0.005) ....>C:.507_:[..A]A-----U[..U]:.566_:C<.... (0.004) |
8 (0.008) ....>C:.549_:[..G]G-----C[..C]:.565_:C<.... (0.004) |
9 (0.005) ....>C:.550_:[..G]G-----C[..C]:.564_:C<.... (0.008) |
10 (0.004) ....>C:.551_:[..G]G-----C[..C]:.563_:C<.... (0.003) |
11 (0.004) ....>C:.552_:[..G]G-----C[..C]:.562_:C<.... (0.005) |
12 (0.004) ....>C:.553_:[..G]G-----C[..C]:.561_:C<.... (0.002) |
13 (0.005) ....>C:.554_:[..U]U-**--A[..A]:.558_:C<.... (0.005) |
14 (0.006) ....>C:.555_:[..U]U-**+-G[..G]:.518_:C<.... (0.005) x
15 (0.007) ....>C:.538_:[..A]A-**--C[..C]:.532_:C<.... (0.003) |
16 (0.015) ....>C:.539_:[..G]G-----C[..C]:.531_:C<.... (0.004) |
17 (0.013) ....>C:.540_:[..G]G-----C[..C]:.530_:C<.... (0.006) |
18 (0.002) ....>C:.541_:[..C]C-----G[..G]:.529_:C<.... (0.007) |
19 (0.007) ....>C:.542_:[..C]C-----G[..G]:.528_:C<.... (0.006) |
20 (0.008) ....>C:.543_:[..G]G-----C[..C]:.527_:C<.... (0.008) |
21 (0.007) ....>C:.544_:[..A]A-**--G[..G]:.526_:C<.... (0.010) |
22 (0.008) ....>C:.510_:[..G]G-----C[..C]:.525_:C<.... (0.002) |
23 (0.004) ....>C:.511_:[..U]U-----A[..A]:.524_:C<.... (0.004) |
24 (0.007) ....>C:.512_:[..C]C-----G[..G]:.523_:C<.... (0.007) |
25 (0.007) ....>C:.513_:[..U]U-*---G[..G]:.522_:C<.... (0.009) |
26 (0.013) ....>C:.514_:[..A]A-**--U[..U]:.508_:C<.... (0.004) |
27 (0.017) ....>C:.515_:[..G]G-**+-C[..C]:.548_:C<.... (0.016) x
28 (0.007) ....>C:.519_:[..G]G-----C[..C]:.556_:C<.... (0.006) +
Дополнительные водородные связи
Ниже представлен фрагмент выхода программы, в котором содержится подробная информация о водородных связях. Здесь присутствуют, как канонические пары, так и так называемые неоднозначные пары оснований G-U (номера 2, 14). Они дополнительно стабилизируют структуру.
1 G-----C [3] O6 - N4 3.24 N1 - N3 3.38 N2 - O2 3.48
2 G-*---U [2] O6 - N3 2.82 N1 - O2 2.86
3 C-----G [3] O2 - N2 2.98 N3 - N1 2.98 N4 - O6 2.99
4 C-----G [3] O2 - N2 2.75 N3 - N1 2.92 N4 - O6 3.00
5 C-----G [3] O2 - N2 2.98 N3 - N1 2.94 N4 - O6 2.82
6 C-----G [3] O2 - N2 2.79 N3 - N1 2.91 N4 - O6 2.99
7 A-----U [2] N6 - O4 3.18 N1 - N3 3.24
8 G-----C [2] N1 - N3 3.79 N2 - O2 3.54
9 G-----C [3] O6 - N4 3.05 N1 - N3 3.06 N2 - O2 2.94
10 G-----C [3] O6 - N4 3.14 N1 - N3 2.99 N2 - O2 2.70
11 G-----C [3] O6 - N4 2.86 N1 - N3 2.85 N2 - O2 2.70
12 G-----C [3] O6 - N4 2.89 N1 - N3 3.04 N2 - O2 3.06
13 U-**--A [2] O2 - N6 2.95 N3 - N7 3.06
14 U-**+-G [1] O2 - N2 2.78
15 A-**--C [1] N6 - O2 3.10
©Бакулин Артемий, 2018