PFAM
Выбранные домены:
PF01966:
ID: HD
Название: HD domain
Число последовательностей: 186,899
PF18211:
ID: Csm1_B
Название: Csm1 subunit domain B
Число последовательностей: 1,031
Поиск был осуществлен следующей командой:
taxonomy:"Bacteria [2]" database:(type:pfam pf01966) database:(type:pfam pf18211)
HMM профиль
Сначала я построил распределние длин белков.
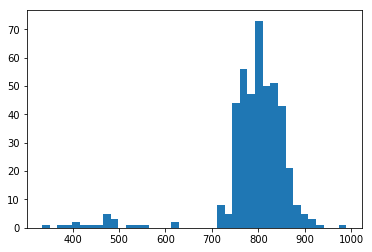
Для того чтобы построить HMM профиль я взял 82 последовательности белков в диапазоне длин от 800 до 810 а.о..
Я скачал последовательности этих белков и построил по ним выравнивание. Скачать выравнивание Посмотрев на выравнивание я удалил несколько колонок с обоих концов выравнивания. Заново выровнял последовательности.
Это выравнивание я использовал для построения HMM профиля. Я запустил серию команд.
hmm2build profile.hmm alignment_pr9.fa
hmm2calibrate profile.hmm
Скачать профиль
Чтобы оценить полученный профиль – я скачал все белки, которые содержат домен pf01966 (186899 последовательностей).
В них я искал белки соответствующие полученному мной профилю:
hmm2search profile.hmm sequences_pr9.fasta
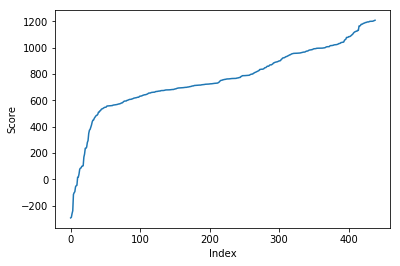
Результаты поиска белков я обработал в python. Скачать скрипт. Я построил ROC curve и зависимость F1 от порога – в принципе они были не нужны. Среди находок было всего два ложно-положительных результатов. Это означает, что HMM профиль очень хороший. Можно взять порог -259.0 и тогда профиль будет иметь специфичность и чувствительность строго равные единице.
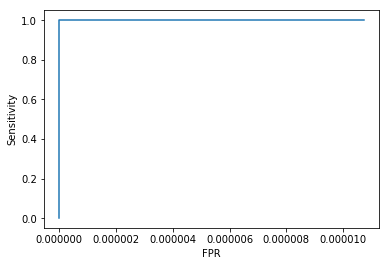
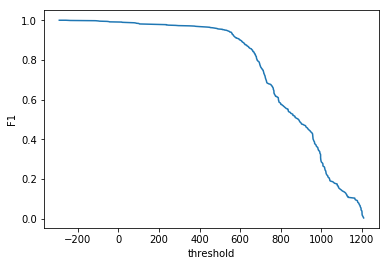
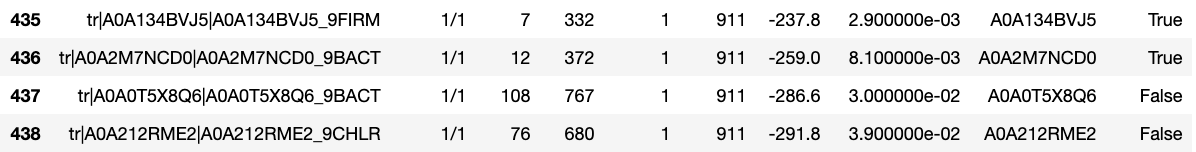
Confusion matrix
| True hits | False hits | |
|---|---|---|
| Not rejected | 437 | 0 |
| Rejected | 0 | 186462 |
Достаточно установить порог -260, даже такого низкого значения будет достаточно, чтобы разделить истинные белки с нужной доменной архитектурой от ложных. Это может быть из-за того, что я выбрал очень специфичные домены.
©Бакулин Артемий, 2018