Практикум 8. Нуклеотидный BLAST
Поиск в геноме эукариота ген, кодирующий δ-субъединицу АТФ-синтазы
В таблице белковых последовательностей для сборки генома Anas platyrhynchos (protein.faa) было найдено 3 записи для δ-субъединицы АТФ-синтазы: XP_038024941.1 (с этой записью дальше проводилась работа), XP_038024942.1, XP_071885323.1. Можно заметить, что их аминокислотные последовательности полностью идентичны, поэтому дальнейшая работа проводилась с одной из них
Файл с последовательностью δ-субъединицы АТФ-синтазы: FASTA файл
Идентификатор белка: XP_038024941.1
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок: NC_092615
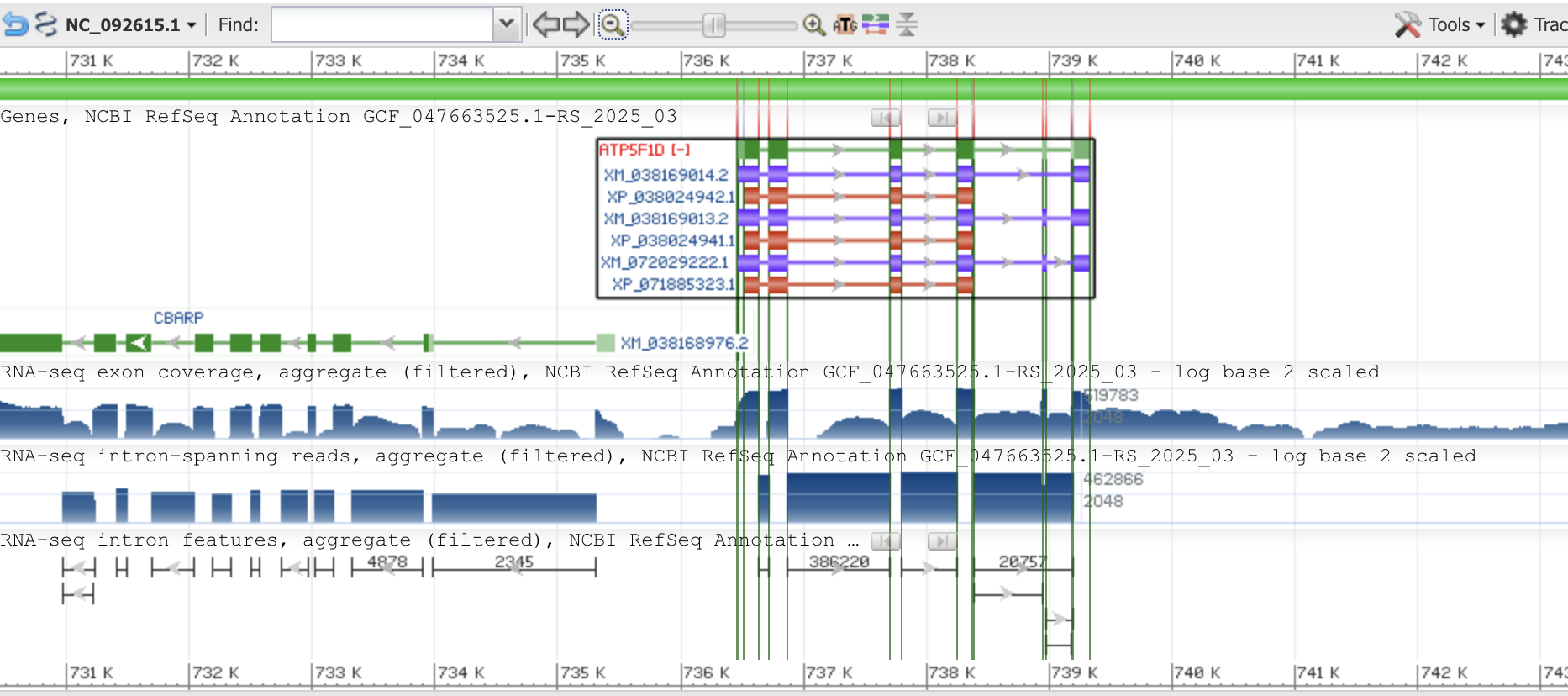
На рисунке 1 можем заметить ген, который находиться рядом с изучаемым: CBARP. Также 3 белка: XP_038024941.1, XP_038024942.1, XP_071885323.1, которые транслируются с соответствующих транскриптомов: XM_038169014.2, XM_038169013.2, XM_072029222.1
Ссылка на файл с последовательностью кодирующей белок части в FASTA-формате (с небольшой последовательностью до и после): FASTA файл
Координаты кодирующей белок части гена:736465..739327
Разные варианты BLAST для фрагмента ДНК
Для выполнения задания было выбрано семейство первичноротых животных, а именно - Пчёлы (Apoidea), для того, чтобы поиск был среди заведомо эволюционно очень далеких организмов. Следовательно, ожидается совсем не большое количество находок (предположим не больше 30-50)
В качестве базы данных для поиска был выбран вариант RefSeq Genome Database (refseq_genomes)
Поиск был проведен 2 методами: blastn и tblastx, по последовательностям геномов из выбранного таксона из базы данных refseq genomes.
blastn использует нуклеотидную последовательность для поиска сходств среди нуклеотидной базы данных. Данный метод был выбран, для того, чтобы посмотреть какие участки смогут оказаться схожими (мое предположение, что вероятность того, что megablast найдет схожие участки больше, поэтому интересно было проверить именно с помощью blastn)
tblastn использует белковую последовательность для поиска сходств среди транслированной нуклеотидной базы данных. Был использован для того, чтобы сравнить число находок по сравнению с blastn, при поиске по белковой последовательности, а не нуклеотидной
Метод blastn использовался с параметром word size 7, так как поиск проводился эволюционно далеких организмов, с данным параметром чувствительность для поиска больше (по умолчанию стоит word size 11). Было найдено 22 последовательности, для поиска использовались нуклеотидные последовательности
Число сборок в RefSeq Genome Database, входящих в выбранный таксон: 38
Число находок: 22
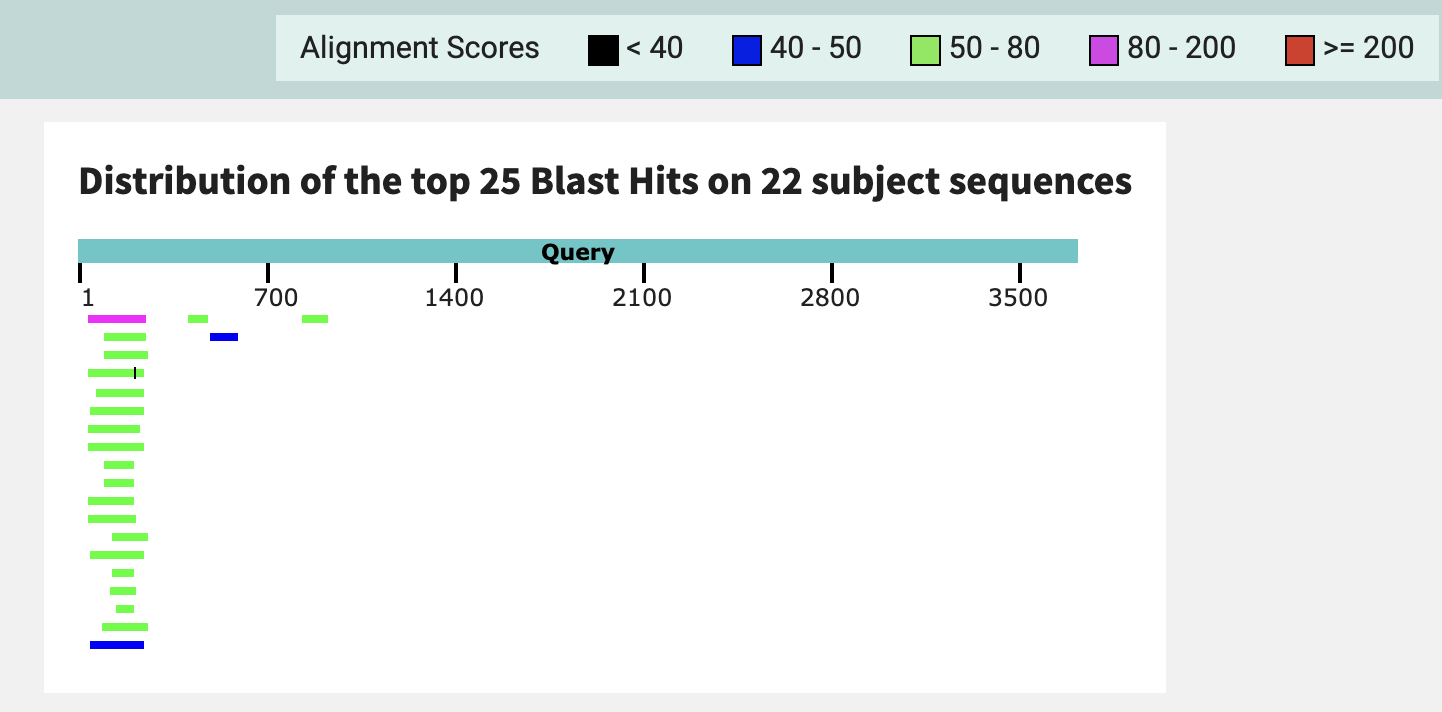
На рис. 2 мы видим, что большинство совпавших участков имеют длину небольшую длину и счет выравнивания менее 80, что является ожидаемым, так как используемые таксоны имеют сильно отличающиеся нуклеотидные последовательности.
Результаты поиска в текстовом виде: текстовый файл
Метод tblastn использовался с параметром word size - 5 (дополнительно также с параметром word size 3, где было получено такое же количество находок). Были использованы белковые последовательности.

На рис. 3 мы видим, что большинство совпавших участков имеют длину меньше 100 нуклеотидов, счет выравнивания всех находится в диапозоне 80-200.
Число сборок в RefSeq Genome Database, входящих в выбранный таксон: 38
Число находок: 38
Результаты поиска в текстовом виде: текстовый файл
Нахождение в геноме эукариота гены основных рибосомальных РНК по далекому гомологу
Создание локальной базы данных nucleotide:
makeblastdb -in GCF_047663525.1_IASCAAS_PekinDuck_T2T_genomic.fna -dbtype nucl -out nucleotide
Затем были скачены последовательности рРНК Escherichia coli
Локальный поиск BLAST отдельно для каждой рРНК:
blastn -task blastn -query 23s_RNA.fasta -db nucleotide -out 23s_RNA.out -word_size 7 -outfmt 7
blastn -task blastn -query 16s_RNA.fasta -db nucleotide -out 16s_RNA.out -word_size 7 -outfmt 7
Пояснения: -task указывает какой алгоритм blast использовать, формат 7 = Tabular with comment lines
Результаты выравнивания с помощью метода blastn:
16s RNA 23s RNA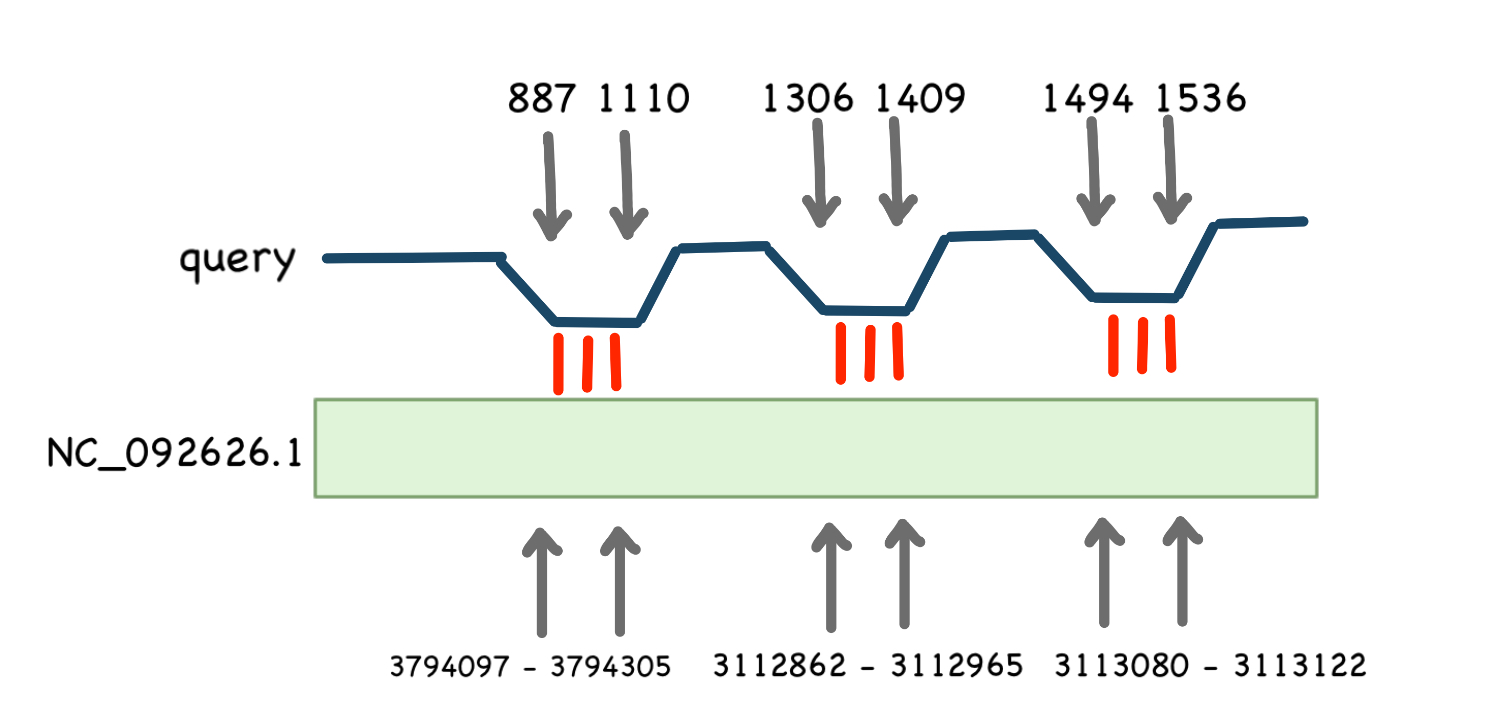
На рис. 4 можно заметить, что последовательность 16s рРНК Escherichia coli оказалась гомологичной "+" цепи 38 хромосомы Anas platyrhynchos(mallard duck) в 3 участках. Интересно, что данные координаты соответствуют гену: LOC113841281 18S ribosomal RNA, что дополнительно подтверждает результаты выравнивания.
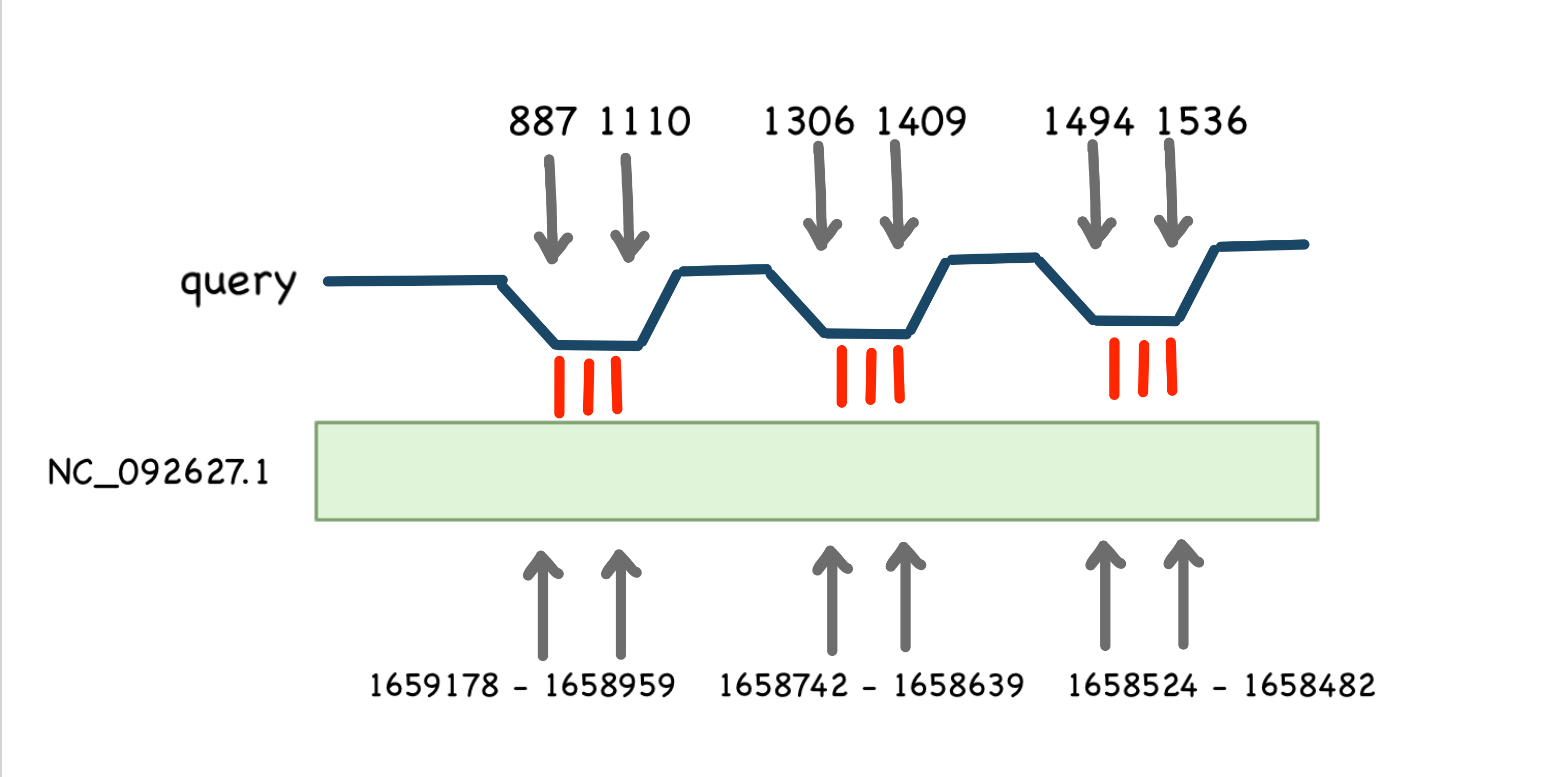
На рис. 5 можно заметить, что последовательность 16s рРНК Escherichia coli оказалась гомологичной "-" цепи 39 хромосомы Anas platyrhynchos(mallard duck) в 3 участках. Интересно, что данные координаты соответствуют гену: LOC140001278 18S ribosomal RNA, что дополнительно подтверждает результаты выравнивания.
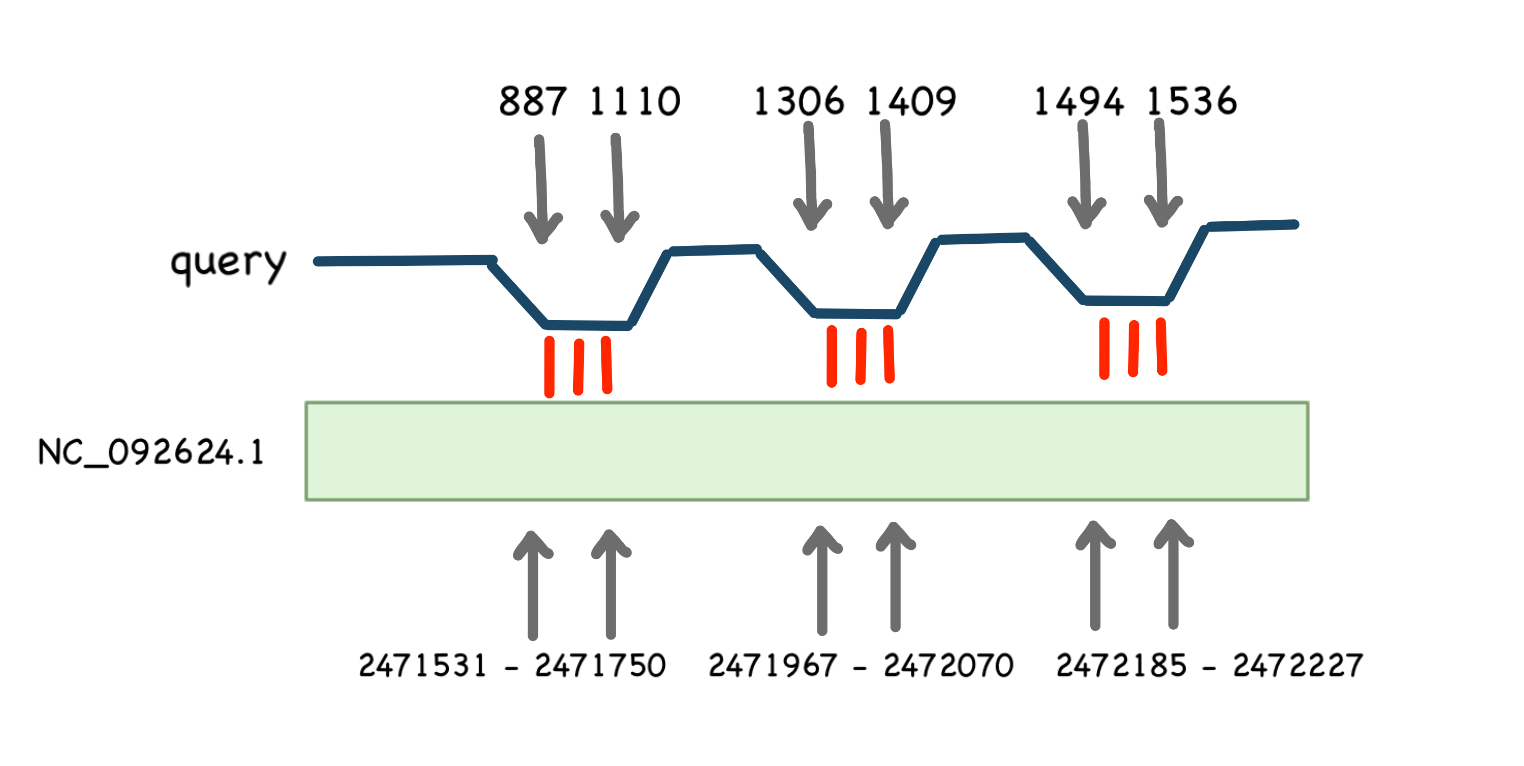
На рис. 6 можно заметить, что последовательность 16s рРНК Escherichia coli оказалась гомологичной "+" цепи 36 хромосомы Anas platyrhynchos(mallard duck) в 3 участках. Интересно, что данные координаты соответствуют гену: LOC140001033 18S ribosomal RNA, что дополнительно подтверждает результаты выравнивания.

На рис. 7 можно заметить, что последовательность 16s рРНК Escherichia coli оказалась гомологичной "+" цепи 31 хромосомы Anas platyrhynchos(mallard duck) в 3 участках. Интересно, что данные координаты соответствуют гену: LOC140000088 18S ribosomal RNA, что дополнительно подтверждает результаты выравнивания.
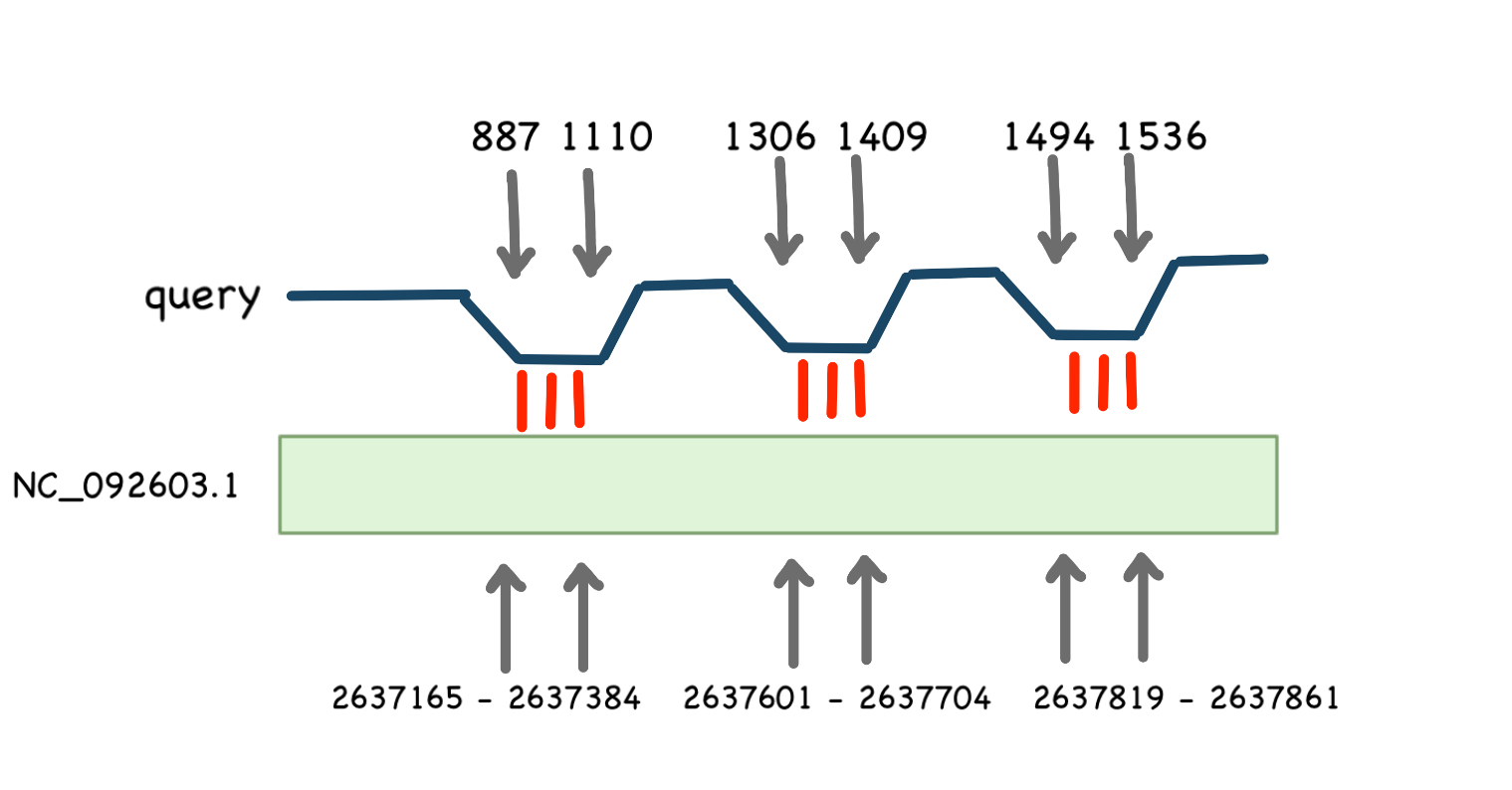
На рис. 8 можно заметить, что последовательность 16s рРНК Escherichia coli оказалась гомологичной "+" цепи 17 хромосомы Anas platyrhynchos(mallard duck) в 3 участках. Интересно, что данные координаты соответствуют гену: LOC139998969 18S ribosomal RNA, что дополнительно подтверждает результаты выравнивания.
Подборка пары геномов и построение карты их локального сходства
В рамках этого задания были выбраны геномы: Blochmannia endosymbiont of Polyrhachis (Hedomyrma) turneri и Blochmannia endosymbiont of Colobopsis nipponica
Были выбраны небольшие геномы, для того чтобы запустить tblastx. Подбирались штаммы одного вида, для которых dotplot, построенный с помощью blastn и megablast отличается. Таким образом, были найдены последовательности, для которых точности megablast недостаточно.
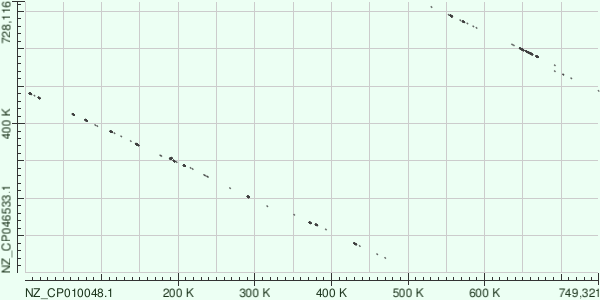
На рис. 9 видим только, что у штаммов бактерий не совпали точки, с которых секвенировали геном.

На рис. 10 мы можем заметить, инверсию, которую megablast пропустил, то есть мы доказали, что megablast бывает не всегда максимально точным.
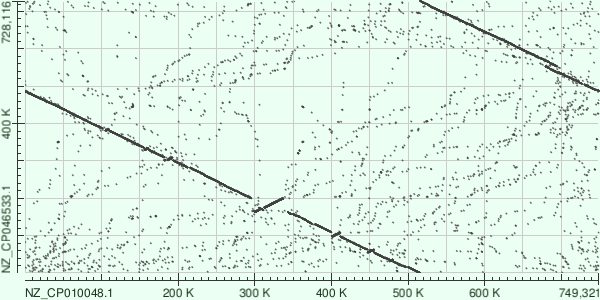
На рис. 11 можем заметить большое количество мелких сходств. Таким образом, можно прийти к выводу, что tblastx обладает максимальной достоверностью, но в процессе выполнения задания были найдены существенные недостатки: ограниченная длина последовательностей и долгое время работы.
Дополнение
Поиск проводился по последовательностям 23s и 16s RNA Escherichia coli. Входящая в малую субъединицу рибосомы, 16s RNA интересна тем, что на ее основе было построенно филогенетическое дерево, также она содержит последовательность, комплементарную последовательности Шайна-Дальгарно, необходимую для распознавания рибосомой старт-кодона. 23s RNA входит в состав большой субъединицы рибосомы, участвует в формировании A, P, E сайтов, необходимых для трансляции.
Для 16s RNA получилось найти 5 гомологов, так как было проверено, что действительно координаты выравненной последовательности утки соответствуют генам 18s RNA.
Для 23s RNA были найдены следующие гены:
Таким образом, было найдено 5 гомологов.