Доменная структура белка YDAF_BACSU по данным Pfam
(по-русски! и желательно с кратким пояснением)
Описание доменной архитектуры белка в соответствии с банком Pfam
С главной страницы Pfam доступны разные виды поиска. В частности, "JUMP TO" позволяет искать по ID белка.
Cхема из Pfam: |
|||||
| Пояснения к схеме |
|||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов (по-русски! и желательно с кратким пояснением) |
Положение в последовательности белка YDAF_BACSU | Клан |
| 1. | PF00583 | Acetyltransf_1 | Семейство ацетилтрансфераз (GNAT). Включает в себя N-ацетилтрансферазные белки, такие как Elp3-связанные белки.
Трансформируют ацетил-группу в гистонах.Семейство доменов названо по названию фермента индол-3-глицерофосфатсинтетазы, |
72-151 | Клан Acetyltrans (CL0257), содержит 30 семейств; у 6 из них неизвестна функция. |
2. Привожу некоторые данные о домене:
1. Домен входит в 299 архитектур.
2. Последовательность известна для 35080 белков, содержащих домен.
3. Пространственная структура определена для 47 разных белков.
4. Сохраняем выравнивание "seed" фрагментов белков, соответствующих домену: seed.msf
(меню "Alignments", формат MSF, Seed (а не Full — все последовательности), "Generate").
3. Описание доменной архитектуры с двумя доменами.
Выбираем архитектуру MarR_2, Acetyltransf_1. Схема: 
Переходим на страничку домена. "Species", далее – "Tree".
К сожалению, для найденного домена моего белка дерево сгенерировать не удалось ни в каком виде (3248 видов). Это говорит о том,
что домен представлен очень широко. Для другого домена все получилось. Выбираем "Expand to depth" = 2.
Получаем дерево. Первая цифра возле названия таксона означает количествово видов,
вторая – количествово последовательностей белков с доменом, третья – общую представленность домена в последовательностях.
|
Таксон
|
Количество белков с доменом PF07687.
|
|
| Эукариоты | Зеленые растения (Viridiplantae) | 5 |
| Грибы (Fungi) | 2 | |
| Животные(Metazoa) | 0 | |
| Археи (Archaea) | 193 | |
| Бактерии (Bacteria) | 7334 | |
| Вирусы (Viruses) | 1 | |
| Неклассифицированные | 16 | |
Домен больше всего представлен у бактерий (7334 белка в 1779 видах). Встречается и у эукариот, но довольно редко.
4. Сравнение описаний мотивов в разных банках семейств, по данным InterPro.
Откроем главную страничку InterPro. По ID UniProt белка найдем описание всех подписей (signatures), интегрированных в InterPro, т.е. имеющих InterPro ID. Получаем:
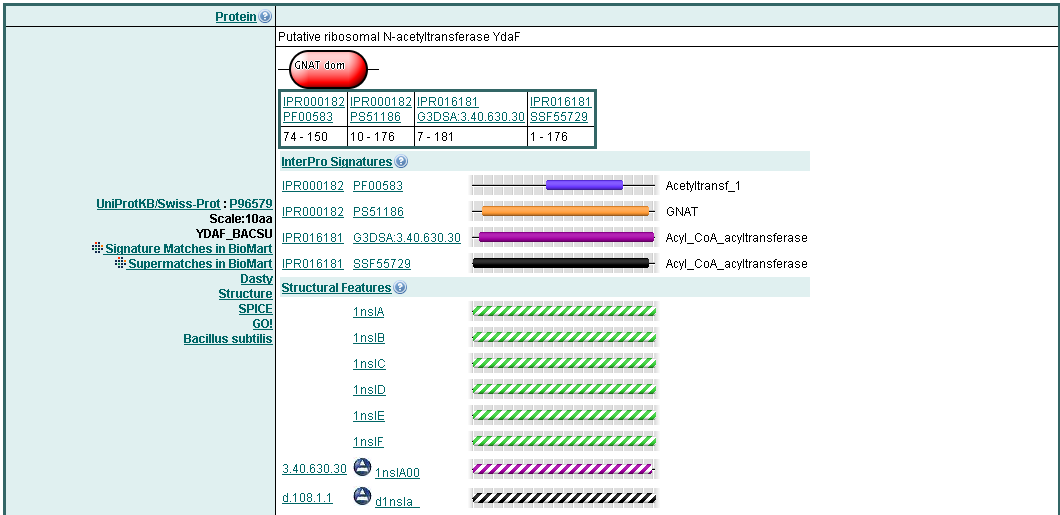
1. Самый короткий мотив называется Acetyltrans, он описан в банке Pfam.
2. Самый длинный мотив называется Acyl_CoA_acyltransfer, описан в банке SuperFamily.
3. В InterPro интегрируемы следующие структурные подписи: 1nslA, 1nslB, 1nslC, 1nslD, 1nslE, 1nslF, 1nslA00, d1nsla (d.108.1.1).
4. Границы структурных доменов от границ доменов Pfam сильно отличаются. Они намного шире (1-183 или 1-179).