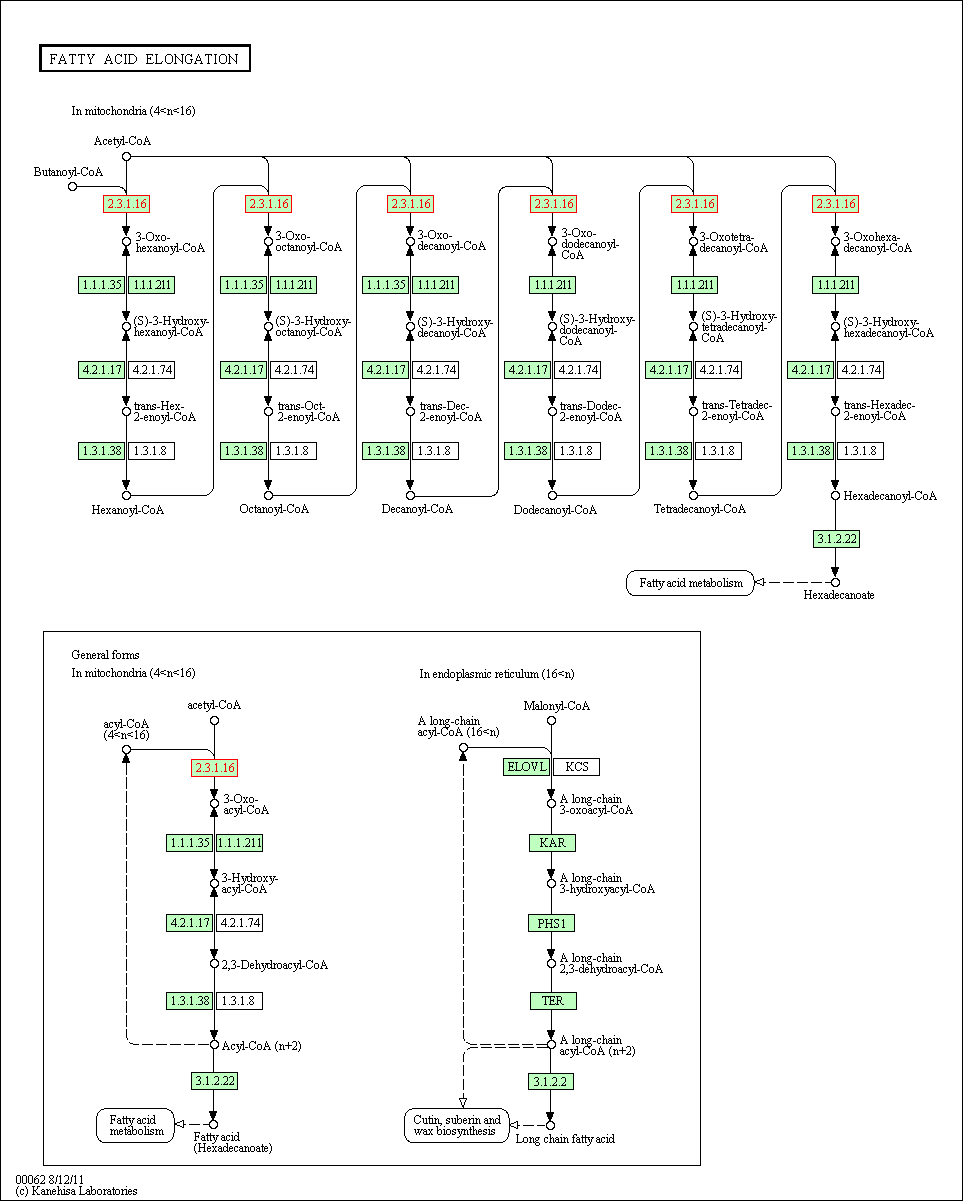
EC: 2.3.1.16
Идентификатор KEGG - hsa:3032
hsa00062, fatty acid elongation, элонгация жирных кислот
hsa00071, fatty acid metabolism, метаболизм жирных кислот
hsa00280, valine, leucine and isoleucine degradation, деградация валина, лейцина
и изолейцина
hsa01100, metabolic pathways, метаболические пути
| Русское название | Английское название | ID | Структурная формула |
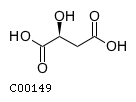
| (S)-малат | (S)-malate | C00149 |
 |
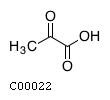
| Пируват | Pyruvate | C00022 |
 |
3. Поиск метаболического пути от одного заданного вещества к другому
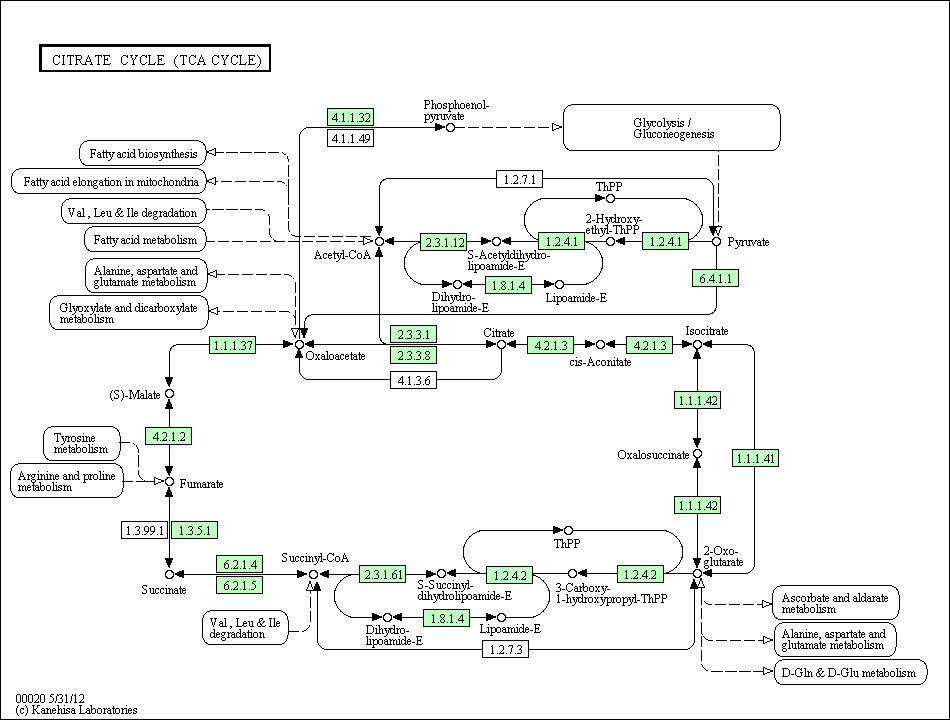
Выбранный путь: Citrate cycle (TCA-cycle)
Цепочка: пируват -> (S)-малат.
Промежуточное соединение - щавелевоуксусная кислота.
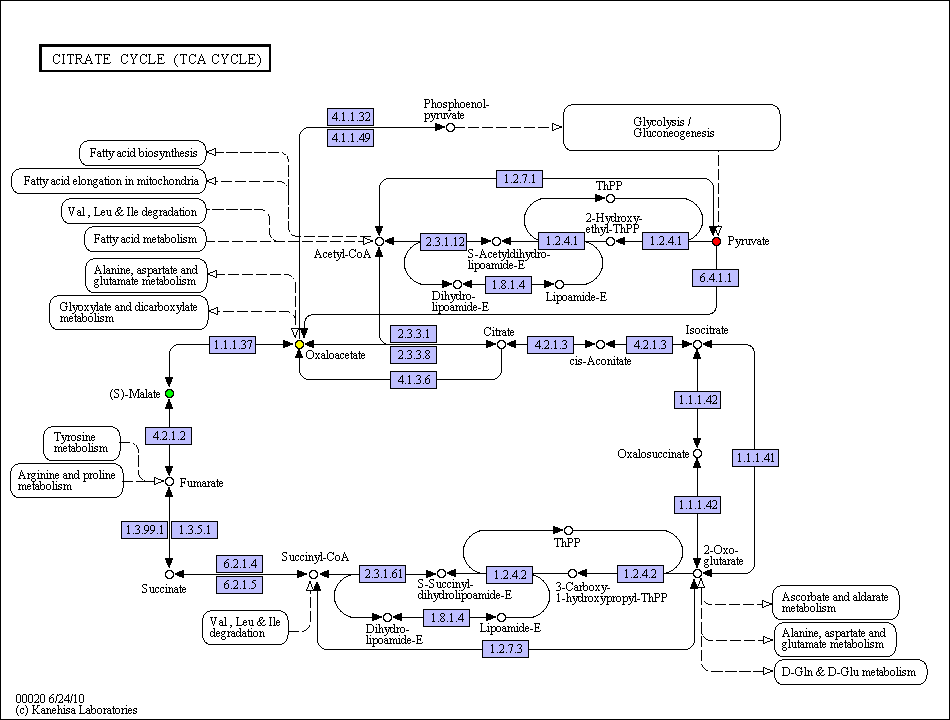
4. Сравнение метаболических путей у разных организмов
| Организм | Цепочка реакций возможна | Обоснование |
| (да/нет/неизвестно) | ||
| Bacillus subtilis | да | есть все ферменты (источник) |
| Archaeoglobus fulgidus | да | есть все ферменты (источник) |
| Arabidopsis thaliana | да | есть все ферменты (источник) |
| Homo sapiens | да | есть все ферменты (источник) |
У всех всех организмов есть ферменты 6.4.1.1 и 1.1.1.37. Ниже приведено изображение TCA-cycle для человека.