| PDB код |
Тип |
Мембрана |
Толщина гидрофобной части мембраны, Å |
Медиана числа остатков в одном трансмембранном участке |
| 1xio |
Спираль |
Внутренняя мембрана Anabaena sp. |
31.9 ± 1.5 |
22 |
| 2j8c |
Спираль |
Внутренняя мембрана Rhodobacter sphaeroides |
29.4 ± 1.1 |
18.5 |
| 4b4a |
Спираль |
Внутренняя мембрана Aquifex aeolicus |
30.4 ± 1.2 |
22 |
| 1qjp |
Баррель |
Внешняя мембрана Escherichia coli |
25.4 ± 1.5 |
9 |
| 2fgg |
Баррель |
Внешняя мембрана Comamonas acidovorans |
25.0 ± 0.9 |
8 |
| 3qra |
Баррель |
внешняя мембрана Yersinia pestis |
25.2 ± 1.4 |
8 |
Задание №1: отбор гомологов
Поиск гомологов проведен с помощью PSI-BLAST.
После 4 итераций с порогом 1e-5 отобрано было 16 гомологов из разных таксонов.
Названия последовательностей отредактированы, создан fasta-файл.
Задание №2: анализ структуры выданного белка
Использована база данных OPM для поиска выданного белка.
| PDB ID |
Организм |
Тип мембраны |
TC-код |
Наклон спиралей к нормали |
Количество трансмембранных спиралей |
Название белка |
| 4AV6 |
Thermotoga maritima |
Внутренняя мембрана |
3.A.10.1 |
0 ± 0°
Отдельно для каждой цепи:
9°, 9°.
|
32 (2 субъединицы) |
K-активируемый натриевый насос |
Описание полей TC-кода:
3.* - первичные активные транспортёры
3.A.* - работающие за счет гидролиза P-P-связей
3.A.10.* - H+, Na+-транспотрирующая пирофосфатаза
3.A.10.1 - K-стимулируемая
Задание №3: анализ множественного выравнивания трансмембранных белков
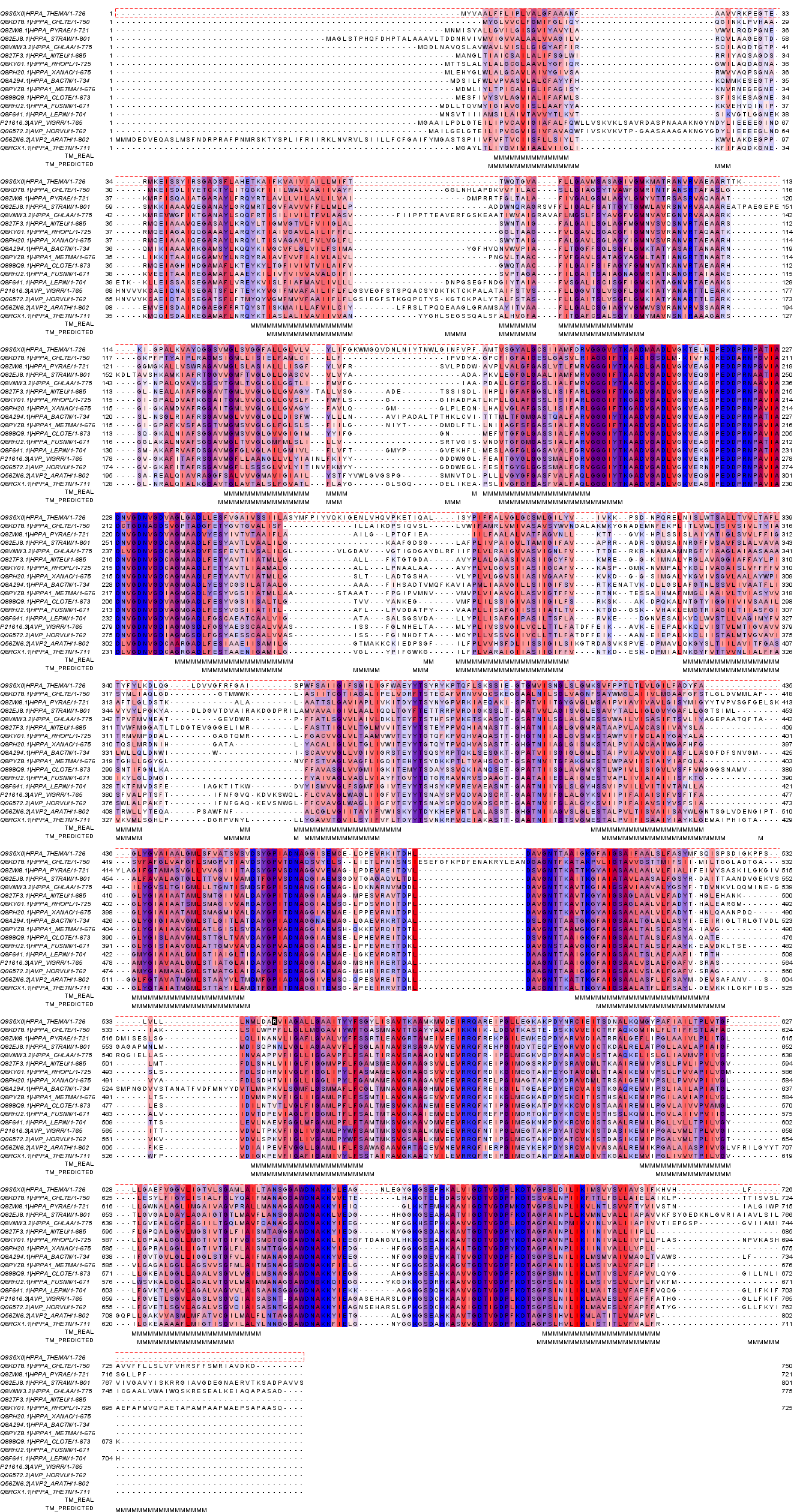
Выравнивание построено программой Muscle и импортировано в Jalview.
Добавлена аннотация положения трансмембранных спиралей.
Помечены участки выравнивания, отвечающие трансмембранным спиралям в строке-аннотации TM_REAL буквой "М".
Добавлено предсказание трансмембранных спиралей, выдаваемых программой TMHMM, для гомолога HPPA_CHLTE.
Эти участки помечены в строке TM_PREDICTED. Получено изображение структуры:
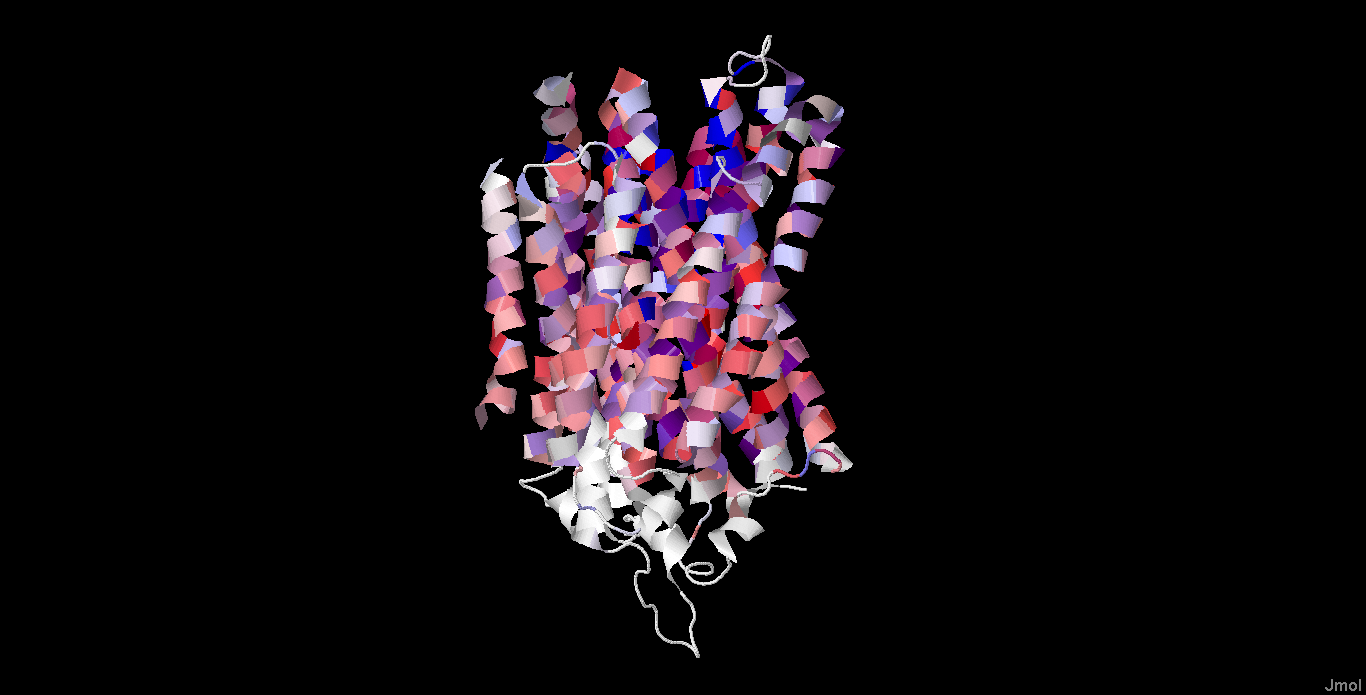 Применена окраска Hydrophobicity, c порогом консервативности 20%.
Самые гидрофобные остатки - красные, а самые гидрофильные - синие.
Цвета на структуре белка отвечают выбранной цветовой схеме.
Часть белка, ориентированная в n-сторону мембраны, расположена сверху, а ориентированная в p-сторону - снизу.
Применена окраска Hydrophobicity, c порогом консервативности 20%.
Самые гидрофобные остатки - красные, а самые гидрофильные - синие.
Цвета на структуре белка отвечают выбранной цветовой схеме.
Часть белка, ориентированная в n-сторону мембраны, расположена сверху, а ориентированная в p-сторону - снизу.
Проведем анализ результатов. Можно заметить, что консервативность на участках трансмембранных спиралей высокая.
Трансмембранные участки состоят в основном из гидрофобных остатков (лейцин, валин, изолейцин, фенилаланин),
а так же встречаются высококонсервативные полярные остатки (глицин, серин, глутамин) и заряженные
(глутаминовая к-та, аспарагиновая к-та, аргинин, лизин).
Возможно, они отвечают за функциональную роль белка. Между спиралями встерчаются и консервативные,
и неконсервативные участки, там преобладают заряженные и полярные остатки.
Результаты TMHMM хорошо пересекаются реальной структурой, но кое-где границы не совпадают.
# Q9S5X0|HPPA_THEMA/1-726 Length: 726
# Q9S5X0|HPPA_THEMA/1-726 Number of predicted TMHs: 13
# Q9S5X0|HPPA_THEMA/1-726 Exp number of AAs in TMHs: 314.08282
# Q9S5X0|HPPA_THEMA/1-726 Exp number, first 60 AAs: 26.01802
# Q9S5X0|HPPA_THEMA/1-726 Total prob of N-in: 0.02491
# Q9S5X0|HPPA_THEMA/1-726 POSSIBLE N-term signal sequence
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 1 3
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 4 26
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 27 59
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 60 82
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 83 124
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 125 147
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 148 166
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 167 189
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 190 244
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 245 267
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 268 287
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 288 310
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 311 324
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 325 344
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 345 356
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 357 379
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 380 423
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 424 446
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 447 499
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 500 522
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 523 541
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 542 564
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 565 611
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 612 631
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 outside 632 635
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 TMhelix 636 658
Q9S5X0|HPPA_THEMA/1-726 TMHMM2.0 inside 659 726
Функции у моего белка и гомолога немного различны; возможно, смещение произошло из-за различий в структурах белков.
 Применена окраска Hydrophobicity, c порогом консервативности 20%.
Самые гидрофобные остатки - красные, а самые гидрофильные - синие.
Цвета на структуре белка отвечают выбранной цветовой схеме.
Часть белка, ориентированная в n-сторону мембраны, расположена сверху, а ориентированная в p-сторону - снизу.
Применена окраска Hydrophobicity, c порогом консервативности 20%.
Самые гидрофобные остатки - красные, а самые гидрофильные - синие.
Цвета на структуре белка отвечают выбранной цветовой схеме.
Часть белка, ориентированная в n-сторону мембраны, расположена сверху, а ориентированная в p-сторону - снизу.
