Пространственная структура yDAF_BACSU (PDB ID 1NSL)
Заголовок структуры: TRANSFERASE
Название структуры: CRYSTAL STRUCTURE OF PROBABLE ACETYLTRANSFERASE
В документе представлены следующие цепи макромолекул:
| Идентификатор цепи | Число остатков | Название молекулы | Комментарии |
| A | 184 | PROBABLE ACETYLTRANSFERASE | REMARK 465 MISSING RESIDUES THE FOLLOWING RESIDUES WERE NOT LOCATED IN THE EXPERIMENT. (RES=RESIDUE NAME; C=CHAIN IDENTIFIER; SSSEQ=SEQUENCE NUMBER; I=INSERTION CODE.) RES C SSSEQI ALA A 41 GLU A 42 ASN A 43 PRO A 44 GLU A 180 GLY A 181 GLU A 182 LYS A 183 ALA B 41 GLU B 42 ASN B 43 PRO B 44 GLU B 180 GLY B 181 GLU B 182 LYS B 183 GLU C 180 GLY C 181 GLU C 182 LYS C 183 GLU D 180 GLY D 181 GLU D 182 LYS D 183 ALA E 41 GLU E 180 GLY E 181 GLU E 182 LYS E 183 ALA F 41 GLU F 42 ASN F 43 GLU F 180 GLY F 181 GLU F 182 LYS F 183 |
| B | 184 | PROBABLE ACETYLTRANSFERASE | |
| C | 184 | PROBABLE ACETYLTRANSFERASE | |
| D | 184 | PROBABLE ACETYLTRANSFERASE | |
| E | 184 | PROBABLE ACETYLTRANSFERASE | |
| F | 184 | PROBABLE ACETYLTRANSFERASE |
| ID | Название | Формула | Число молекул | Русское название |
| MSE | SELENOMETHIONINE | C5 H11 N O2 SE | 18 (по 3 в каждой цепи) | Селенметионин |
| CL | CHLORIDE ION | CL 1- | 6 (по одной в каждой цепи) | Хлорид-ион |
| HOH | WATER | H2 O1 | 19 | Вода |
Создание скрипта
С помощью программы RasMol был создан скрипт, последовательно генерирующий заданные изображения структуры белка. Скрипт создаёт следующие картинки.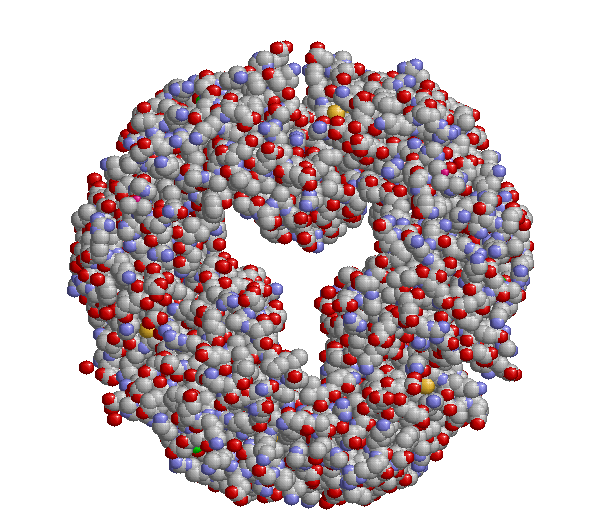 1. Вся структура в шариковой модели
1. Вся структура в шариковой модели
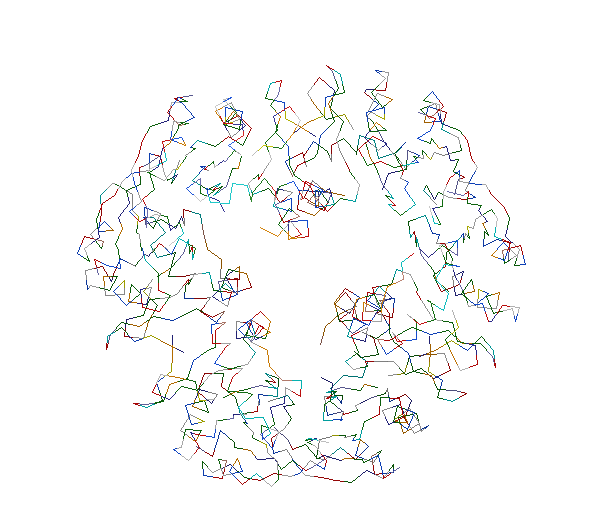 2. Структура только белка в остовной модели
2. Структура только белка в остовной модели
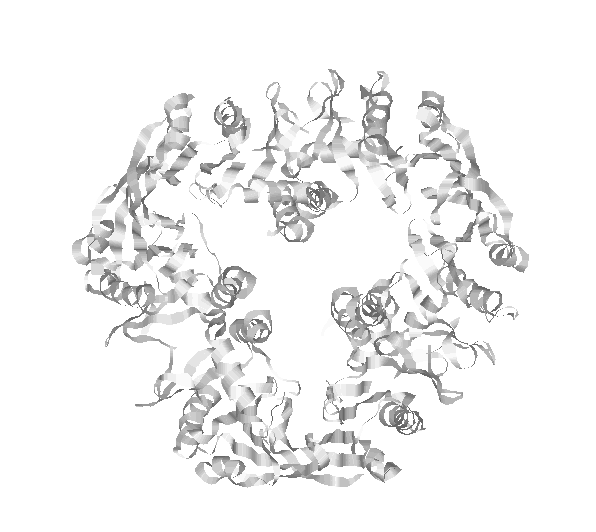 3. Структура только белка в ленточной модели
3. Структура только белка в ленточной модели
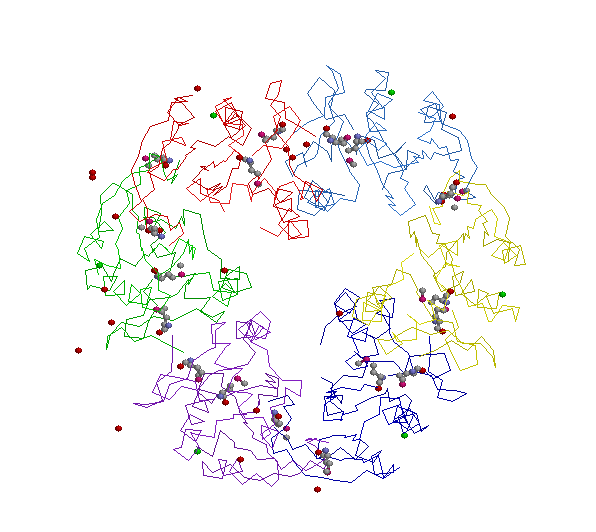 4. Изображение всех компонентов структуры
4. Изображение всех компонентов структуры
Поскольку последние остатки цепи не обнаружены, взял в качестве конца цепи последний обнаруженный остаток – триптофан. Первым же возьмем селенметионин (он идет под числом 1, а не 0, как указано в последовательности pdb-файла для глицина, который обозначен как CLONING ARTIFACT)
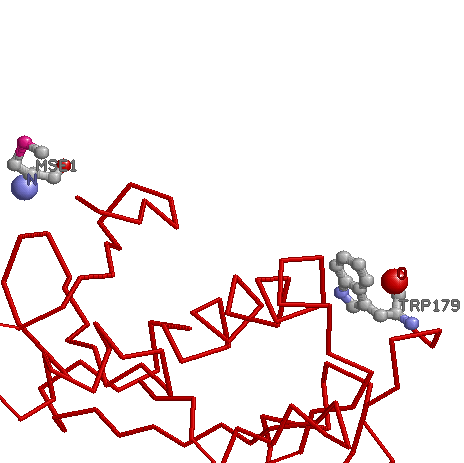 5. Изображение концевых остатков у одной полипептидной цепи
5. Изображение концевых остатков у одной полипептидной цепи
Расчеты
Аппроксимируем белок наиболее подходящей фигурой – цилиндром с диаметром основания 80 ангстрем и высотой в 40 ангстрем. Переведя все значения в более удобные нанометры, получаем 8 и 4 нм соответственно. Объем равен V=π*(8/2)2*4=201.0619≈201 нм3
Кишечную палочку мы также представим в виде цилиндра, с радиусом 500 нм и высотой 2000 нм. V=π*500 Если предположим, что все 4400 белков были синтезированы одновременно и в равных количествах, то количество каждого белка равно 7814907/4400=1776,115≈1776 молекул (опять-таки, не может же отдельно существовать 0,115 молекулы белка).
При вычислении размера для видимого под световым микроскопом объекта для увеличения площади разместим молекулы таким образом, чтобы они плотно и основаниями вверх заполнили круг, и возьмем длину световой волны за 500 нм. Предел разрешения тогда будет 250 нм, площадь круга S=π*2502=196349,541 нм2. Площадь основания цилиндра молекулы белка S=π*42=50,265 нм2. При допущении, что при заполнении круга основаниями цилиндров не остается промежутков, получаем 196349,541/50,265=3906,287≈3906 молекул белка.
В ChemSketch я по совету уважаемого Андрея Викторовича строил изображение метионина (так как тринадцатым остатком в белке является глицин), который (внезапно!) отсутствует в моем белке, Поэтому решил сделать задание на основе аланина.
Мы видим, что в полученной в программе RasMol структуре отсутствует атом кислорода. Он потребовался для образования воды при образовании пептидной связи. (Действительно, ведь в ACD ChemSketch мы «рисовали» саму аминокислоту, а не остаток.) Также мы видим, что во втором изображении отсутствуют атомы водорода, будучи слишком малыми для распознавания при анализе структуры белка.
Сравнение изображений в ACD ChemSketch и RasMol
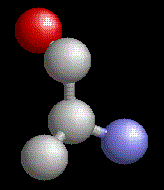 Аланин в RasMol’е
Аланин в RasMol’е
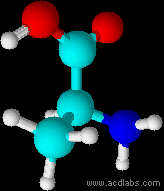 Аланин в ChemSketch’е
Аланин в ChemSketch’е
PBD файл белка
Скрипт