Укоренение в среднюю точку
P.S. Методом UPGMA уже получаем укоренённое дерево, укоренять повторно - бессмысленно.
- Скопируем файл retree.exe с директории P:/y11/Term_4/PHYLIP в рабочую директорию.
- Скобочную форму дерева, полученную методом "Neighbour Joining Using % Identity", сохраним в файле intree
- Запустим программу retree, подтвердив параметры по умолчанию нажатием кнопки "Y".
- Укореним дерево в среднюю точку нажатием кнопки "M".
- Завершим работу с программой нажатием "X", после чего выберем сохранить полученное дерево кнопкой "Y", причём оставим его укоренённым (кнопка "R").
- Результат запишется в файл outtree. После этого нажмём "Enter" для выхода из программы.
- Изображение дерева, полученное с помощью программы Mega:

- Оригинальное неукоренённое дерево:
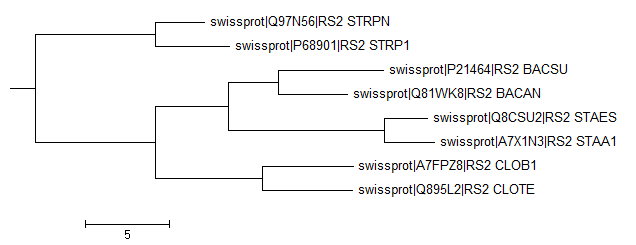
- Результаты:
- Укоренение произошло в ветвь {STRP1, STRPN} против {CLOTE, CLOB1, BACAN, BACSU, STAA1, STAES}
- Данное укоренение вряд ли можно считать правильным, поскольку организмы рода Streptococcus филогенетически ближе к организмам родов Bacillus и Staphylococcus (принадлежат к одному классу), в отличие от организмов рода Clostridium, что получилось в случае укоренённого дерева.
- При укоренении удалилсь идентификаторы организмов в названии белка. Пришлось впоследствии дописывать их вручную.
- Кроме того, скобочная форма стала записываться в виде 4х строчек, вместо одной.
Использование внешней группы
- Добавим к файлу последовательностей 30S рибосомных белков последовательность RS2_ECOLI, после чего выровняем последовательности программой muscle и получим файл rs2_align_new.fasta, в котором оставим в качестве имён последовательностей лишь идентификаторы организмов
- Откроем полученный файл в программе Mega и построем дерево методом "Maximum Parsimony".
- Укореним полученное дерево в ветвь {ECOLI}vs{CLOTE, CLOB1, STRP1, STRPN, STAA1, STAES, BACAN, BACSU}, выбрав "Root" в меню "Subtree".
- Уберём из дерева последовательность белка E. coli с помощью кнопки "Show Subtree Separately".
- Полученное дерево:
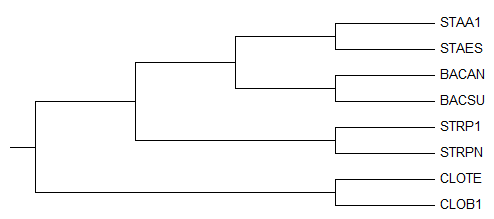
- Результаты:
- Укоренение произошло в ветвь {CLOTE, CLOB1} против {STRP1, STRPN, BACAN, BACSU, STAA1, STAES}.
- Данное укоренение - наиболее правильное, так как полученное дерево совпадает с верным, и каждая ветвь соответствует выделяет какой-либо таксон.
Бутстрэп
При построении очередного дерева в программе Mega методом "Maximum Parsimony" в меню "Phylogeny Test" выберем "Bootstrap Method", указав число реплик, равное 100.
Полученные деревья:
| Original tree |
Bootstrap consensus tree |
 |
 |
Дерево, построенное без применения метода "Bootstrap" ближе к правильному ввиду полного совпадения. Однако, если учитывать только нетривиальные ветви, то в обоих деревьях совпадают 5 из 5 таковых.
Таким образом, в полученных реконструкциях отсутствуют неправильные нетривиальные ветви.




