| |
Для него в ncbi Genome нашлось три сборки. При том две из них опубликованы одновременно группой из Мельбурнского университета 11 июня 2014 (BioProject ID PRJNA208415 ), но для организмов разных полов.
Три проекта по секвенированию с девятью образцами.
Сборка GCA_000701005.1 - одна из тех двух для разных полов - содержит больше всего белков. Для этой сборки использовался образец с BioSmple ID SAMN02423286 - взрослый 49-дневный самец Trichuris suis. (Всего в проекте было 6 образцов.)
В таблице 1 представлены характеристики сборки. По ссылке можно посмотреть таблицу контигов. В таблице 2 представлены ссылки на файлы самым коротким и самым длинным контигом.
| Табл.1. Информация о сборке GCA_000701005.1. |
Табл.2. Файлы с контигами. |
 |
| |
Длина |
Ссылка на файл с последовательностью |
| Самый длинный контиг |
468,935 |
JMHY01000143 |
| Самый короткий контиг |
73 |
JMHY01000942 |
|
Задание 2
В этом задании надо было описать транскриптом выданного мха. Мне достался Sphagnum palustre - сфагнум болотный. В России он встречается повсеместно в заболоченных лесах и на болотах. Например, на Симе. (Рис.2.)
По запросу Sphagnum palustre genome refseq в ncbi Nucleotide выдается единственный результат - запись с полным геномом моего мха в базе данных RefSeq с ID NC_024521.1 . Всего в геноме 40 генов кодируют белки и 27 - РНК, среди которых 24 тРНК и 3 рРНК.
По ссылке можно скачать таблицу с генами сфагнума болотного, отсортированными по началу в геноме. |
 Рис.2. Sphagnum palustre - моя фотография с летней практики на ЗБС. |
Задание 3
На странице сайта ncbi, посвященной GenBank есть ссылка на release notes . В этом файле приведены help'ы, 10 из которых описаны ниже.
Примеры взяты из этой записи ( ссылка на ncbi ).
| regulatory - регуляторный участок. GC-участок встречается в промоторах постоянно работающих генов. |
 |
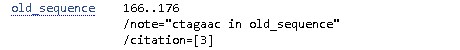
| old_sequence - повторяет раннее описанную версию последовательности. |
 |
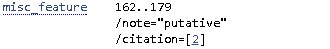
| misc_feature - участок с биологической значимостью, которому не соответствует ни один из ключей. |
 |
| source - организм-источник генетической последовательности. |
 |
| prim_transcript - участок, на котором синтезируется первичный транскрипт. |
 |
| gene - участок с элементами, контролирующими начало и конец считывания или без, кодирующий функциональный ген, у которого есть название. |
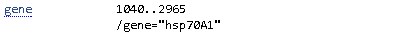 |
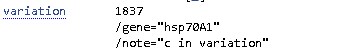
| variation - популяция содержит устойчивую мутацию. |
 |
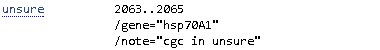
| unsure - авторы не уверены в последовательности этого участка. |
 |
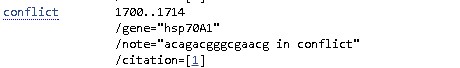
| conflict - на этом участке есть различия в независимых результатах определения последовательности. |
 |
| CDS - кодирующая последовательность со стоп-кодоном. |
 |
Задание 4
В ncbi с помощью BLASTN найдены следующие последовательности, похожие на чистую последовательность из практикума 6. (Рис.3-4)
Первые находки из одного таксона и их процент идентичности исходной последовательности равен 99. Наверняка это и есть она - последовательность гена цитохромоксидазы 1 из Ophiopholis aculeata.
Таксонимия, полученная из ncbi:taxonomy cellular organisms; Eukaryota; Opisthokonta; Metazoa; Eumetazoa; Bilateria; Deuterostomia; Echinodermata; Eleutherozoa; Asterozoa; Ophiuroidea; Ophiuridea; Ophiurida; Ophiurina; Gnathophiurina; Gnathophiuridea; Ophiactidae; Ophiopholis
Т.к. первыми идут последовательности из одного таксона, для построения парных выравниваниваний с исходной последовательностью были выбраны не самые лучшие находки. Они отмечены галочками. С находками из других таксонов: Ophiopholis japonica и Ophiopholis kennerlyi 86% и 84% совпадающих позиций. Парные и множественное выравнивание(ссылка на jvp-проект) исходной последовательности с выбранными получены в JalView с помощью Muscle и Pairwise Alignment.
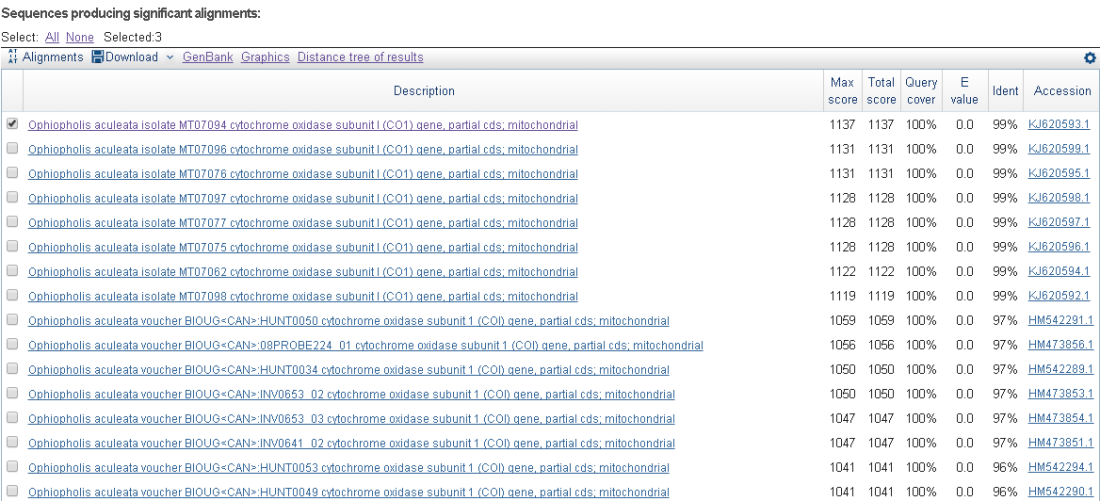
 Рис.3-4. Находки BLASTN. Первая галочка соответствует первой находке. Вторая и третья - 36 и 37.
|