На главную
Третий семестр
Упражнения
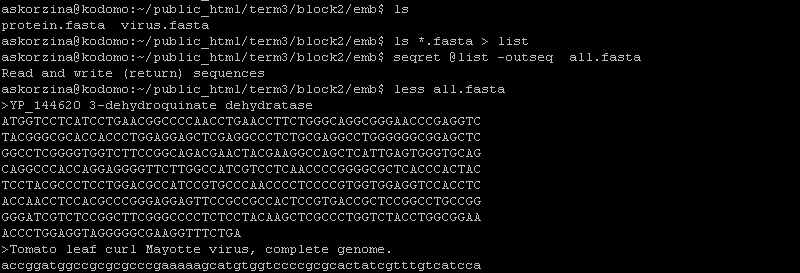
Для выполнения упражнений взяла последовательности белка и вируса из первого семестра.1) seqret объединяет несколько файлов в формате fasta в единый(virus.fasta и protein.fasta в all.fasta).
|
Меню
На главную Третий семестр |
УпражненияДля выполнения упражнений взяла последовательности белка и вируса из первого семестра.1) seqret объединяет несколько файлов в формате fasta в единый(virus.fasta и protein.fasta в all.fasta). 
|
|
2) seqretsplit разделяет файл с несколькими последовательностями на отдельные fasta файлы(all.fasta на tomato.fasta и yp_144620.fasta). 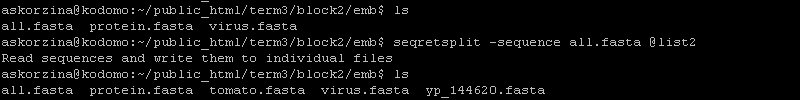
10) shuffle - создает новый файл из того же набора нуклеотидов. 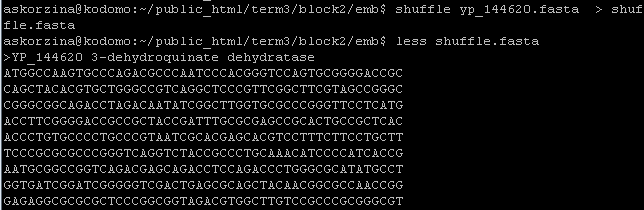
11) cusp - считает частоты кодонов в входных последовательностях. 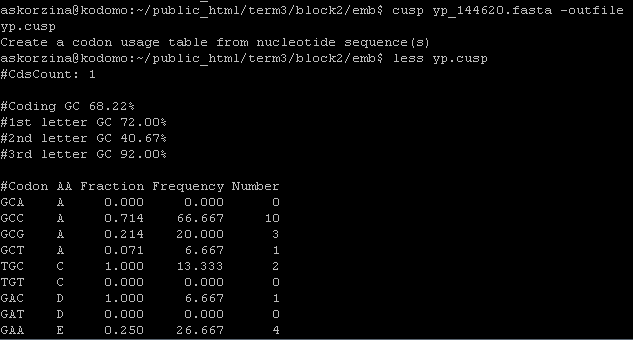
12) compseq - считает частоты динуклеотидов. (Можно ввести длину слова 3 и посчитать частоту кодонов.) 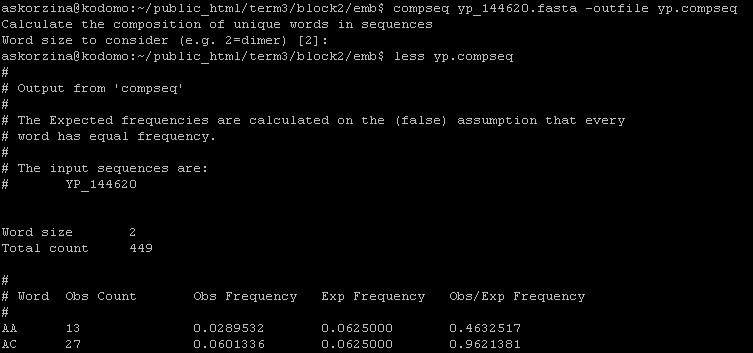
|
Задание 1.В этом практикуме я работала с бактерией из первого семестра - Thermus Thermophilus HB8(NC_006461). Для получения списка трансляций открытых рамок были использованы следующие команды.(Рис.2) getorf из пакета EMBOSS ищет открытые рамки и выдает файл в формате .orf(Рис.1), который можно открыть с помощью Excel. Каждая запись в таком файле содержит две строки: на первой информация о рамке считывания(имя входного файла_номер записи, координаты [начала - конца] считывания, организм, из которого взята последовательность), на второй - последовательность.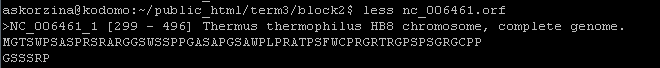 Рис.1. Пример записи в файле .orf, полученном с помощью getorf. Опции к getorf:-sequence - fasta-файл с последовательностью, в которой надо найти открытые рамки; -outseq - имя выходного файла; -table - выбор из меню, к какой группе относится организм: 11 - бактерия; -minsize - минимальная длина считанных последовательностей в парах нуклеотидов; -find 1 - считывание от стоп-кодона до стоп-кодона; -circular - кольцевая хромосома. Опции к infoseq: -only - выдает только те столбцы, которые идут после этой опции; -name - столбец с именем записи; -description - столбец с описанием, в которое входят координаты рамки и организм-источник последовательности; -sprotein1 -length - столбец с длиной белка, который может соответствовать этой записи. Команды для преобразования файла в требуемый вид: echo - записывает нужные названия столбцов в nc.xlsx; grep - выводит только строки, содержащие "NC_", т.е. убирает ненужные название столбцов; awk {'gsub(/*/,"**"); print $i, $j'} - заменяеет символ * на **, выводит из строки только i-ое и j-ое слова, в том же порядке, в котором они стоят после print; sort -nk N - сортирует числа по N столбцу. 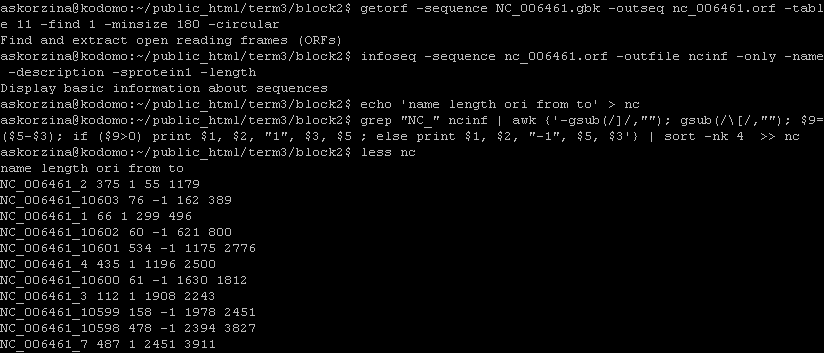 Рис.2. Команды, использованные для получения таблицы координат открытых рамок считывания. С помощью Excel сохранила таблицу в формате .xlsx.Задание 2.Из архива old refseq на ncbi скопировала файлы NC_006461.faa с последовательностями белков и NC_006461.ptt со списком генов белков.awk - удаляет ненужные столбцы и меняет местами нужные, чтобы они были в таком же порядке, что и в таблице, полученной getorf; tail -n+K - выдает все строки файла, начиная с К-ой. 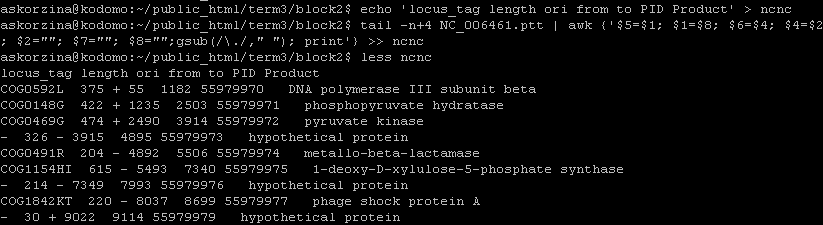 Рис.3. Команды, использованные для преобразования таблицы генов белков. Полученная Excel-таблица.Задание 3. Сравнение таблиц.Общая таблица. В ней аннотированные белки в фиолетовых ячейках, а открытые рамки считывания в оранжевых.Генов белков записей в 5 раз меньше, чем записей в таблице с открытыми рамками считывания. Причина в том, что не каждой открытой рамке соответствует белок. Длина на три нуклеотида меньше в последовательностях из таблицы getorf, потому что при поиске открытых рамок не учитывается стоп-кодон. На длине белка это не отражается. (Рис.4-5) 

Рис.4-5. Разница в длине открытых рамок на прямой(сверху) и обратной(снизу) цепи. На рис. 6 представлен ген белка и соответсвующий ему ORF. Но здесь разница есть не только в стоп-кодоне, но и в старт-кодоне. У бактерий их несколько. ORF начинается с самого первого, поэтому она и длиннее. Рис.6. Разница в длине из-за разных старт-кодонов. Рамка длиной 276 соответствует белку. Внутри нее и рядом есть другие рамки, которые не соответствуют белку. (Рис.7)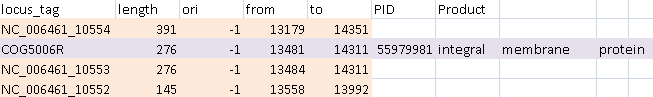 Рис.7. На месте одного белка сразу три открытых рамки разной длины. Сортировка по длине последовательности помогла найти все белки, которым точно не будет соответсвующей рамки считывания, потому что длина рамки оганичена снизу 180 нуклеотидами. (Рис.8,9) Но они могут быть частью какой-нибудь более длинной рамки.(Рис.10)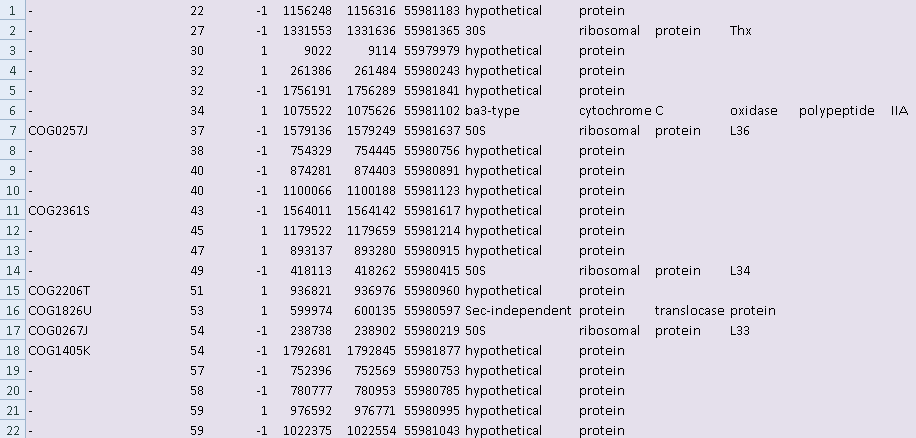 Рис.8.Белки длиной меньше 60 аминокислот.  Рис.9 Белок, которому не нашлось ORF на обратной цепи, но нашлось на прямой.  Рис.10.Белок, который попал в ORF подлиннее. Перекрывание открытых рамок на антипараллельных цепяхНа рисунке 11 показаны перекрывающиеся открытые рамки. Длииа указана в аминокислотах, поэтому длина перекрывания больше 150 п.н. Белок находится на прямой цепи, рамки есть и на прямой, и на обратной.В этом месте находится ген белка фосфопируват гидратазы.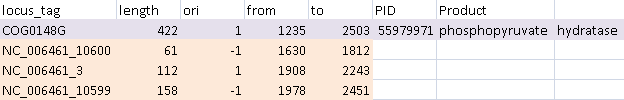
Рис.11. Перекрывающиеся открытые рамки. |
© Корзина Анастасия, 2015