Получение нуклеотидных последовательностей рибосомальной РНК 16S
В базе геномов NCBI в файлах с расширением .frn хранятся нуклеотидные последовательности РНК. Оттуда они были скопированы в общий файл для построения выравнивания в JalView с помощью Muscle.Для бактерии Bradyrhizobium diazoefficiens не нашлось генома по названию, но нашлись 4 вида этого же рода. Чтобы выбрать вид, был найден идентификатор этой бактерии - NC_004463. С такими же названиями лежат файлы в директории Bradyrhizobium japonicum_USDA_110. Значит, это второе название Bradyrhizobium diazoefficiens.
Дерево было построено алгоритмом Maximum Likelyhood в программе MEGA.(Рис.1) Совпадает с правильным, полученным на первом занятии.
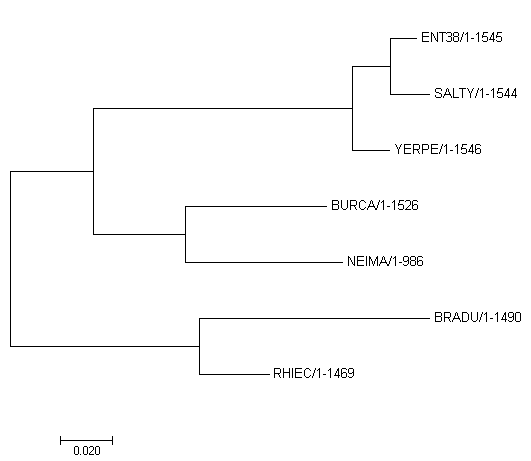
Рис.1. Дерево для последовательностей 16S РНК.
Ветви дерева
Дерево содержит пять нетривиальных ветвей:1. (BRADU, RHIEC) против (BURCA, NEIMA, YERPE, ENT38, SALTY)
2. (BRADU, RHIEC, BURCA, NEIMA) против (YERPE, ENT38, SALTY)
3. (BRADU, RHIEC, BURCA, NEIMA, YERPE) против (ENT38, SALTY)
4. (BRADU, RHIEC, YERPE, ENT38, SALTY) против (BURCA, NEIMA)
5. (BRADU, RHIEC, ENT38, SALTY) против (YERPE, BURCA, NEIMA)
Таксономия
| Название | Мнемоника | Таксономия |
| Bradyrhizobium diazoefficiens | BRADU | cellular organisms; Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Bradyrhizobiaceae; Bradyrhizobium |
| Rhizobium etli | RHIEC | cellular organisms; Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium |
| Burkholderia cenocepacia | BURCA | cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia cepacia complex |
| Neisseria meningitidis | NEIMA | cellular organisms; Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria |
| Enterobacter sp. 638 | ENT38 | cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Enterobacter |
| Salmonella typhimurium | SALTY | cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Salmonella; Salmonella enterica; Salmonella enterica subsp. enterica |
| Yersinia pestis | YERPE | cellular organisms; Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia; Yersinia pseudotuberculosis complex |
Нетривиальные ветви отражают разбиение бактерий на классы:
1. Alphaproteobacteria - (BRADU, RHIEC)
2. Betaproteobacteria - (BURCA, NEIMA)
3. Gammaproteobacteria - (YERPE, ENT38, SALTY)
Укоренение в среднюю точку
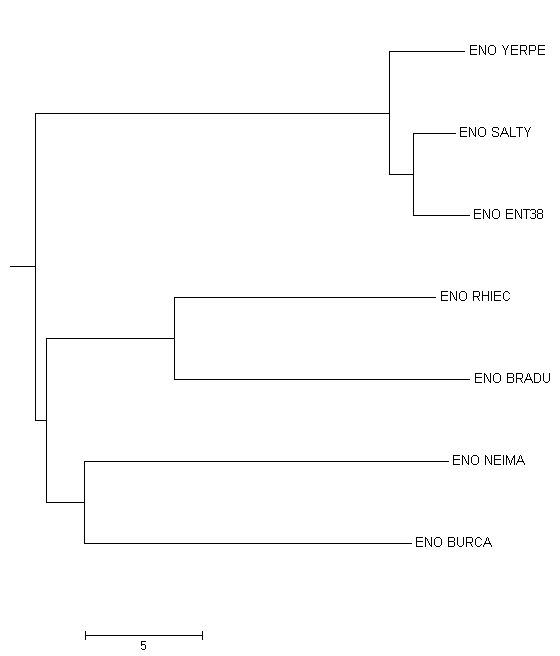
Рис.2. Дерево, полученное после укоренения.
Укоренение снова привело к тому, что альфапротеобактерии(BRADU, RHIEC) и бетапротеобактерии(BURCA, NEIMA) оказались на одной ветви, а гаммапротеобактерии(YERPE, ENT38, SALTY) на другой, хотя в "правильном" дереве альфабактерии отстоят от общей ветви бетапротеобактерий и гаммапротеобактерий.Задание 2. Построение и анализ дерева, содержащего паралоги
Два гомологичных белка будем называть ортологами, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования. Два гомологичных белка из одного организма будем называть паралогами.Двумя красными скобками показаны две ортологические группы, внутри которых все белки являются ортологами друг другу. Для всех организмов, кроме NEIMA, белки CPLX и HSLU являются паралогами. Для организма YERPE так же есть еще два паралога. Все паралоги YERPE подчеркнуты синими линиями на рисунке 3.
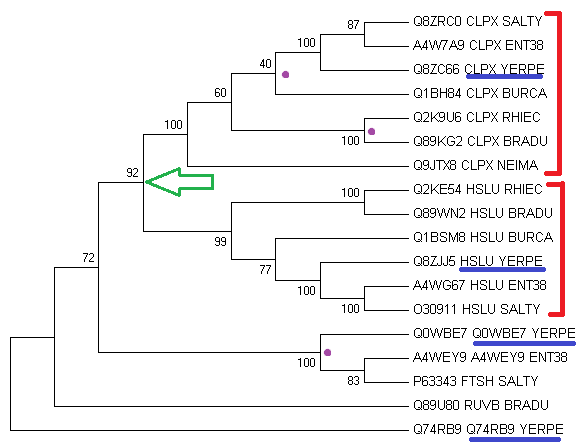
Рис.3. Дерево гомологов белка CLPX.
Примеры эволюционных событий:- дупликация гена(зеленая стрелка указывает на разветветвление, разделяющее две ортологичные группы)
- разделение путей эволюции белков в результате видообразования(примеры таких разветвлений отмечены фиолетовыми точками)