| |

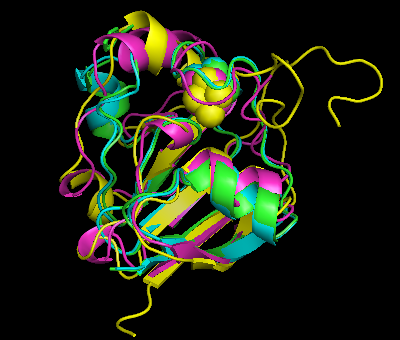
Рис.2. Выравнивание гомологов ферредоксина, построенное по структурам .

Рис.3. Выравнивание гомологов ферредоксина, построенное Muscle.
| Выравнивание, построенное Muscle, на первый взгляд кажется более успешным, потому содержит больше консервативных колонок. Несовпадающие консервативные колонки между выравниваниями были проверены.
Колонка №13 (Muscle) содержит все глицины, но в выравнивании по структуре видно, что эти глицины не расположены в одинаковых позициях. GLY 12 из гомолога с PDB ID 1a70 не находится рядом с остальными глицинами (Рис.4).
Колонка №66 (Muscle) не является консервативной, тогда как в структуре валины или лейцины всех гомологов находятся рядом и занимают одну позицию в выравнивании по структуре (Рис.5). Таким образом, структурное выравние лучше, потому в правильных позициях отражает сходство между белками.
|

Рис.4. Пример несоответствия выравниваний по структуре и по последовательности. Консервативная колонка №13 (Muscle) не выровнена по структуре.
|
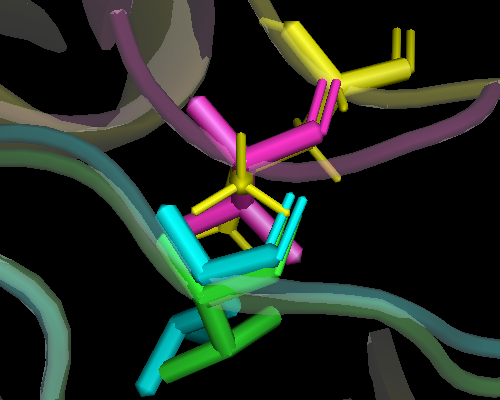
Рис.5. Пример несоответствия выравниваний по структуре и по последовательности. Неконсервативная колонка №66 (Muscle) выровнена по структуре.
|
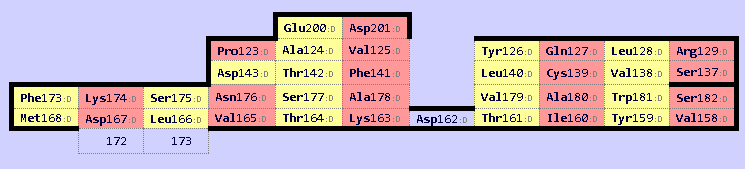
Рис.6. Бета-лист из альфа-цепи T-клеточного рецептора. Консервативный цистеин в позиции 139.
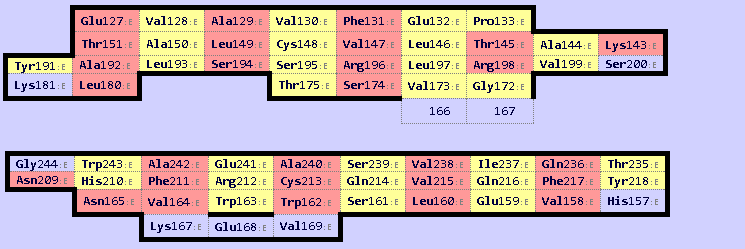
Рис.7. Бета-листы из бета-цепи T-клеточного рецептора. Верхний бета-лист соответствует листу из альфа-цепочки. Консервативный цистеин в позиции 148.
|
|
Были выбраны два домена с разных цепей константного домена T-клеточного рецептора. PDB ID: 1bd2.
Координаты выбранных доменов:
d:118-203
e:119-247
С помощью сервиса SheeP
были построены карты бета-листов (Рис.6-7). Лист из альфа-цепочки (цепь d) соответствует верхнему листу из бета-цепочки (цепь e).
Консервативный цистеин в альфа-цепи №139 и в бета-цепи №148.
Далее было построено выравнивание структур по этому цистеину.
Также для выравнивания были использованы остатки перед и после этих цистеинов, потому что они тоже консервативны. Остатки, спаренные с цистеинами неконсервативные, (а функция pair_fit требует одинакового количества атомов,) поэтому для выравнивания были использованы только их остовные углероды CA.
|
Команда в PyMOL для построения выравнивания:
select D1, chain D and resi 138-140 or chain D and resi 127+180+160 and name CA
select E1, chain E and resi 147-149 or chain E and resi 130+195+175 and name CA
pair_fit E2, D2
Файл с выравниванием.
Домены хорошо выравниваются в районе бета-листов с консервативными цистеинами, но при этом другие структуры не совпадают (Рис.8).
Например, один из бета-тяжей бета-цепи в выровненных бета-листах, соответствует петле в альфа-цепи.
Таким образом, можно сделать вывод, что топология доменов различна.
| |
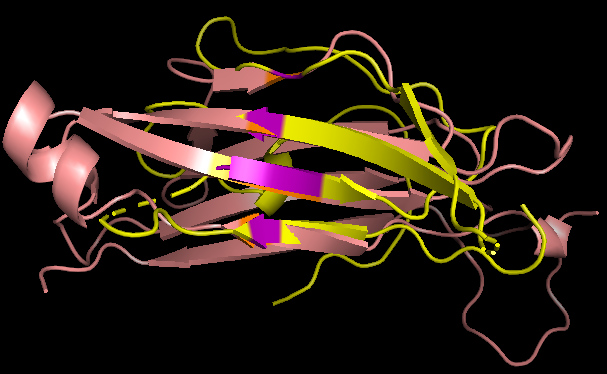 Рис.8. Выравнивание структур доменов с разных цепей T-клеточного рецептора. Желтым выелена альфа-цепь, розовым - бета-цепь. Участки, по которым строилось выравнивание, выделены фиолетовым на альфа-цепи и оранжевым на бета-цепи.
|
|
|