1) Скачиваем последовательности с заданными идентификаторами с UniProt в FASTA-формате.
2) Открываем JalView. Нам необходимо объединить ве скаченные последовательности в один файл. Для этого: File, затем нажимаем Add sequences и From File.
3) Теперь строим множественное выравнивание: Web Service, затем - Alignment и Muscle with Defaults.
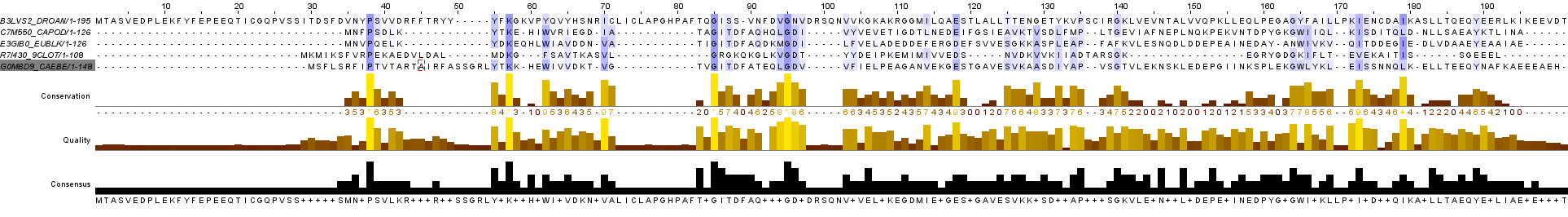
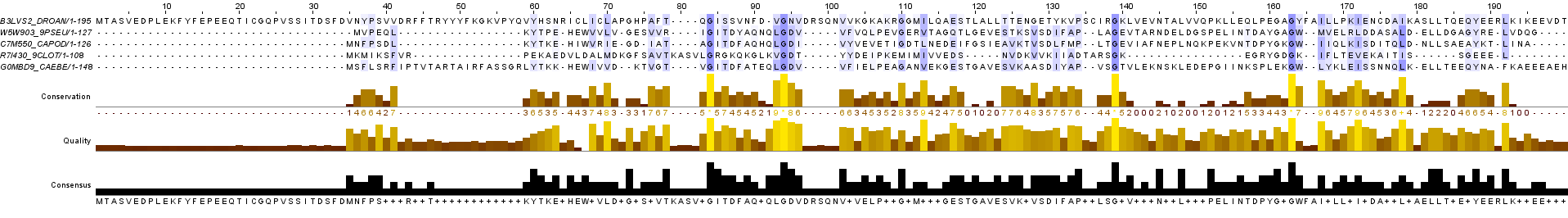
| В данном множестве последовательностей невозможно найти последовательность, сильно отличающуюся от остальных. Чтобы выявить не гомологичную остальным, я рассмотрела все возможные комбинации выравниваний из 5 последовательностей. Наибольшее число совпадений получила в 2 случаях: при удалении белков с идентификаторами E3GIB0_EUBLK и W5W903_9PSEU. Но все же более 4 консервативных колонок с полным совпадением во всех 5 последовательностях я не нашла. В случае удаления белка с идентификатором W5W903_9PSEU мы получаем на одну консервативную колонку со сходными аминокислотами больше, чем при удалении белка с идентификатором E3GIB0_EUBLK. Поэтому я размещу изображения 2 выравниваний далее. Таким образом: |
Идентификатор лишней последовательности: W5W903_9PSEU или E3GIB0_EUBLK.
Рис.1 Выравнивание без белка с идентификатором W5W903_9PSEU в раскраске BLOSUM62 с порогом по консервативности 30%
 |
Рис.2 Выравнивание без белка с идентификатором E3GIB0_EUBLK в раскраске BLOSUM62 с порогом по консервативности 30%
 |
Ссылки на выравнивание, полученное во втором задании:
Fasta: Без белка с идентификатором W5W903_9PSEU и без белка с идентификатором E3GIB0_EUBLK.
MSF: Без белка с идентификатором W5W903_9PSEU (1) и без белка с идентификатором E3GIB0_EUBLK(2).
Ссылка на проект JalView с 5 окнами - выравнивание из первого задания (окно: 6_seq_alignment) + 2 выравнивания из задания 2 в раскрасках BLOSUM62 (окна: Blosum62 для случая (1) и Blosum62-2 для случая (2)) и ClustalX (окна: Clustalx-1 и Clustalx-2).
© Kalashnikova Anastasia, 2016