
|
Мне был выдан контиг с идентификатором NW_013546656. В данном контиге не нашлось генов с альтернативным сплайсингом.
Поэтому был взят скэффолд № 3 из резерва (РЕЗЕРВ3) с идентификатором NW_016683393.
Латинское название: Xenopus tropicalis; Русское название: Когтистая шпорцевая лягушка; Таксономия: Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi; Amphibia; Batrachia; Anura; Pipoidea; Pipidae; Xenopodinae; Xenopus; Silurana. Размер скэффолда в парах оснований: 1742295 bp; Число генов: 27; Число белок-кодирующих последовательностей (CDS): 40. Ссылка на файл скэффолда в формате fasta |
Рис. 1 - Xenopus tropicalis[1]
 |
| Краткая характеристика вида: единственный диплоидный вид в роде шпорцевых лягушек. Геном был полностью секвенирован, поэтому организм используют в качестве модельного. Место обитания: тропики и субтропики. Охотятся на мелких беспозвоночных. |
Рис. 2 - Ген с альтернативным сплайсингом (для увеличения необходимо нажать на картинку)
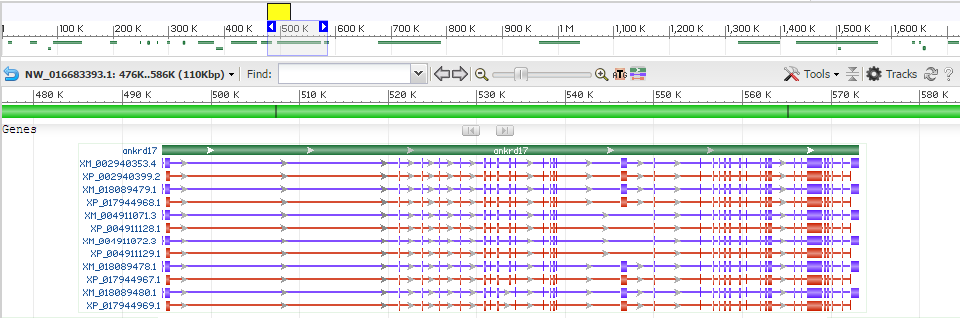 |
| Был найден вручную ген (ankrd17), для которого был предсказан альтернативный сплайсинг по базе данных. На рис. 2 представлен данный ген. Здесь присутствует несколько изоморф - наиболее значимые связаны с отсутствием/наличием экзона в промежутке с 546235 по 546987. |
| С помощью web-сервера AUGUSTUS были предсказаны гены и белок-кодирующие области в данном скэффолде. В качестве организма для параметров модели был выбран Gallus gallus domesticus. Выбор организма: были отобраны позвоночные, начиная с рыб, но исключая человека, и было построено дерево с помощью Taxonomy Browser на NCBI (рис. 3). Остальные параметры запуска - по умолчанию. |
Рис. 3 - Taxonomy tree
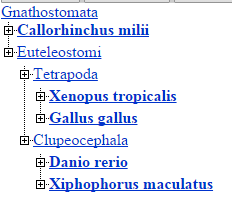 |
| Был выдан predictions.tar.gz (архив), который при распаковке выдает следующие файлы: |
| Название файла | Описание | Ссылка на файл |
| augustus.aa | Файл с транслированными из генов аминокислотными последовательностями (fasta) | Ссылка |
| augustus.cdsexons | Файл с экзонами предсказанных генов (fasta) | Ссылка |
| augustus.codingseq | Файл с кодирующими последовательностями (fasta) | Ссылка |
| augustus.gbrowse | Файл с координатами генов, м-РНК и т.д. (fasta) | Ссылка |
| augustus.gff | Файл с предсказанными генами в формате gff | Ссылка |
| augustus.gtf | Файл с предсказанными генами в формате gtf | Ссылка |
|
Программа AUGUSTUS предсказала 64 гена против 27, заявленных GenBank. Причем, было выявлено всего одно полное соответствие (gene 6 в GenBank - координаты: 249050..250585). Таким образом, можно сделать вывод, что программа выдает довольно малый процент верно найденных генов. Совпадение предсказания AUGUSTUS и данных GenBank для гена, приведенного в задании 1, не наблюдается. Не совпадает ни одна координата - ни начальная, ни конечная, зато совпадает направление цепи. Можно предположить, что различия в предсказаниях вызваны тем, что на роль организма для параметров модели был выбран организм эволюционно далекий от исходного организма. |
© Kalashnikova Anastasia, 2016