|
В данном упражнении необходимо было предсказать вторичную структуру тРНК путем поиска инвертированных повторов.
Программа einverted из пакета EMBOSS позволяет найти инвертированные участки в нуклеотидных последовательностях.
Было необходимо подобрать параметры, с помощью которых мог бы быть получен наиболее соответствующий реальности результат.
В результате многочисленных попыток был найден самый приемлимый результат: 1gts_b-2.inv .
Параметры запуска: Gap penalty: 9; Match score: 5; Mismatch score: -6; Minimum score threshold: 15. |
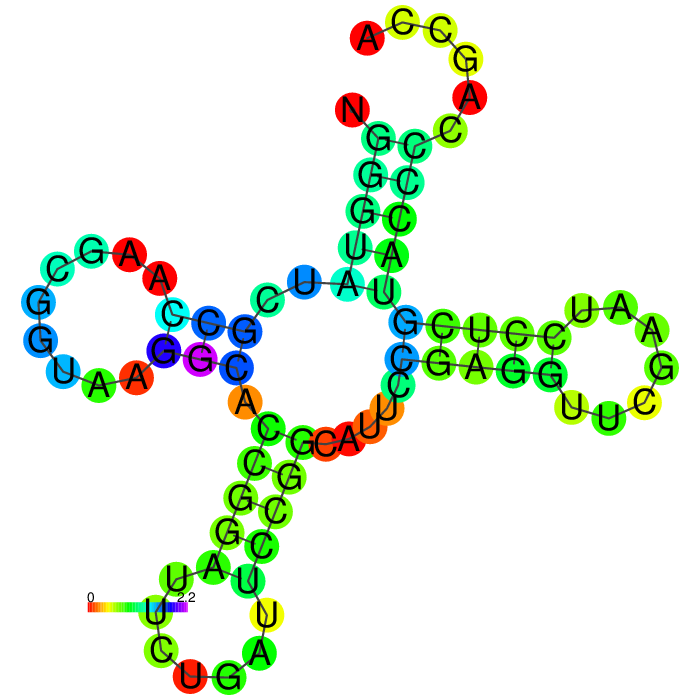
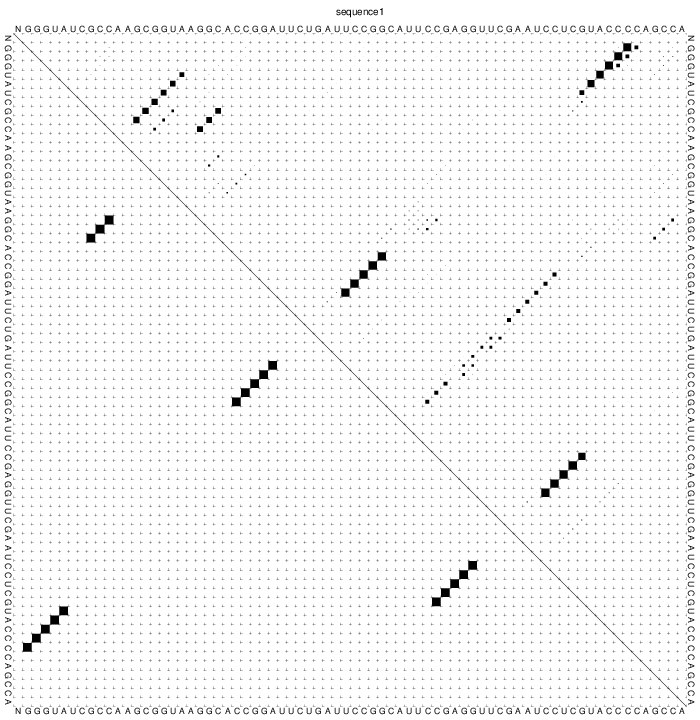
| Чтобы указать путь к RnaFold была использована команда (1). RnaFold реализует алгоритм Зукера. Команда (2) должна была предсказать структуру, но программа не сработала. Тогда был использован web вариант программы. В отчете приведены рисунки 1 и 2, изображающие minimum free energy (MFE) structure и график предсказаний связей между основаниями. Данные по парам были получены из файла 1gts_detailed.txt с детальным описанием. |
|
(1) export PATH=${PATH}:/home/preps/golovin/progs/bin
(2) cat 1gts.fasta | RNAfold --MEA > rnafold.fasta |
|---|
Рис. 1 и 2 - Minimum free energy (MFE) structure и график предсказаний связей между основаниями
 |
 |
Таблица 1. Реальная и предсказанная вторичная структура тРНК из файла 1gts.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-2-71-3' 5'-7-66-3' Всего 6 пар. |
Предсказано 0 пар. | Предсказано: с 5'-2-68-3' по 5'-6-64-3', таким образом пары не совпали. Всего 5 пар. |
| D-стебель | 5'-10-25-3' 5'-12-23-3' Всего 3 пары. |
Предсказано 0 пар. | Предсказано: с 5'-9-23-3' по 5'-11-21-3', таким образом пары не совпали. Всего 3 пары. |
| T-стебель | 5'-49-65-3' 5'-53-61-3' Всего 5 пар. |
Предсказано: с 5'-47-63-3' по 5'-53-56-3', совпало 3 пары. Всего 7 пар. | Предсказано: с 5'-47-63-3' по 5'-51-59-3', совпало 3 пары. Всего 5 пар. |
| Антикодоновый стебель | 5'-26-44-3' 5'-33-37-3' Всего 8 пар. |
Предсказано 0 пар. | Предсказано: с 5'-25-41-3' по 5'-29-37-3', пары не совпали. Всего 5 пар. |
| Общее число канонических пар нуклеотидов | Всего 20 пар (3 неканонические пары в антикодоновом стебле). | Всего 7 пар. | Всего 18 пар. |
|
Таблица сравнения показывает, что обе программы: и RnaFold, и einverted предсказали верно одни и те же пары, но все же RnaFold использовать предпочтительнее, т.к. данная программа указывает на верные участки в большинстве случаев хотя бы по 1 цепи. |
|
Для анализа ДНК-белковых контактов: 1pp8.pdb, взять цепи: M,R,Y.
В данном упражнении необходимо было создать скрипт-файл с некоторыми множествами. Set 1: множество атомов кислорода 2'-дезоксирибозы; Set 2: множество атомов кислорода в остатке фосфорной кислоты; Set 3: множество атомов азота в азотистых основаниях. |
|
Кнопка, запускающая выполнение скрипта |
После нажатия кнопки Start: изображение всей структуры. После каждого нажатия кнопки Resume переходим к следующему пункту: 1) Изображение только ДНК в проволочной модели; 2) Изображение только ДНК в проволочной модели с выделенными шариками множеством атомов set1; 3) Изображение только ДНК в проволочной модели с выделенными шариками множеством атомов set2; 4) Изображение только ДНК в проволочной модели с выделенными шариками множеством атомов set3. |
|
|
Кнопка, продолжающая выполнение скрипта Текст скрипта |
| В данном упражнении необходимо описать ДНК-белковые контакты в заданной структуре. За полярные принимались атомы кислорода и азота, неполярные - атомы углерода, фосфора и серы. За полярный контакт принималась ситуация, когда расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5 A. Неполярный контакт - пара неполярных атомов на расстоянии меньше 4.5 A. |
Таблица 2. Контакты разного типа в комплексе 1pp8.pdb
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| Остатками 2'-дезоксирибозы | 0 | 11 | 11 |
| Остатками фосфорной кислоты | 8 | 5 | 13 |
| Остатками азотистых оснований со стороны большой бороздки | 2 | 3 | 5 |
| Остатками азотистых оснований со стороны малой бороздки | 2 | 0 | 2 |
|
Ожидаемо, неполярных контактов оказалось больше, нежели полярных, так как расстояние для неполярных было задано больше, к тому же "неполярные" атомы встречаются намного чаще. Большая бороздка предполагает большую площадь контакта, поэтому контактов больше, чем со стороны малой бороздки. |
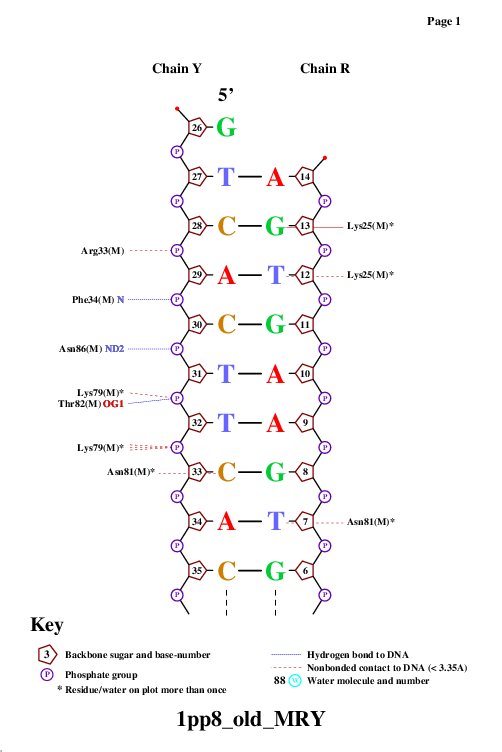
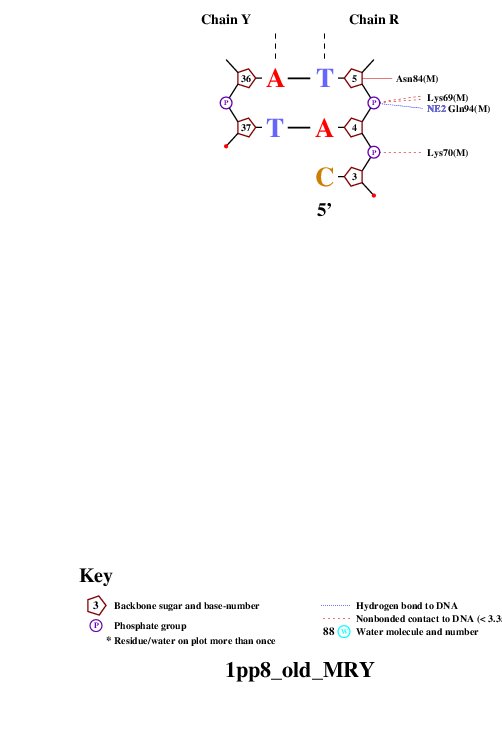
| В данном упражнении необходимо было получить популярную схему ДНК-белковых контактов с помощью программы nucplot. Изначальный файл не выдавал необходимый результат, поэтому было решено вырезать нужные цепи: M; R и Y. И с полученным файлом были произведены те же операции. В итоге была получена схема: см. рис. 3. |
Рис. 3 - Популярная схема ДНК-белковых контактов (нажать на картинку для увеличения)
 |
 |
|
Изначально, контактов на схеме изображено немного.
Однако, контактов с остовом обнаружено всего 5: Lys25 (2); Asn81 (2); Asn84 (1).
К тому же, ни один остаток не имеет более 2 контактов.
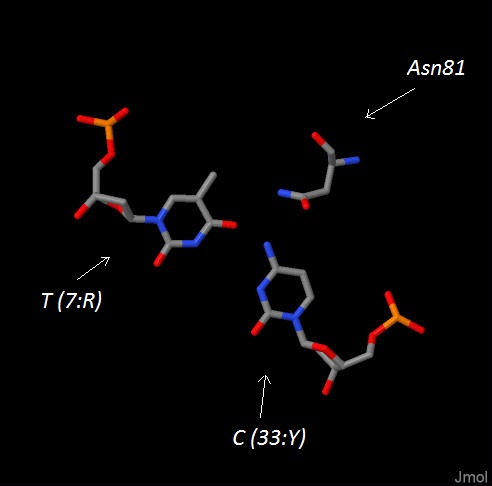
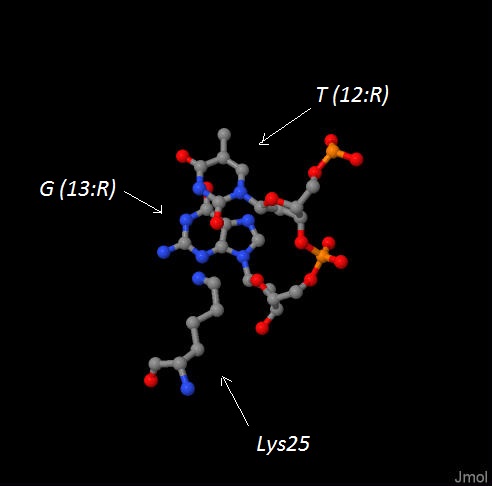
Необходимо было выбрать: 1) Аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК; 2) Аминокислотный остаток наиболее важный для распознавания последовательности ДНК. В качестве наиболее важного для распознавания последовательности необходимо было взять остаток, связанный с остовом. Таким образом, были выбраны остатки: Lys25 и Asn81. |
Рис. 4 и 5 - Asn81 и Lys25 и нуклеотиды, с которыми они взаимодействуют
 |
 |
© Kalashnikova Anastasia, 2016