Практикум 4
Задание 1: Построение дерева по нуклеотидным последовательностям
В данном задании необходимо было построить филогенетическое дерево тех же бактерий,
что и в предыдущих практикумах, используя 16S rRNA (последовательность РНК малой субъединицы рибосомы).
Ход работы:
Берем одну из последовательностей 16S rRNA из каждого файла формата frn,
полученного с помощью базы полных геномов NCBI,
переимеименовываем, указывая вид организма, и объединяем все последовательности в один fasta-файл (ссылка).
Были взяты: Bacillus anthracis (штамм Ames Ancestor),
Bacillus subtilis (штамм 168), Clostridium tetani (штамм Massachusetts / E88),
Lactobacillus acidophilus (штамм NCFM), Lactobacillus delbrueckii subsp. bulgaricus (штамм ATCC 11842),
Streptococcus pyogenes serotype M1 (штамм M1 476),
Streptococcus pneumoniae serotype 4 (штамм TIGR4).
Далее последовательности были выровнены с помощью MUSCLE на EMBL-EBI (онлайн):
(ссылка на выравнивание CLUSTAL).
Рис. 1 - Выравнивание последовательностей в раскраске Clustalx
 |
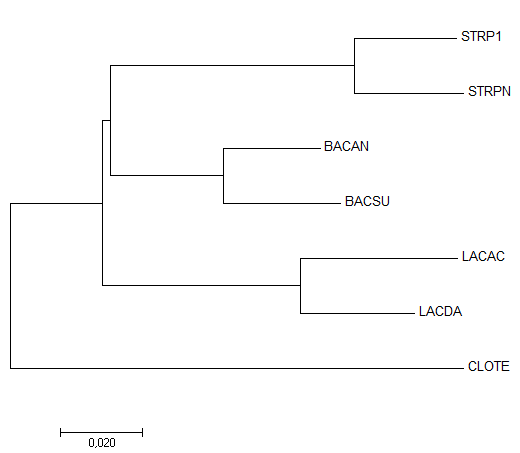
Далее программой MEGA, методом Neighbor-Joining, было построено дерево (рис. 2). 2 других метода (Minimum evolution и Maximum likelihood) дают на выход это же дерево. Данное дерево совпадает с построенным по белкам в практикуме 2 (откуда можно сделать вывод, что качество реконструкции примерно одинаковое у этих 2 методов), и, как следствие, не совпадает с правильным. Реконстрикция отличается от правильного дерева тем, что в правильном - (LACAC, LACDA) и (STRPN, STRP1) объединены в одну ветвь, а в данном - (BACAN,BACSU) и (STRPN, STRP1). То есть ветви (BACAN,BACSU) и (LACAC, LACDA) поменялись местами.
Рис. 2 - Изображение построенного дерева
 |
Задание 2: Построение и анализ дерева, содержащего паралоги
В данном задании необходимо было найти в данных бактериях достоверные гомологи белка CLPX_BACSU (обозначение на дереве).
Для нахождения гомологов использовался Blast (NCBI), несколько раз фильтруя по организму.
В качестве банка использовался "nr", E-value: 0.01. Результаты поиска предоставлены в таблице 1
(также был проведен поиск среди белков Bacillus subtilis 168:
был взят белок HslU--HslV peptidase ATPase subunit [Bacillus] (межвидовой) с id: WP_003245556.1 - ссылка (HSLU_BASCU)).
Было построено выравнивание этих гомологов в JalView (рис. 3).
Рис. 3 - Выравнивание последовательностей в раскраске Clustalx Таблица 1. Результаты Blast поиска

| Организм (запрос при поиске) | Комментарии результатов | Ссылка на файл с гомологом |
| Bacillus anthracis | Было найдено 5 результатов с E-value - 0.0, причем 4 из них имеют 100% Query cover. Были выбраны ATP-dependent protease ATP-binding subunit HslU [Bacillus anthracis] с id: AIM07675.1 (HSLU_BACAN) и ATP-dependent Clp protease ATP-binding subunit clpX [Bacillus anthracis str. H9401] с id: AFH85878.1 (CLPX_BACAN). | ссылка 1
ссылка 2 |
| Clostridium tetani E88 | Найдено 2 результа с E-value - 0.0 и с в точности совпадающими остальными параметрами. Была выбрана ATP-dependent Clp protease ATP-binding subunit ClpX [Clostridium tetani] с id: WP_035108885.1 (CLPX_CLOTE). | ссылка |
| Lactobacillus acidophilus NCFM | Обнаружены всего 2 находки. Таким образом, выбраны ATP-dependent Clp protease ATP-binding subunit ClpX [Lactobacillus acidophilus] с id: WP_003546864.1 (CLPX_LACAC) и HslU--HslV peptidase ATPase subunit [Lactobacillus acidophilus] с id: WP_011254308.1 (HSLU_LACAC). | ссылка 1
ссылка 2 |
| Lactobacillus delbrueckii subsp. bulgaricus ATCC 11842 | Найдена одна последовательность с E-value - 0.0. Всего находок 2. Эта же последовательность имеет лучшие значения по остальным показателям. Были выбраны ATP-dependent Clp protease ATP-binding subunit ClpX [Lactobacillus delbrueckii] с id: WP_003623562.1 (CLPX_LACDA) и HslU--HslV peptidase ATPase subunit [Lactobacillus delbrueckii] с id: WP_011543971.1 (HSLU_LACDA). | ссылка 1
ссылка 2 |
| Streptococcus pyogenes serotype M1 | Выдается всего 1 находадка, которая обладает E-value - 0.0 и приемлимыми значениями по остальным показателям. Была выбрана ATP-dependent Clp protease ATP-binding subunit ClpX [Streptococcus pyogenes] с id: WP_002984948.1 (CLPX_STRP1). | ссылка |
| Streptococcus pneumoniae TIGR4 | Запрос по Streptococcus pneumoniae serotype 4 не выполнялся, поэтому поиск был произведен по 2-му названию штамма, полученого с UniProt. Выдана всего 1 находка. Она обладает E-value - 0.0 и приемлимыми значениями по остальным показателям. Была выбрана ATP-dependent Clp protease ATP-binding subunit ClpX [Streptococcus pneumoniae] с id: WP_000106346.1 (CLPX_STRPN). | ссылка |
Было построено дерево этих гомологов программой MEGA, методом Neighbor-Joining. Далее необходимо было указать несколько пар паралогов и две-три группы попарно ортологичных белков, считая реконструкцию дерева - верной.
Паралоги - 2 гомологичных белка из 1 организма.
Ортологи: 1) HSLU_BASCU, HSLU_BACAN, HSLU_LACAC, HSLU_LACDA (содержат субъединицу HslU); 2) CLPX_BACAN, CLPX_BACSU, CLPX_CLOTE, CLPX_LACAC, CLPX_LACDA, CLPX_STRP1, CLPX_STRPN (содержат субъединицу ClpX).
Паралоги: CLPX_LACDA и HSLU_LACDA; CLPX_LACAC и HSLU_LACAC; HSLU_BACAN и CLPX_BACAN; CLPX_BACSU и HSLU_BASCU.
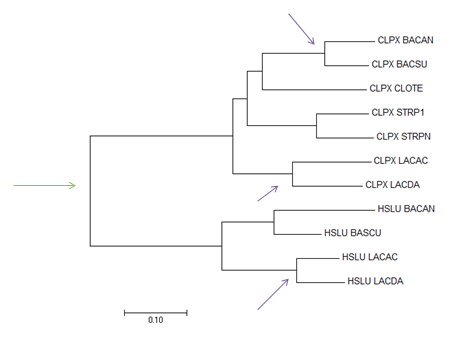
Далее необходимо было привести примеры 2 типов эволюционных событий, наблюдаемых на дереве (рис. 5): 1) Дупликация гена (пример обозначен зеленой стрелкой, он оказался всего 1); 2) Разделение путей эволюции белков в результате видообразования (3 примера обозначены фиолетовыми стрелками).
Рис. 4 - Изображение построенного дерева
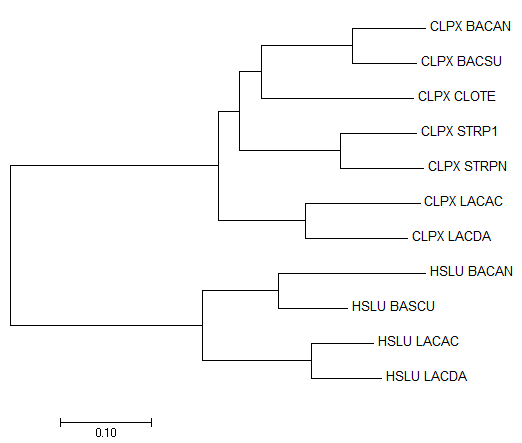 |
Рис. 5 - Изображение дерева с эволюционными событиями
 |