Электронная плотность в PyMol
Для работы была выбрана глицил-тРНК синтетаза человека (Glycyl-tRNA synthetase или GARS
(данное сокращение довольно распространено и используется в случае Homo sapiens)).
Данный фермент имеет целый ряд интересных свойств, включая неканонические.
Одним из ключевых неканонических свойств GARS является связывание с IRES элементом полиовируса [1].
Было показано, что GARS довольно прочно связывает пятый домен IRES элемента данного вируса, тем самым стимулируя его трансляцию.
Механизм данного явления на данный момент не известен.
К каноническим свойствам относят способность катализировать аминоацилирование тРНК (рис. 1) [2].
Рис. 1 - Одна из катализируемых GARS реакций [3]
| ATP + glycine + tRNA(Gly) = AMP + diphosphate + glycyl-tRNA(Gly) |
|---|
Я выбрала файл с PDB-ID: 4kr2. В таблице 1 представлена как выборочная характеристика фермента, так и краткая характеристика PDB-файла. Для данной структуры выполняет ряд необходимых для работы условий: наличие EDS map, файла структурных факторов и статьи, опубликованной по результатам расшифровки.
Конформация фермента отличается у E. coli, в которой авторы структуры и статьи производили наработку белка, от человека: в первом случае - гетеротетрамер, во втором - гомодимер. Данный факт также отмечен в литературном обзоре к статье. Конформация не влияет на большинство свойств глицил-тРНК синтетазы, как было показано в одной из статей [4], но авторы данной статьи пишут в утвердительной форме об отсутствии межвидовой активности фермента и не учитывают в экспериментальной части своей работы того, что при экспрессии в E. coli данного белка могут возникнуть проблемы с фолдингом/наработкой (и т.д.) и полученный белок может иметь конформацию/структуру, несоответствующую реальности. По вышеперечисленным причинам статья и структура вызывают подозрения, поэтому при работе с данными файлами следует проявлять осторожность.
Таблица 1. Краткая характеристика фермента/PDB-файла
| Параметр | Значение |
| Название белка на русском | Глицил-тРНК синтетаза человека |
| Название белка на английском | Glycyl-tRNA synthetase, GARS |
| Тип фермента | Лигаза |
| Теоретический вес | 72.52 кДа |
| PDB-ID | 4kr2 |
| Разрешение структуры | 3.292 Å |
| R | 0.238 |
| R_free | 0.277 |
| Конформация | Для человека: гомодимер; в файле полипептидная цепь обозначена как: Chain A |
Задание 1
В данном задании необходимо было построить изображение электронной плотности вокруг остова полипептидной цепи,
предварительно убрав боковые цепи.
На рисунке 2 изображена ленточная структрура,
на рисунках 3-5 представлены изображения электронной плотности вокруг остова при различных уровнях подрезки
(0.5; 1.5; 2.5). Для создания картинок были использованы команды:
|
(1) fetch 4kr2A
(2) isomesh 0.5, 4kr2_map, 0.5, sele, carve=2 (3) isomesh 1.5, 4kr2_map, 1.5, sele, carve=2 (4) isomesh 2.5, 4kr2_map, 2.5, sele, carve=2 |
|---|
Рис. 2 - Структура GARS (обозначен зеленым) в комплексе с тРНК (обозначена голубым)
 |
Рис. 3 и 4 - Изображения электронной плотности вокруг остова полипептидной цепи при σ = 0.5 и σ = 1.5
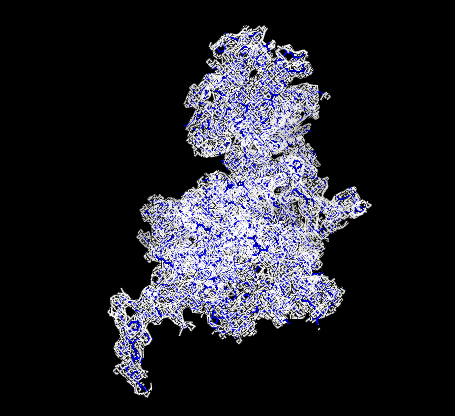
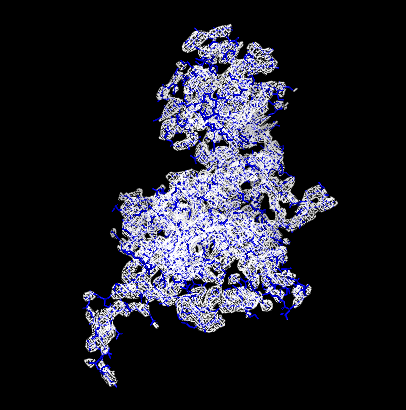 |
Рис. 5 - Изображение электронной плотности вокруг остова полипептидной цепи при σ = 2.5
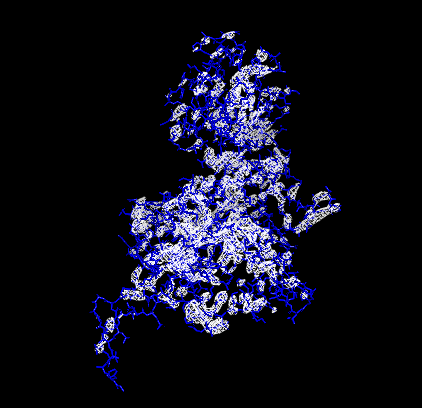 |
Задание 2
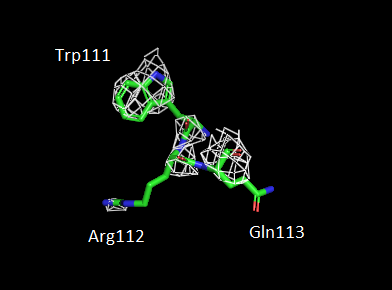
В данном задании были построены изображения электронной плотности вокруг 3 различных аминокислотных остатков при 3 уровнях подрезки ЭП:
я выбрала последовательно расположенные триптофан-111, аргинин-112 и глутамин-113.
Как и в предыдущем задании, можно заметить, что чем больше σ,
тем более "разрежена" ЭП. А также то, что остов (вторичная структура) прогнозируется (расшифровывается) лучше, чем радикалы.
Можно отметить и то, что при σ = 1.5 изображение ЭП довольно неплохо "совпадает" со структурой.
Рис. 6 и 7 - Изображения электронной плотности вокруг 3 аминокислотных остатков при σ = 0.5 и σ = 1.5
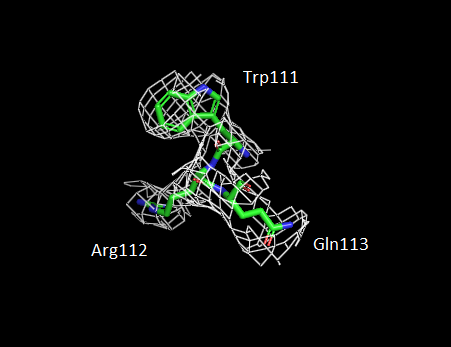
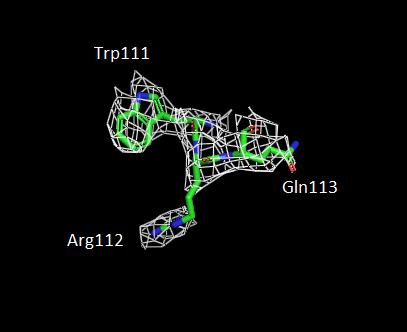 |
Рис. 8 - Изображение электронной плотности вокруг 3 аминокислотных остатков при σ = 2.5
 |
Ссылки
[1] Andreev, Dmitry & Hirnet, Juliane & Terenin, Ilya & Dmitriev, Sergey & Niepmann, Michael & Shatsky, Ivan. (2012). Glycyl-tRNA synthetase specifically binds to the poliovirus IRES to activate translation initiation. Nucleic acids research. 40. 5602-14. 10.1093/nar/gks182.
[2] http://www.xumuk.ru/encyklopedia/210.html
[3] https://www.ebi.ac.uk/pdbe/entry/pdb/4kr2
[4] Chien, C. I., Chen, Y. W., Wu, Y. H., Chang, C. Y., Wang, T. L., & Wang, C. C. (2014). Functional substitution of a eukaryotic glycyl-tRNA synthetase with an evolutionarily unrelated bacterial cognate enzyme. PloS one, 9(4), e94659. doi:10.1371/journal.pone.0094659