Поиск по PDB и содержание PDB файлов
В данном практикуме необходимо было привести примеры некоторых ситуаций.
В описаниях подпунктов приведены PDB-идентификаторы и строчки из PDB файла в подтверждение.
Occupancy
В данном задании нужно привести пример атомов с коэффициентом заполнения, не равным 1 (то есть те, у которых есть альтернативные позиции), а также altercode для них. Для этого была найдена структура (crystal structure of EphA6/Odin complex) с PDB-идентификатором: 5zry, решенная с помощью X-ray с разрешением 1.3 ангстрема. На рисунке 1 приведены строчки из данного PDB файла, где столбик "Occupancy" выделен черным.
Рис. 1 - Атомы с "Occupancy" не равным 1
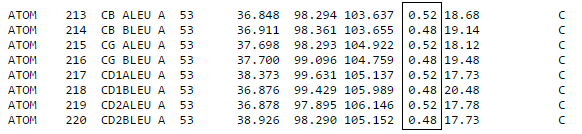 |
Missing residues
В данном задании приведены не расшифрованные аминокислотные остатки (Missing residues). Для этого я использовала тот же PDB файл, что и в предыдущем задании.
Рис. 2 - Missing residues
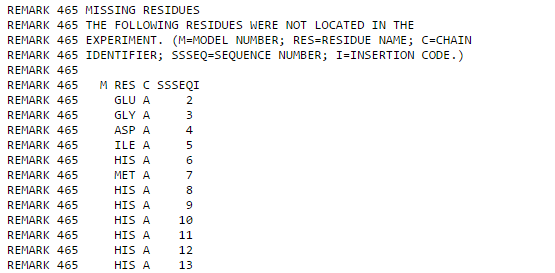 |
Последовательности
Здесь нужно было привести пример структуры белка, у которой не совпадают последовательность природного белка из Uniprot и последовательность белка, который кристаллизовали. Была выбрана структура (human ERp44 zinc-bound form) с PDB-идентификатором: 5xwm с помощью advanced search (Sequence Features - Wild Type protein). На рисунке 3 представлены некоторые SEQADV строчки, которые показывают различия между DBREF (кросс-референс данной последовательности и последовательностей из других БД) и данной последовательностью. Можно отметить, что в данном случае последовательности отличаются наличием экспрессионного тэга.
Рис. 3 - Различия в последовательностях
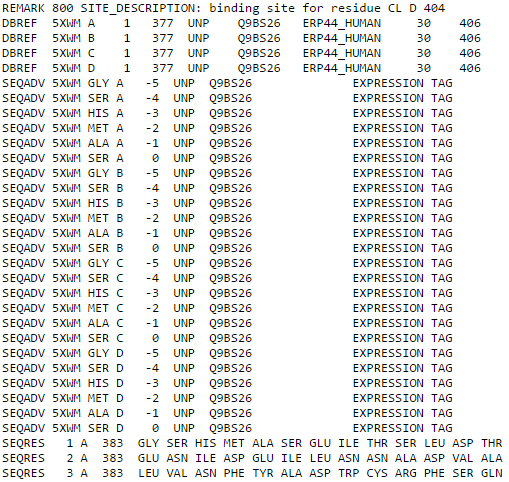 |
Худший и лучший B-фактор в структуре
Для выполнения данного задания я использовала ту же структуру, что и в предыдущем задании.
Худший и лучший B-фактор выбирала на глаз: на рисунке 4 представлены строчки с соответствующими атомами и B-факторами.
Худший B-фактор: 206.21; лучший B-фактор: 20.40.
Рис. 4 - Худший и лучший B-факторы
 |