В данном практикуме необходимо было поработать с данными проекта по секвенированию бактерии Buchnera aphidicola. Мне достался код доступа SRR4240387
[1]. Был скачан заархивированный файл формата fastq, который был перенесен в
папку /nfs/srv/databases/ngs/atitova/pr14 и там же распакован с помощью команды gunzip:
gunzip SRR4240387.fastq.gz.
Buchnera aphidicola - вид гамма-протеобактерий, являющихся первичными эндосимбионтами гороховых тлей Acyrthosiphon pisum.
 |
| Рис. 1. Buchnera aphidicola в клетке хозяина
[2] |
Гороховая тля (лат. Acyrthosiphon pisum) — вид семейства настоящих тлей (Aphididae).
 |
| Рис. 2. Acyrthosiphon pisum[3]
|
Очистка чтений
Перед началом работы адаптеры из директории /P/y16/term3/block3/adapters были собраны в один файл adapters.fasta.
Команда, удаляющая остатки адаптеров:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240387.fastq SRR4240387_no_ad.fastq ILLUMINACLIP:adapters.fasta:2:7:7
После работы команды:
Input Reads: 15032810 Surviving: 15029657 (99,98%) Dropped: 3153 (0,02%) (т.е. из 15032810 входных чтений были отброшены 3153 чтений).
Команда, обрезающая с концов чтений нуклеотидов с качеством ниже 20 и отбирающая чтения длины не менее 30:
java -jar /usr/share/java/trimmomatic.jar SE -phred33 SRR4240387_no_ad.fastq SRR4240387_trim.fastq TRAILING:20 MINLEN:30
После работы команды имеем:
Input Reads: 15029657 Surviving: 4437416 (29,52%) Dropped: 10592241 (70,48%) (т.е. из 15029657 входных чтений были отброшены 10592241 чтений).
Подготовка k-меров
Подготовка k-меров была произведена с помощью программы velveth, которая предназначена для создания набора данных, которые далее могут
обрабатываться программой velvetg. В нашем случае было необходимо подготовить k-меры длины 29 для коротких непарных чтений (-short) из
файла в формате fastq (-fastq). Выходные файлы по умолчанию записывались в папку velveth.
velveth velveth 29 -fastq -short SRR4240387_trim.fastq.
Cборка на основе k-меров
Далее для обработки данных из предыдущей части была использована программа velvetg, которая строит граф де Брайна (граф ориентированный,
в его вершинах расположены последовательности символов длины n. Граф отражает пересечения между последовательностями символов).
Команда
velvetg velveth
В результате работы имеется граф с 1164 вершинами, N50 = 1367. Таблицу, содержащую информацию о самых
длинных контигах вы можете видеть ниже.
| Таблица 1. Самые длинные контиги. |
| ID |
Длина |
Покрытие |
| 78 |
7399 |
33.421679 |
| 26 |
6230 |
25.778652 |
| 21 |
5261 |
23.969397 |
Для поиска контигов с аномально большим или аномально малым покрытием (более чем в 5 раз отличающимся от "типичного") с помощью средств Excel была
определена медиана, которая оказалась равной 14.809562, а также среднее занчение = 527.0518744. (
Ссылка на таблицу.)
Это говорит о большом разбросе значений. Ниже вы
можете видеть таблицу, содержащую информацию о некоторых контигах с аномальными покрытиями (в скобках указано во сколько раз значение покрытия
больше или меньше среднего значения).
| Таблица 2. Контиги с аномальным покрытием. |
| ID |
Длина (k-мер) |
Покрытие |
| 979 |
1 |
565714 (больше в ~1074 раз) |
| 1046 |
1 |
8392 (больше в ~16 раз) |
| -(~15 контигов) |
1 - 5 |
1 (меньше в ~527 раз) |
Анализ
С помощью программы megablast каждый из трёх самых длинных контигов был сравнен с уже имеющейся хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
| Таблица 2. Сравнение контигов с наибольшей длиной и хромосомы Buchnera aphidicola. |
| ID |
Координаты |
Max score |
Total score |
Query cover (%) |
E-value |
Identities |
Alignment length |
Gaps |
| 78 |
35162:42578 |
6935 |
6935 |
1 |
0.0 |
6227/7446(84%) |
7427 |
68/7446(0%) |
| 26 |
536553:542799 |
4577 |
4577 |
0 |
0.0 |
5037/6288(80%) |
6258 |
113/6288(1%) |
| 21 |
584329:587054 |
1277 |
1277 |
0 |
0.0 |
2100/2777(76%) |
5289 |
110/2777(3%) |
Ниже вы можете видеть карты локального сходства для соответствующих контигов. (Ось X - бактериальный геном, Y - контиг).
 |
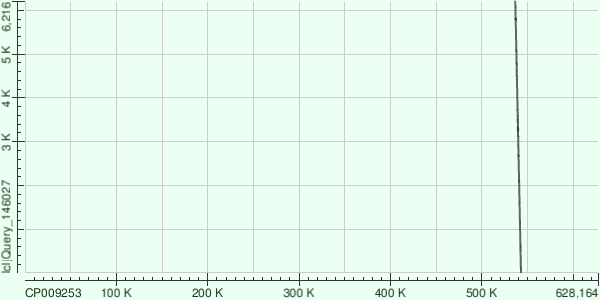
| Рис. 3. NODE_78 |
Как видно из карты локального сходства, контиг 78 инвертирован относительно бактериального контига. Выравнивание было одно. Вся находка расположилась
без разрывов на карте, что говорит о достоверности этой находки.
 |
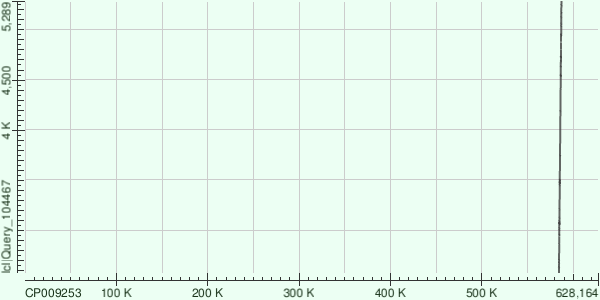
| Рис. 4. NODE_26 |
Ситуация аналогична предыдущей: последовательность контига 26 инвертирована, расположена без разрывов.
 |
| Рис. 5. NODE_21 |
В данном случае инверсии не наблюдается. Находка вновь расположена без разрывов.
Источники:
[1]: ENA (European Nucliotide Archive). Run: SRR4240387
[2]: Wikipedia. Buchnera (bacterium)
[3]: Wikipedia. Acyrthosiphon pisum



