Чтение последовательности ДНК на основании данных, полученных из капиллярного секвенатора по Сэнгеру. Отчёт о проблемах
при чтении хроматограмм
Капиллярный секвенатор выдает файлы с хроматограммой и автоматически прочтённой последовательностью в формате .ab1.
Нам дано два файла в формате .ab1, соответствующие прочтению прямой и обратной цепочки секвенируемой ДНК.
(Ссылки на исходные файлы в .ab1 формате: прямая и обратная цепи. )
Для просмотра хроматограмм и редактирования автоматического прочтения использовалась программа Chromas (Lite).
Некоторые термины:
Проблемный нуклеотид — тот, для которого было принято решение, отличное от предложенного программой, или мы согласились с программой, но
необходимо было проанализировать хроматограммы и принять решение.
Проблемные нуклеотиды в последовательности выделены строчными буквами.
Полиморфизм — нуклеотид, про который было решено, что в секвенируемой ДНК встречаются два (или более) варианта.
Полиморфизмы обозначены кодами вырожденных нуклеотидов.
Ссылка на исправленную версию прямой цепи в FASTA - формате.
Ссылка на исправленную версию обратной цепи в FASTA - формате.
JalView-проект с выравниванием прямого и обратного прочтений (тех частей, которые пригодны); выделены проблемные нуклеотиды и
полиморфизмы.
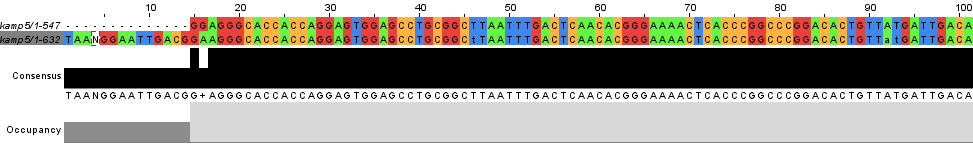 |
| Рис. 1. Фрагмент выравнивания прямой цепи и цепи комплементарной обратной, полученное с помощью needle. |
Обоснование решений для проблемных нуклеотидов или полиморфизмов
| Таблица 1. Проблемные участки в хроматограммах |
| Изображение | Обоснование
решения проблемы |
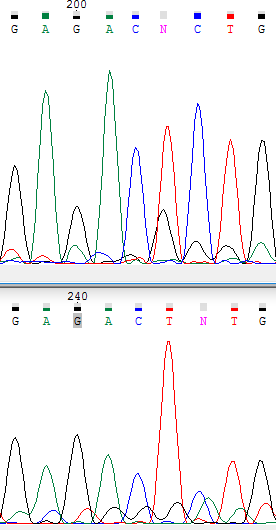 |
Высокий уровень шума не позволяет программе автоматически определить нуклеотид, однако в цепи комплементарной обратной в данной
позиции очевидно находится тимин. Следовательно на позиции 203 - тимин. |
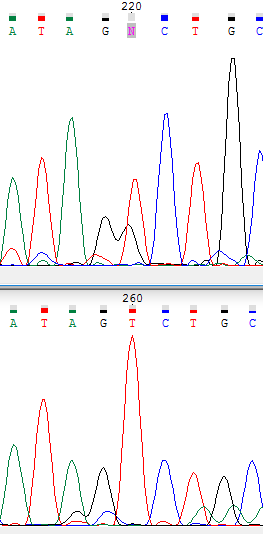 |
Размывание пиков, а также высокий шум не позволяет автоматически определить нуклеотид, но в цепи комплементарной обратной в этой
позиции находится ярко выраженный пик тимина. Поэтому считаем, что в прямой цепи на 220 позиции также находится тимин. |
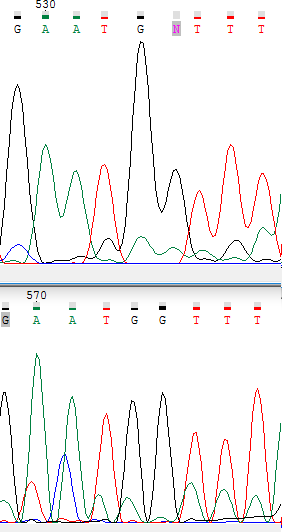 |
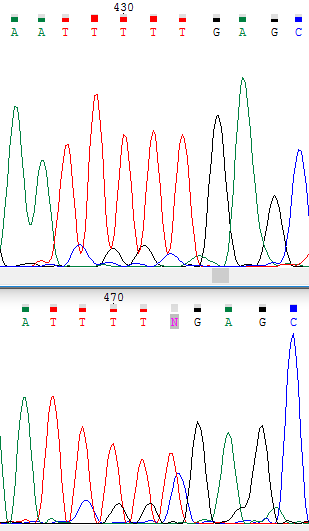
Сильное размывание пиков затруднило автоматическое определение нуклеотида, однако в цепи комплементарной обратной на этом месте
пики не размыты, а на данной позиции находится гуанин, следовательно считаем, что на 534 позиции - гуанин. |
 |
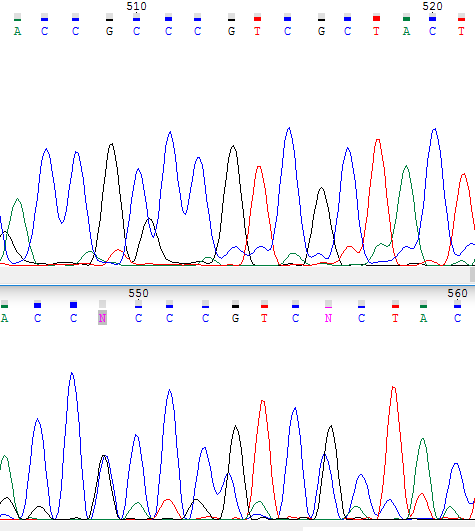
Высокий уровень шума не позволяет программе автоматически определить нуклеотид в цепи комплементарной обратной, но в прямой цепи в
данной позиции пики очевидно разделены, что позволяет нам заключить о том, что на 472 позиции находится тимин. |
 |
В данном случае в цепи комплементарной обратной в позиции 549 очевидно наблюдается полиморфизм. Поэтому в данном случае в соответсвии с
Nucleotide ambiguity code[1] в исправленной версии на этом месте
стоит S (strong). |
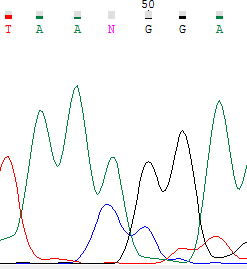 |
В позиции 49 в цепи комплементарной обратной произошло сильное размывание пиков и наложение на них шума, что не позволило
программе автоматически определить, какой нуклеотид находится здесь. Решение данной проблемы затрудняется тем фактом, что в прямой цепи отсутствует
такой участок. Поэтому на данную позицию было решено поставить M (amino) в соответствии с Nucleotide ambiguity code[1] |
Характеристика хроматограммы
| Таблица 2. Нечитаемые участки хроматограммы |
| 5' | 3' |
| Прямая цепь | 1 - 19 (19 нуклеотидов) | 567 - 693 ?(126 нуклеотидов) |
| Обратная (комплементарная) цепь | 1 - 45 (45 нуклеотидов) | 678 - 697 (20 нуклеотидов) |
Общая характеристика хроматограммы прямой цепи:
- мощность сигнала в среднем составляет 1000, мощность шума — 150;
- сигнал и шум в среднем соотносятся как 1:10;
- сигнал распределен вдоль читаемого участка последовательности равномерно (но иногда пурин дает более высокий сигнал);
- распределение шума тоже равномерно.
Общая характеристика хроматограммы цепи комплементарной обратной:
- мощность сигнала в среднем составляет 1000, мощность шума — 100;
- отношение сигнала к шуму составляет около 1:8;
- сигнал распределен вдоль последовательности равномерно;
- распределение шума не совсем равномерно: в первой и во второй половине он на одном уровне с сигналом.
В итоге, можно сказать, что качество первой хроматограммы значительно лучше. Там оказалось значительно меньше проблемных участков.
Пример нечитаемого фрагмента хроматограммы
На рисунке ниже вы можете видеть фрагмент очень плохой хроматограммы: уровень шума очень высокий, не возможно разделить пики,
практически во всех позициях присутствуют сразу
несколько пиков примерно одикаковых по высоте, что говорит о сильной загрязненности образца. Возможно, в препарате считалось сразу несколько различных
ДНК. Также можно наблюдать широкие размытые пики (заключены в зеленые круги), которые могут быть пятнами краски.
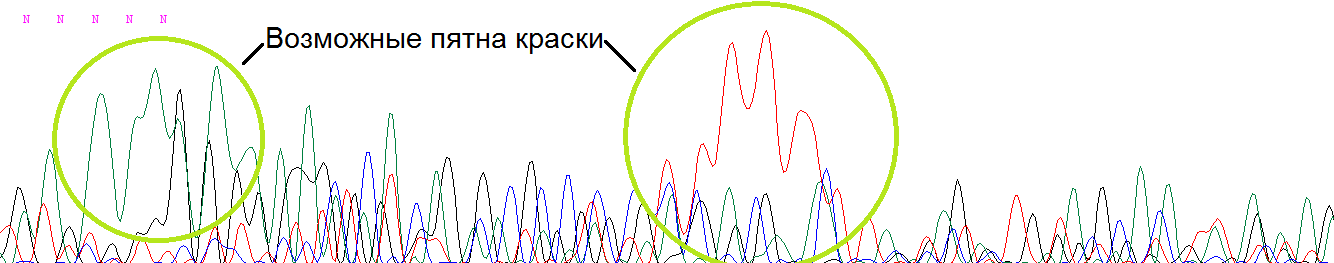 |
| Рис. 2. Пример нечитаемого фрагмента хроматограммы. |
Источники:
[1]: Nucleotide ambiguity code
|
Titova Anastasiya, 2017 ©
|



