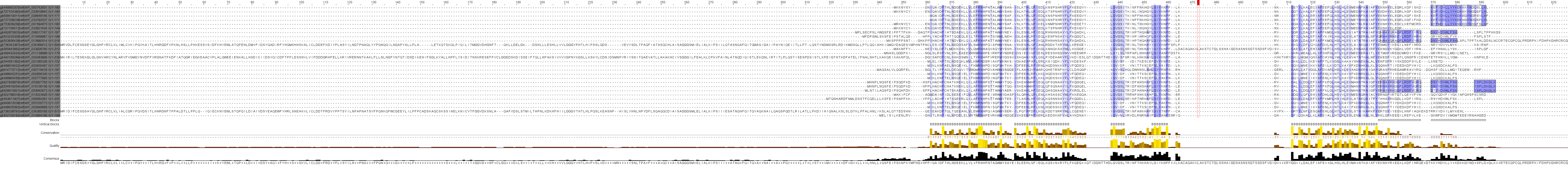
Был проведен поиск с ограничением в 1000 результатов и пределом по E-value 0.02. Параметры поиска можно скачать здесь. Было найдено 575 последовательностей. Среди них много высокогомологичных с Identical > 95%. Я считаю критерий гомологии E-value < 0.01 и Cover > 70% хорошим и подходящим в данном случае.
В таблице приведены параметры для лучшей, трех средних и худшей находок.
| Организм | Длинна выравнивани | Bit Score | E-Value | Идентичные остатки | Сходные остатки | |
|---|---|---|---|---|---|---|
| 1 | Helicobacter pylori | 180 | 925 | 2e-126 | 100% | 100% |
| 2 | Helicobacter pylori | 180 | 865 | 2e-117 | 94% | 97% |
| 3 | Helicobacter pylori | 171 | 836 | 5e-113 | 95% | 97% |
| 4 | Pseudomonas syringae | 161 | 117 | 9e-05 | 27% | 46% |
| 5 | Desulfitobacterium dehalogenans | 186 | 101 | 0.018 | 26% | 44% |
В этом выравнивании ясно видны обширные негомологичные участки на C- и -N
концах. Также было выявлено несколько вертикальных блоков (помечены В) множество
блоков из части последовательностей выделены символом H.

Участки отличающиеся в разных выравниваниях 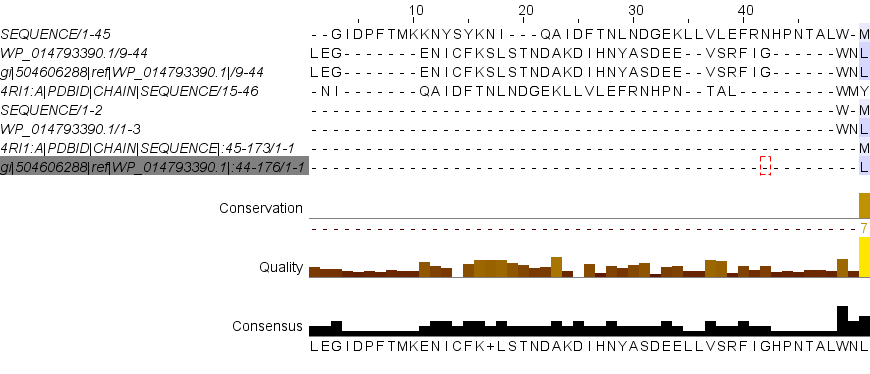
Первые две последовательности выровняны needle. Вторые две water.
