Практикум 11
Предсказание генов прокариот
Была выдана плазмида CP014303 длинной 89,596 bp содержит 4578 генов из которых 4377 белок-кодирующие. Организм происхождения: Hymenobacter sp. PAMC26628.
С помощью программы seqret были получены файлы plasmid.fasta и plasmid.gff коммандами:
seqret embl:CP014303 plasmid.fasta
seqret embl:CP014303 -feature plasmid.gff
Далее при помощи программы Prodigal с параметрами:
-i (указывает специфичный входной файл формата FASTA), -o (указывает специфичный выходной файл; по умолцанию stdout) и -f (выбор формата вывода: gbk, gff или sco; по умолчанию gbk) взят sco, как самый минималистический,
был получен файл plasmid.pro. Команда:
prodigal.windows.exe -i plasmid.fasta -o plasmid.pro -f sco
Сравнение файлов
Для сравнения предсказанных генов с известными был написан скрипт на Python принимающий в качестве аргументов имена файлов plasmid.pro и plasmid.gff.
Алгоритм работы скрипта: Программа в начале переводит оба файла в вид массива из пар чисел: N- и C- концов соответственно, при этом концы обратных генов переворачиваются. Затем идет сравнение с основой - известными парыми чисел. Им ищется соответстви в предсказании, при нахождении полного или частичного совпадения концов соответствующие счетчики увеличиваются. Вывод: число предсказанных и проценты.
| Предсказание генов в базе данных и программой Prodigal |
| Число генов, совпадающих обоими концами |
58 |
| Процент генов, совпадающих обоими концами |
75% |
| Число генов с неверным N-концом |
14 |
| Процент генов с неверным N-концом |
18% |
| Число генов с неверным C-концом |
0 |
| Процент генов с неверным C-концом |
0% |
| Число несовпавших генов |
5 |
| Процент несовпавших генов |
6% |
Причины ошибок
Рассмотрим к примеру гены:
9902-8865 Ошибка по N- Prodigal определил лишние нуклеотиды из-за более распространенного старт кодона ATG вместо TTG
Предсказание Prodigal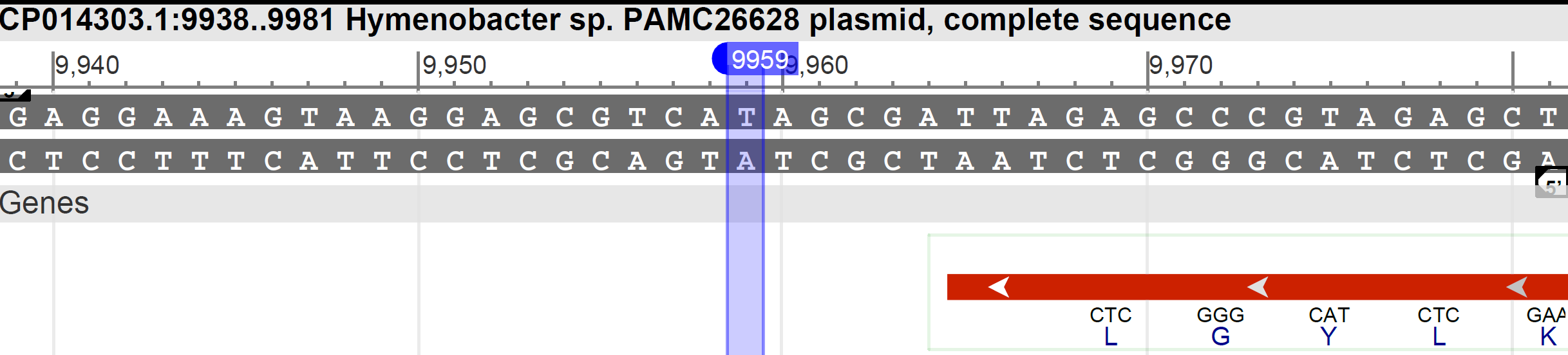 Из GenBank
Из GenBank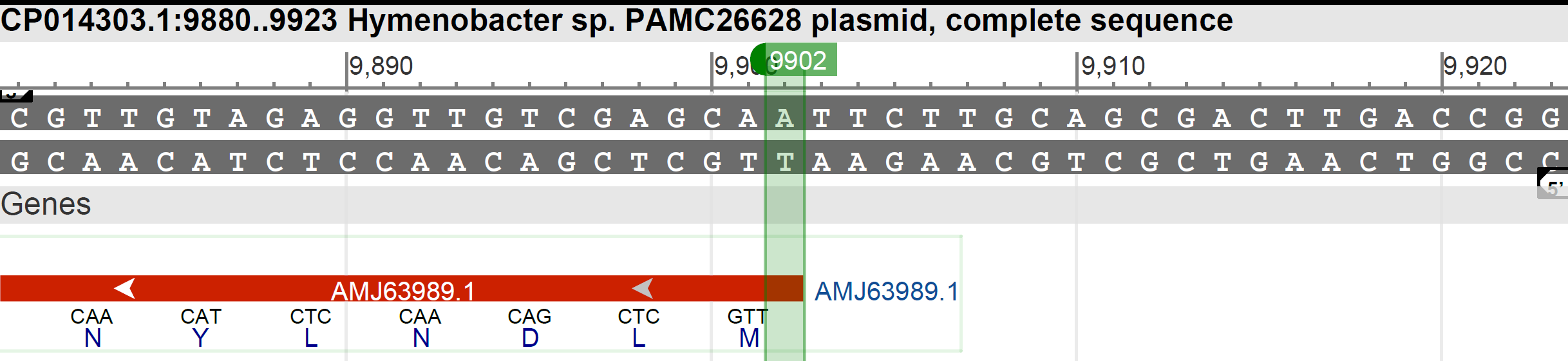 21846-24209 Ошибка по N- Prodigal определил лишние нуклеотиды поскольку нашел еще один старт кодон ATG ранее по последовательности.
21846-24209 Ошибка по N- Prodigal определил лишние нуклеотиды поскольку нашел еще один старт кодон ATG ранее по последовательности.
Предсказание Prodigal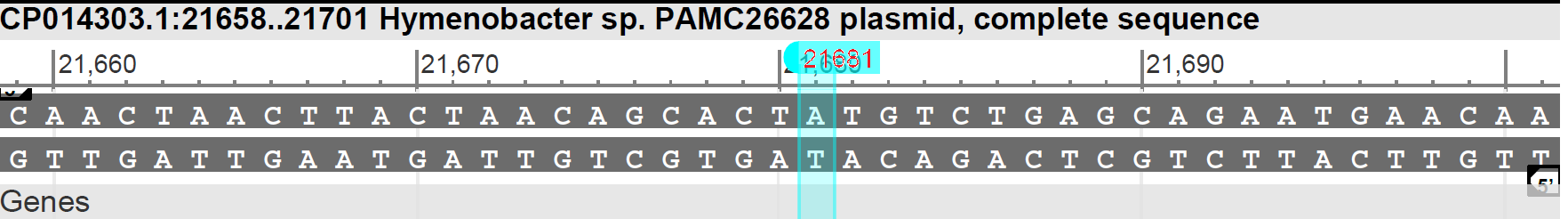 Из GenBank
Из GenBank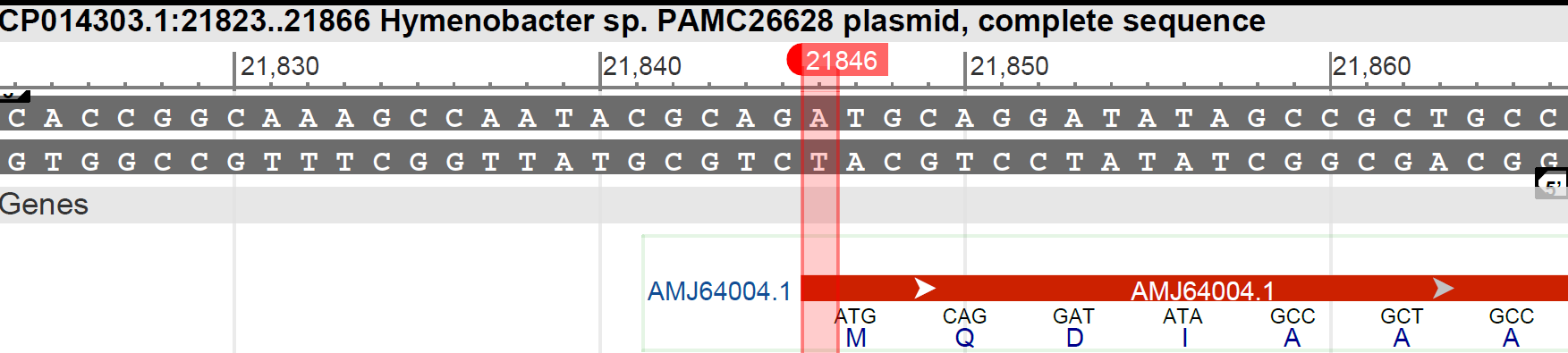 Term 3
Main page
Term 3
Main page
© Artemiy Polozhintsev (Артемий Положинцев) 2016
 Из GenBank
Из GenBank 21846-24209 Ошибка по N- Prodigal определил лишние нуклеотиды поскольку нашел еще один старт кодон ATG ранее по последовательности.
21846-24209 Ошибка по N- Prodigal определил лишние нуклеотиды поскольку нашел еще один старт кодон ATG ранее по последовательности. Из GenBank
Из GenBank Term 3
Main page
Term 3
Main page