Была взята последовательность. Проведен Blastn. Лучшая находка со схожестью 99% и покрытием 84% соответcвует гену нистона H3 организма Brada inhabilis
/protein_id="AHZ58436.1" /translation="TGGKAPRKQLATKAARKSAPATGGVKKPHRYRPGTVALREIRRY QKSTELLIRKLPFQRLVREIAQDFKTDLRFQSSAVMALQEASEAYLVGLFEDTNLCAI HAKRVTIMPKDMS"
Поскольку для следуйщих находок совпадение падает до 91%, можно считать определенность таксона односзначной.
Сравним выдачи алгоритмов blastn, megablast и discontiguous megablast для той же последовательности, но с ограничением по таксонам: поиск только внутри семейства Flabelligeridae, исключая род Brada.
Основные параметры blast:
Database: Others (nr etc.) Optimize for: как раз 3 варианта, 3 алгоритма Max target sequences: 1000 Expect threshold: 10 Word size: 11 (для blastn) Max matches in a query range: 0 Match/Mismatch Scores: 2,-3 Gap Costs: Existence: 5 Extension:2 Filter; Low complexiy regions Mask: Mask for lookup table only
Кроме того, запрос discontiguous megablast имеет два дополнительных параметра: Template length:18 и Window size:40.
Megablast
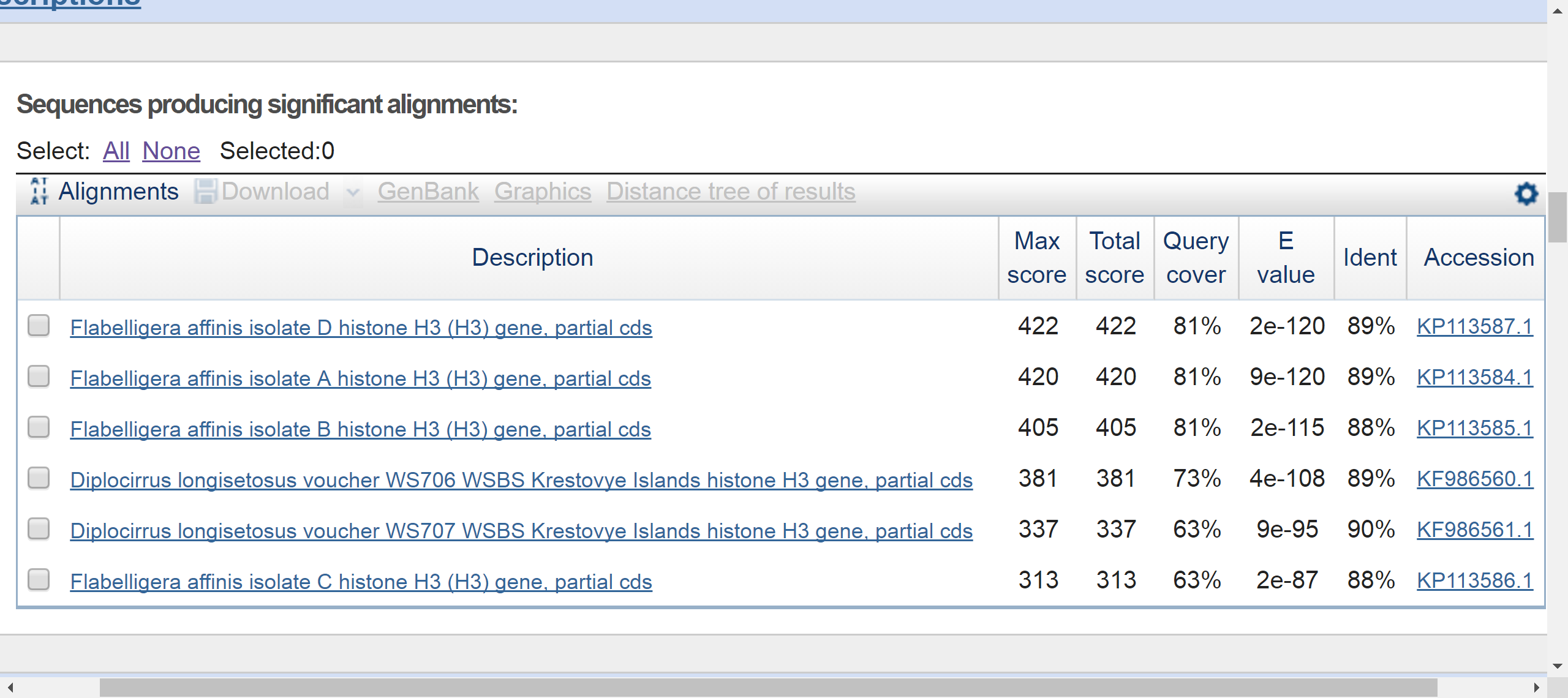
Discontiguous megablast
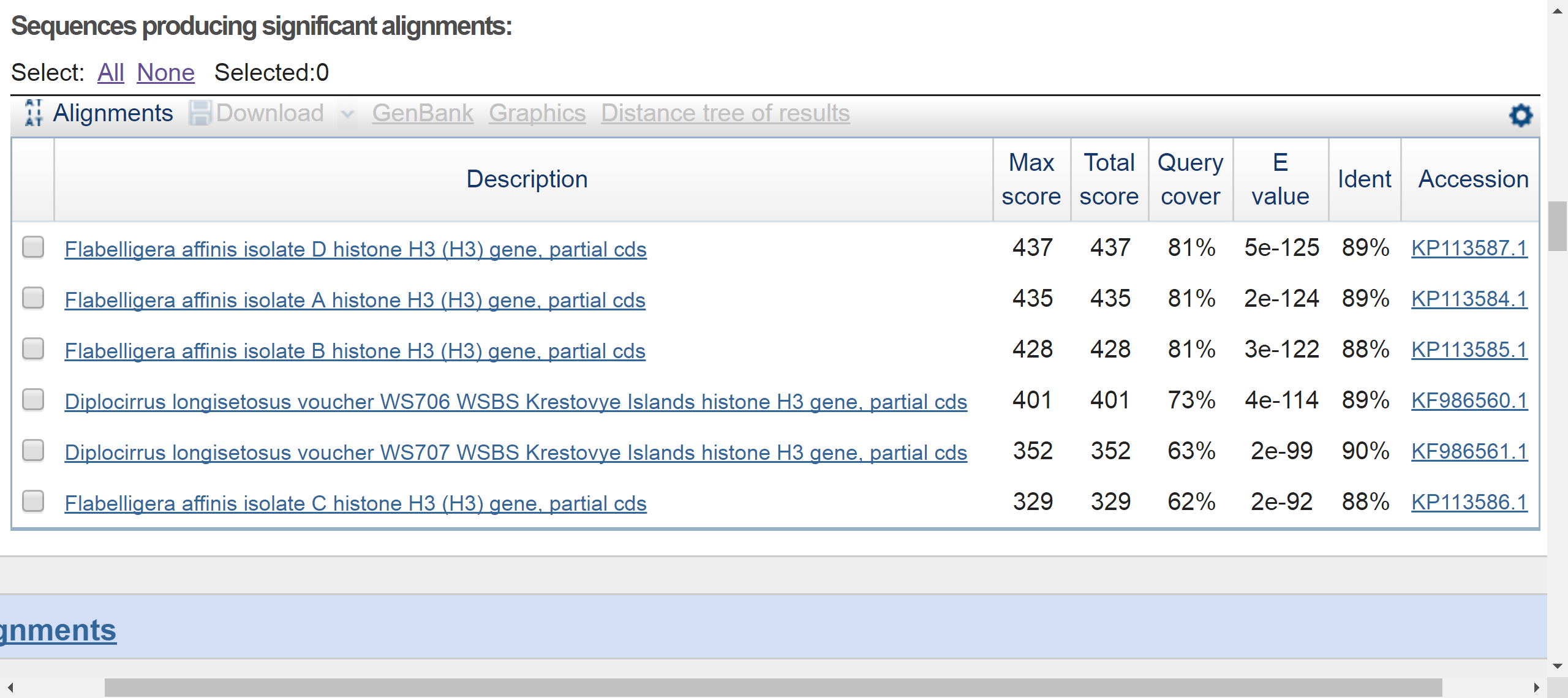
Blastn
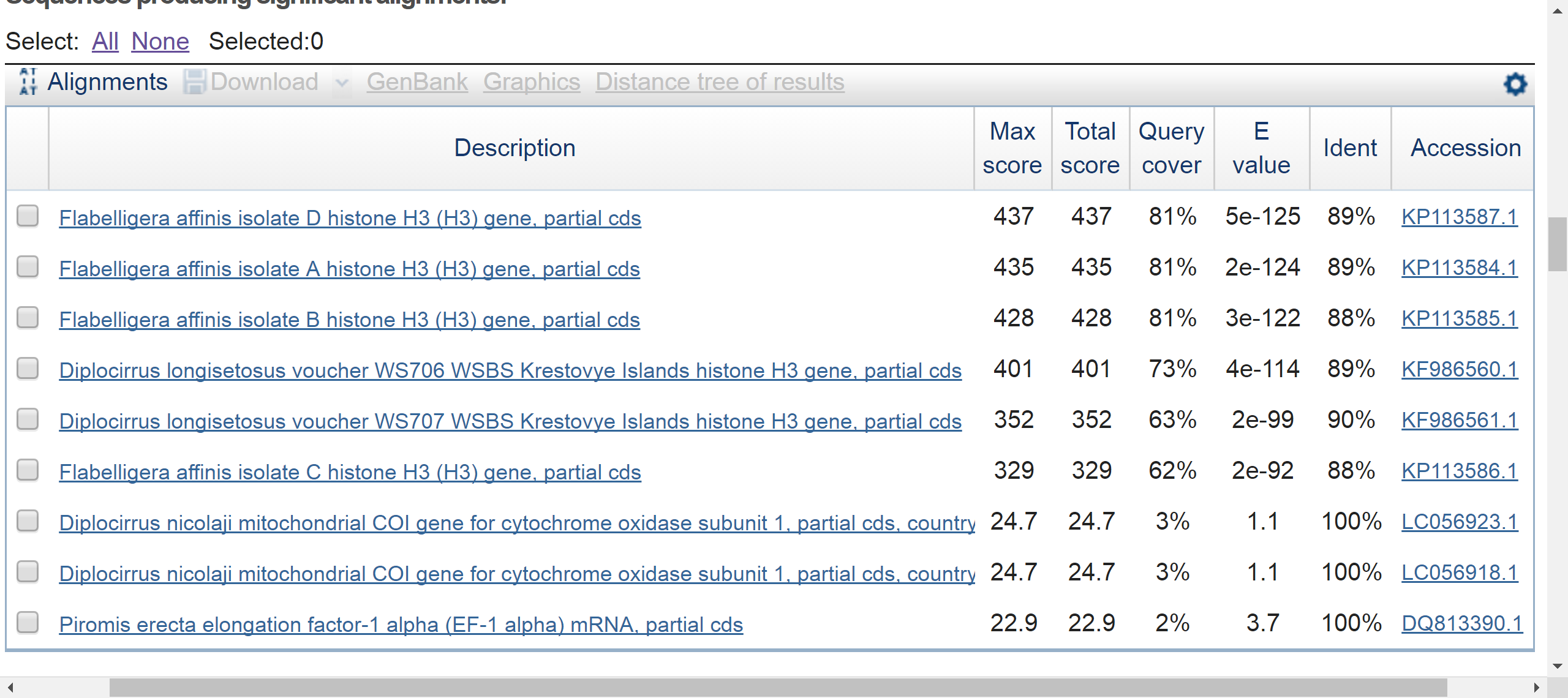
У некоторых организмов в выдаче Blastn очень низкое покрытие (2-3%) и очень высокий E-value, это их и отличает от остальных находок. Это демонстрирует, что алгоритм blastn с той же длиной слова, что discontiguous blast, однако, в отличие от него, blastn представляет даже достаточно далекие последовательности, т.е. все "гомологи". Т.е., в отличие от discontiguous blast и megablast, blastn не отсекает находки с низким Query covery и E-value.
Разница между двумя другими алгоритмами небольшая: у discontiguous megablast минимальный E-value несколько выше, навярняка это связано с тем что длина слова у него 11 в отличии от 28 у megablast, который лучше подходит для поиска среди высоко сходных последовательностей.
Проверим гомологию следуйщих белков: CISY_HUMAN, RPB1_HUMAN, UBB_HUMAN
CISY_HUMAN. Есть находки с отличным E-value, можно сделать вывод, что гомолог присутствует.
Score E Sequences producing significant alignments: (Bits) Value scaffold-693 565 2e-180 scaffold-157 564 5e-180 scaffold-287 100 3e-22 scaffold-212 92.4 1e-19
RPB1_HUMAN. Есть находки даже с нулевым E-value, что говорит о гомологии.
Score E Sequences producing significant alignments: (Bits) Value scaffold-300 1495 0.0 scaffold-157 1390 0.0 scaffold-44 197 2e-50 scaffold-100 194 3e-49 unplaced-712 132 6e-35
UBB_HUMAN. Есть находки с отличным E-value, можно сделать вывод, что гомолог присутствует.
Score E Sequences producing significant alignments: (Bits) Value scaffold-105 442 2e-141 scaffold-17 420 5e-134 scaffold-378 144 1e-43 scaffold-693 150 3e-40 scaffold-157 149 3e-40 scaffold-451 132 2e-34
Применим blastx для поиска гена белка в scaffold-456 сборки, использованной в предыдущем задании. В этом скэффолде 11344 п.н., поэтому в нем возможно наличие гена.
Потребовавшиеся команды:
infoseq <имя файла> -only -name -length seqret <имя файла>:<имя последовательности> -out <имя выходного файла>
Были изменены параметры алгоритма: с ограничением по таксону: Opisthokonta (taxid:33154), максимальным числом находок: 50 и порогом ожидания: 0.001. Остальные параметры использовались по умолчанию. В результате были получены следующие находки:
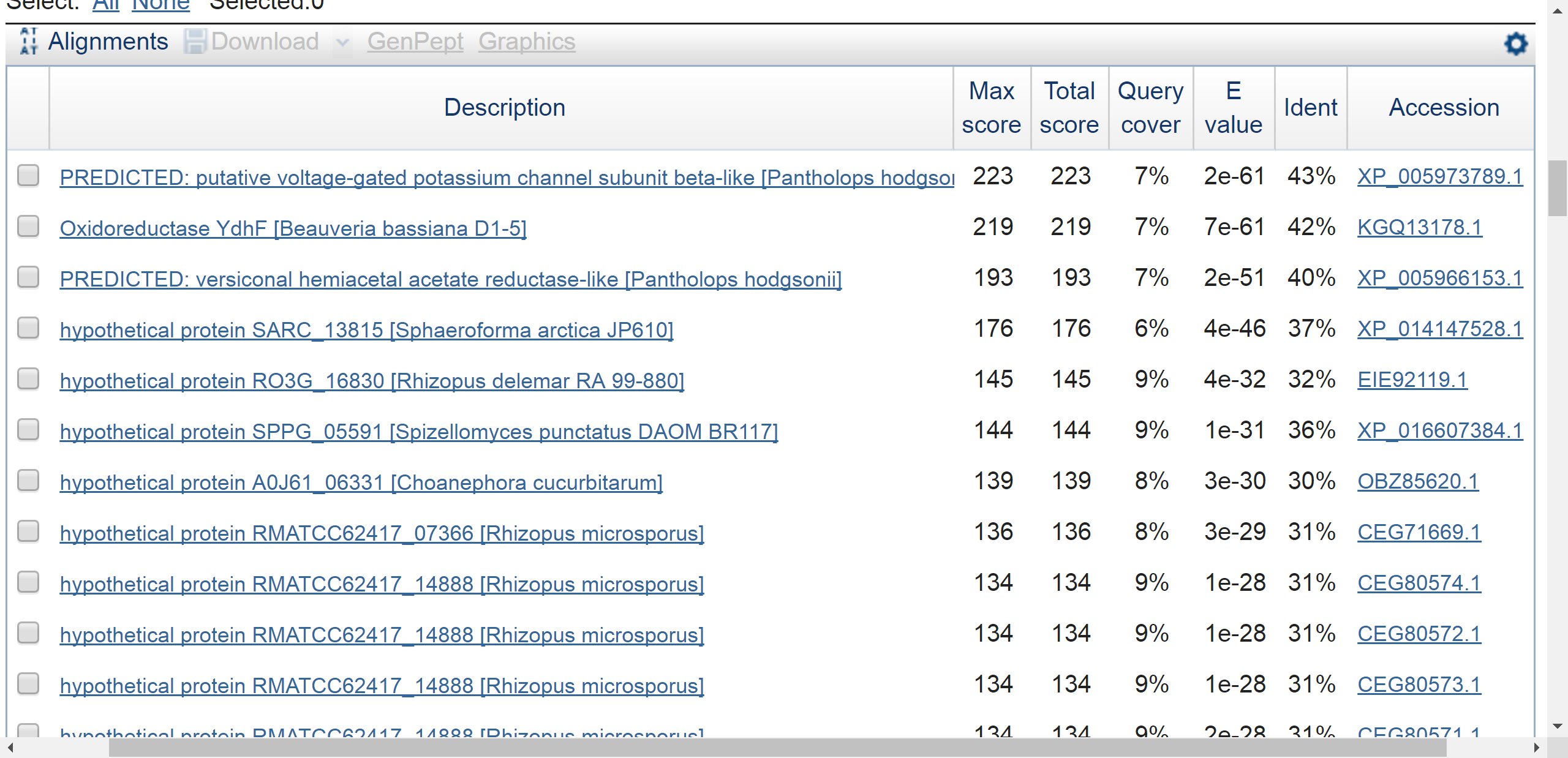
Лучшая находка с E-value 2e-61 принадлежит бета-субъединице контролируемых напряжением калиевых каналов организма Pantholops hodgsonii XP_005973789.1 Можно уверенно говорить о наличии гомолога этого белка в скэффолде.
Term 3 Main page