| A-форма | B-форма |
|---|---|
 |
 |
| Атомы тимина в трех формах ДНК | |||
|---|---|---|---|
| A-форма | B-форма | Z-форма | |
| В сторону большой бороздки обращены атомы: | [DT]31:B.N1 [DT]31:B.C2 [DT]31:B.O2 |
[DT]31:B.C4 [DT]31:B.O4 [DT]31:B.C5 [DT]31:B.C7 [DT]31:B.C6 |
- |
| В сторону малой бороздки обращены атомы: | [DT]31:B.C5 [DT]31:B.C4 [DT]31:B.C7 [DT]31:B.O4 |
[DT]31:B.N1 [DT]31:B.C2 [DT]31:B.O2 [DT]31:B.N3 |
- |
| Остальные атомы основания: | [DT]31:B.C6 [DT]31:B.N3 |
- | - |
C помощью Jmol были измерены основные параметры спирали для каждой из форм:
| A-форма | B-форма | Z-форма | |
|---|---|---|---|
| Тип спирали | правая | правая | левая |
| Шаг спирали (Å) | 28.03 | 33.75 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16.81 Å [DT]7:A.P #126,[DC]36:B.P #720 |
17.21 Å [DC]28:B.P #556,[DA]10:A.P #187 |
16.08 Å [DC]58:B.P #1171,[DC]22:A.P #433 |
| Ширина малой бороздки (Å) | 7.98 Å [DA]26:B.P #515,[DG]9:A.P #165 |
11.69 Å [DT]31:B.P #618,[DA]14:A.P #269 |
7.2 Å [DG]21:A.P #411,[DG]63:B.P #1272 |
Для измерения углов был взят нуклеотид [DT]31:B.
| Торсионные углы разных форм ДНК в градусах | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Далее сравнивал торсионные углы в структурах А-, В- и Z-форм ДНК с помощью пакета 3DNA. Так же сделан анализ для выданных тРНК и ДНК-белкового комплекса.Файл с расчетами
| Средние значения торсионных углов в градусах | |||||||
|---|---|---|---|---|---|---|---|
| α | β | γ | δ | ε | ζ | χ | |
| A-форма | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| B-форма | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
| Z-форма (гуанин) | 51.9 | 179.0 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
| Z-форма (цитозин) | -139.5 | -136.8 | 50.9 | 137.6 | -96.5 | 82.0 | -154.3 |
| тРНК (PDB ID 1gts) | -36,50 | 53,79 | 59,03 | 87,43 | -109,10 | -65,11 | -131,75 |
| ДНК (PDB ID 1pp8) | -55,33 | 30,46 | 34,71 | 135,53 | -141,44 | -100,24 | -123,78 |
Далее в цепи тРНК с помощью программ find_pairs и analyze были найдены группы спаренных остатков азотистых оснований. Результатом работы программ является файл 1gts.out, приведенный ниже.
RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.015) ....>B:...2_:[..G]G-----C[..C]:..71_:B<.... (0.014) |
2 (0.012) ....>B:...3_:[..G]G-----C[..C]:..70_:B<.... (0.005) |
3 (0.017) ....>B:...4_:[..G]G-----C[..C]:..69_:B<.... (0.010) |
4 (0.012) ....>B:...5_:[..G]G-----C[..C]:..68_:B<.... (0.008) |
5 (0.020) ....>B:...6_:[..U]U-----A[..A]:..67_:B<.... (0.011) |
6 (0.011) ....>B:...7_:[..A]A-----U[..U]:..66_:B<.... (0.014) |
7 (0.017) ....>B:..49_:[..C]C-----G[..G]:..65_:B<.... (0.010) |
8 (0.021) ....>B:..50_:[..G]G-----C[..C]:..64_:B<.... (0.008) |
9 (0.009) ....>B:..51_:[..A]A-----U[..U]:..63_:B<.... (0.017) |
10 (0.012) ....>B:..52_:[..G]G-----C[..C]:..62_:B<.... (0.012) |
11 (0.010) ....>B:..53_:[..G]G-----C[..C]:..61_:B<.... (0.007) |
12 (0.009) ....>B:..54_:[..U]U-**--A[..A]:..58_:B<.... (0.011) |
13 (0.015) ....>B:..55_:[..U]U-**+-G[..G]:..18_:B<.... (0.014) x
14 (0.010) ....>B:..37_:[..A]A-**--U[..U]:..33_:B<.... (0.017) |
15 (0.018) ....>B:..38_:[..U]U-*---U[..U]:..32_:B<.... (0.019) |
16 (0.014) ....>B:..39_:[..U]U-----A[..A]:..31_:B<.... (0.011) |
17 (0.011) ....>B:..40_:[..C]C-----G[..G]:..30_:B<.... (0.008) |
18 (0.010) ....>B:..41_:[..C]C-----G[..G]:..29_:B<.... (0.012) |
19 (0.008) ....>B:..42_:[..G]G-----C[..C]:..28_:B<.... (0.009) |
20 (0.010) ....>B:..43_:[..G]G-----C[..C]:..27_:B<.... (0.008) |
21 (0.012) ....>B:..44_:[..C]C-**--A[..A]:..26_:B<.... (0.014) |
22 (0.014) ....>B:..10_:[..G]G-----C[..C]:..25_:B<.... (0.008) |
23 (0.009) ....>B:..11_:[..C]C-----G[..G]:..24_:B<.... (0.016) |
24 (0.010) ....>B:..12_:[..C]C-----G[..G]:..23_:B<.... (0.015) |
25 (0.010) ....>B:..13_:[..A]A-**+-A[..A]:..45_:B<.... (0.012) |
26 (0.013) ....>B:..14_:[..A]A-**--U[..U]:...8_:B<.... (0.030) |
27 (0.013) ....>B:..15_:[..G]G-**+-C[..C]:..48_:B<.... (0.011) x
28 (0.015) ....>B:..19_:[..G]G-----C[..C]:..56_:B<.... (0.009) +
Note: This structure contains 8[7] non-Watson-Crick base-pairs.
Cтебли образуются при комплементарном взаимодействии следующих пар участков:
B2-7:----:B71-66
B49-53:----:B65-61
B39-44:----:B31-26
B10-12:----:B25-23
Кроме водородных связей, поддерживающих стебли, можно заметить другие, стабилизирующие третичную структуру тРНК:
B:..54_:[..U]U-**--A[..A]:..58_:B
B:..55_:[..U]U-**+-G[..G]:..18_:B
B:..37_:[..A]A-**--U[..U]:..33_:B
B:..38_:[..U]U-*---U[..U]:..32_:B
B:..13_:[..A]A-**+-A[..A]:..45_:B
B:..15_:[..G]G-**+-C[..C]:..48_:B
B:..13_:[..A]A-**+-A[..A]:..45_:B
B:..14_:[..A]A-**--U[..U]:...8_:B
B:..15_:[..G]G-**+-C[..C]:..48_:B
В структуре присутствует 8 неканонических пар, они и указаны выше.
А так же еще одна пара: B:..44_:[..C]C-**--A[..A]:..26_:B
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 3.94( 2.63) 0.00( 0.00) 0.11( 0.00) 0.45( 0.00) 4.50( 2.63)
2 GG/CC 3.47( 2.13) 0.00( 0.00) 0.32( 0.00) 0.06( 0.00) 3.85( 2.13)
3 GG/CC 4.03( 2.40) 0.00( 0.00) 0.43( 0.00) 0.00( 0.00) 4.46( 2.40)
4 GU/AC 6.72( 4.06) 0.00( 0.00) 0.00( 0.00) 3.98( 2.46) 10.69( 6.52)
5 UA/UA 0.57( 0.00) 0.00( 0.00) 0.99( 0.81) 0.27( 0.00) 1.83( 0.81)
6 AC/GU 3.17( 1.94) 0.00( 0.00) 0.00( 0.00) 6.23( 3.27) 9.40( 5.21)
7 CG/CG 0.00( 0.00) 0.00( 0.00) 5.38( 2.44) 0.00( 0.00) 5.38( 2.44)
8 GA/UC 4.81( 3.25) 0.00( 0.00) 0.00( 0.00) 2.38( 0.36) 7.19( 3.62)
9 AG/CU 2.80( 2.62) 0.00( 0.00) 0.43( 0.00) 0.16( 0.00) 3.38( 2.62)
10 GG/CC 3.44( 1.90) 0.00( 0.00) 1.26( 0.00) 0.00( 0.00) 4.70( 1.90)
11 GU/AC 6.64( 3.76) 0.00( 0.00) 0.00( 0.00) 4.74( 1.88) 11.38( 5.63)
12 UU/GA 4.64( 1.93) 0.00( 0.00) 0.00( 0.00) 6.32( 3.38) 10.96( 5.31)
13 UA/UG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
14 AU/UU 4.44( 0.95) 0.00( 0.00) 0.00( 0.00) 4.06( 2.00) 8.49( 2.95)
15 UU/AU 1.18( 0.28) 0.00( 0.00) 0.00( 0.00) 6.21( 3.83) 7.39( 4.11)
16 UC/GA 0.00( 0.00) 0.00( 0.00) 0.02( 0.00) 2.71( 1.21) 2.73( 1.21)
17 CC/GG 0.31( 0.00) 0.00( 0.00) 0.45( 0.00) 3.81( 2.49) 4.57( 2.49)
18 CG/CG 0.00( 0.00) 0.00( 0.00) 5.89( 2.70) 0.00( 0.00) 5.89( 2.70)
19 GG/CC 3.71( 2.31) 0.00( 0.00) 0.60( 0.00) 0.06( 0.00) 4.37( 2.31)
20 GC/AC 6.71( 3.74) 0.00( 0.00) 0.00( 0.00) 4.45( 2.11) 11.16( 5.85)
21 CG/CA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
22 GC/GC 4.62( 1.70) 0.00( 0.00) 0.00( 0.00) 5.86( 2.85) 10.47( 4.54)
23 CC/GG 0.06( 0.00) 0.00( 0.00) 0.34( 0.00) 2.63( 1.12) 3.03( 1.12)
24 CA/AG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
25 AA/UA 0.00( 0.00) 2.55( 0.10) 0.00( 0.00) 0.00( 0.00) 2.55( 0.10)
26 AG/CU 3.78( 1.42) 0.00( 0.00) 0.04( 0.00) 0.00( 0.00) 3.82( 1.42)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
****************************************************************************
Note: This structure contains 8[7] non-Watson-Crick base-pairs.
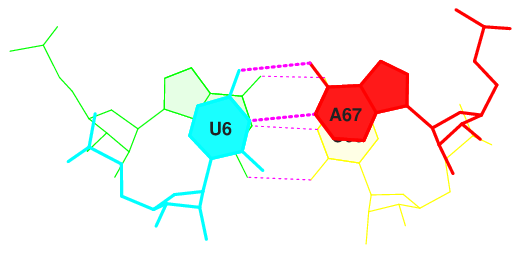
В таблице из фрагмента файла 1gts.out последнем столбце вне скобок указана площадь пересечения между многоугольниками, образованными атомами нуклеотидов, в скобках — только пересечение многоугольников, образованных кольцом азотистого основания. Таким образом, реальное стекинг-взаимодействие по предположению должно быть наиболее сильным для той четверки оснований, где число в скобках максимально.
Это верно для четвёртой четвёрки оснований GU/AC.
B:...5_:[..G]G-----C[..C]:..68_:B
B:...6_:[..U]U-----A[..A]:..67_:
В файле stacking.pdb это Section #0004. Далее было получено стандартное изображение стекинг-взаимодействия. Картинка ниже.
 Term 3
Main page
Term 3
Main page