Для работы был выбран домен B_lectin, способный специфически связывать D-маннозу, находящуюся на поверхности клеток.
Данный домен (AC в Pfam: PF01453) имеется в 3619 известных белках и встречается в составе 201 архитектуры. Все виды архитектур представлены по ссылке.
Для дальнейшей работы было выбрано 2 архитектуры:

Первая архитектура

Вторая архитектура
Первая архитектура встречается в 232 последовательностях. В ее состав входят 3 домена: B_lectin, S_locus_glycop и Pkinase.
S_locus_glycop, как указано в описании на сайте Pfam (AC в Pfam: PF00954), является гликопротеином, участвующим в системе узнавания свой/чужой у растений. За работу данной системы отвечает набор аллелей в S-локусе. Данный домен был назван доменом, так как рядом с ним расположены другие известные домены.
Домен Pkinase (AC в Pfam: PF00069) входит в состав протеинкиназ - белков, способных фосфорилировать другие белки, то есть переносить на них фосфатную группу (обычно гамма-фосфат из NTP, например, ATP). Этот процесс приводит к активации или инактивации эффекторных белков и, следовательно, играет важную роль во многих клеточных процессах.
Вторая архитектура встречается в 181 последовательности. В ней также присутствуют 3 домена: B_lectin, S_locus_glycop и PAN_2.
Про функции домена PAN_2 (AC в Pfam: PF08276) практически ничего не извество. Похожий на него домен PAN_1 содержит консервативное ядро с тремя дисульфидными связями. Возможные функции - регуляция белок-белковых взаимодействий или взаимодействий белок-углевод.
Для построения выравнивания было получено выравнивание всех белков, содержащих этот домен (их доменных участков). Это выравнивание доступно в формате fasta и в проекте JalView
Также была создана таблица в Excel, содержащая информацию обо всех последовательностях белков, имеющих данный домен B_lectin (лист "proteins"), и об их таксономической принадлежности (лист "taxonomy"). Сводная таблица находится на листе "all", отобранные для выравнивания последовательности - на листе "selected".
Ссылка на таблицу: info.xlsx. Стоит отметить, что таблицы, содержащие таксономию, сложно редактировать, так как каждая последовательность описана с разной степенью точности, а у многих указанных таксонов нет общепринятого ранга. Поэтому в таблице часто встречаются названия столбцов "нет ранга", а распределение таксонов по столбцам соответсвуюет наиболее представленным организмам, с которыми и производилась дальнейшая работа.
Затем был выбран таксон Magnoliophyta (можно считать его аналогом отдела Покрытосеменные растения) и два его подтаксона: Eudicotyledons (безранговый таксон Эвдикоты, к которому в основном относят пердставителей класса Двудольные) и Liliopsida (класс Лилейные или Однодольные). Были отобраны последовательности белков с описанными доменными архитектурами из организмов, принадлежащих к данным подтаксонам, и составлено их выравнивание (на основании полного выравнивания всех представителей домена). Данное выравнивание доступно в формате fasta и в проекте JalView. Стоит отметить, что из выравнивания предвариетльно были удалены плохо выровненные последовательности, пустые гэповые колонки и участки с N- и C-концов последовательностей. Белки, имеющие разные доменные архитектуры, объединены в группы Pkinase и PAN2 (по названию отличающегося домена).
На основании полученного выравнивания было построено филогенетическое дерево последовательностей домена. Для построения использовался метод Maximum Likelihood в программе MEGA. Изображение дерева представлено на рисунке 3. На рисунке 4 представлено неукорененное круговое дерево. Дерево в Newick-формате (скобочная формула): tree.nwk.
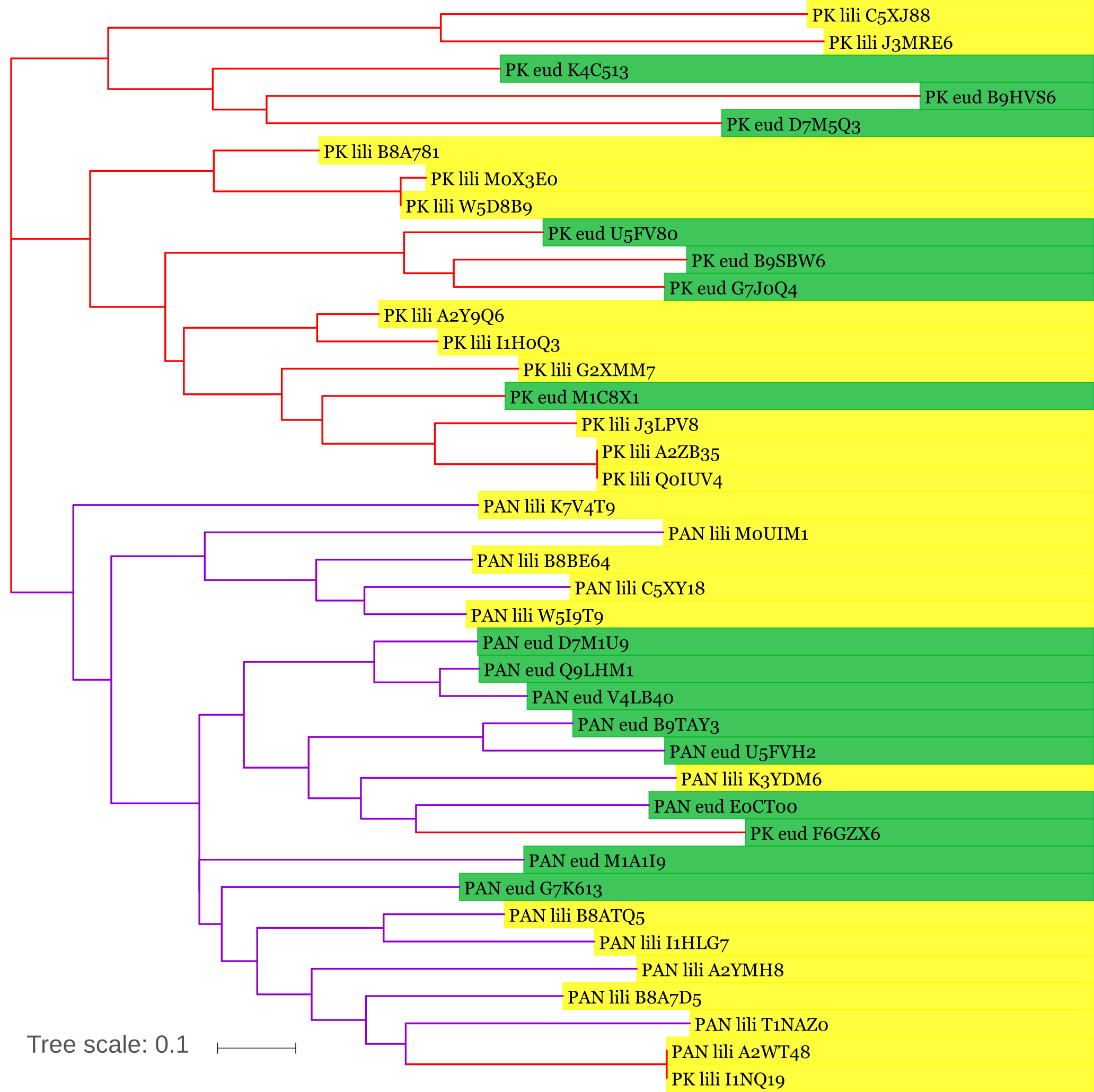
Изображение укорененного дерева, полученное из выравнивания доменов B_lectin из белков с разной доменной архитектурой: PAN (фиолетовые ветви на дереве) - архитектура из рисунка 2, PK (красные ветви) - архитектура из рисунка 1. Желтым отмечены последовательности из таксона Liliopsida, зеленым - из таксона Eudicotyledons. Изображение получено с помощью Itol.
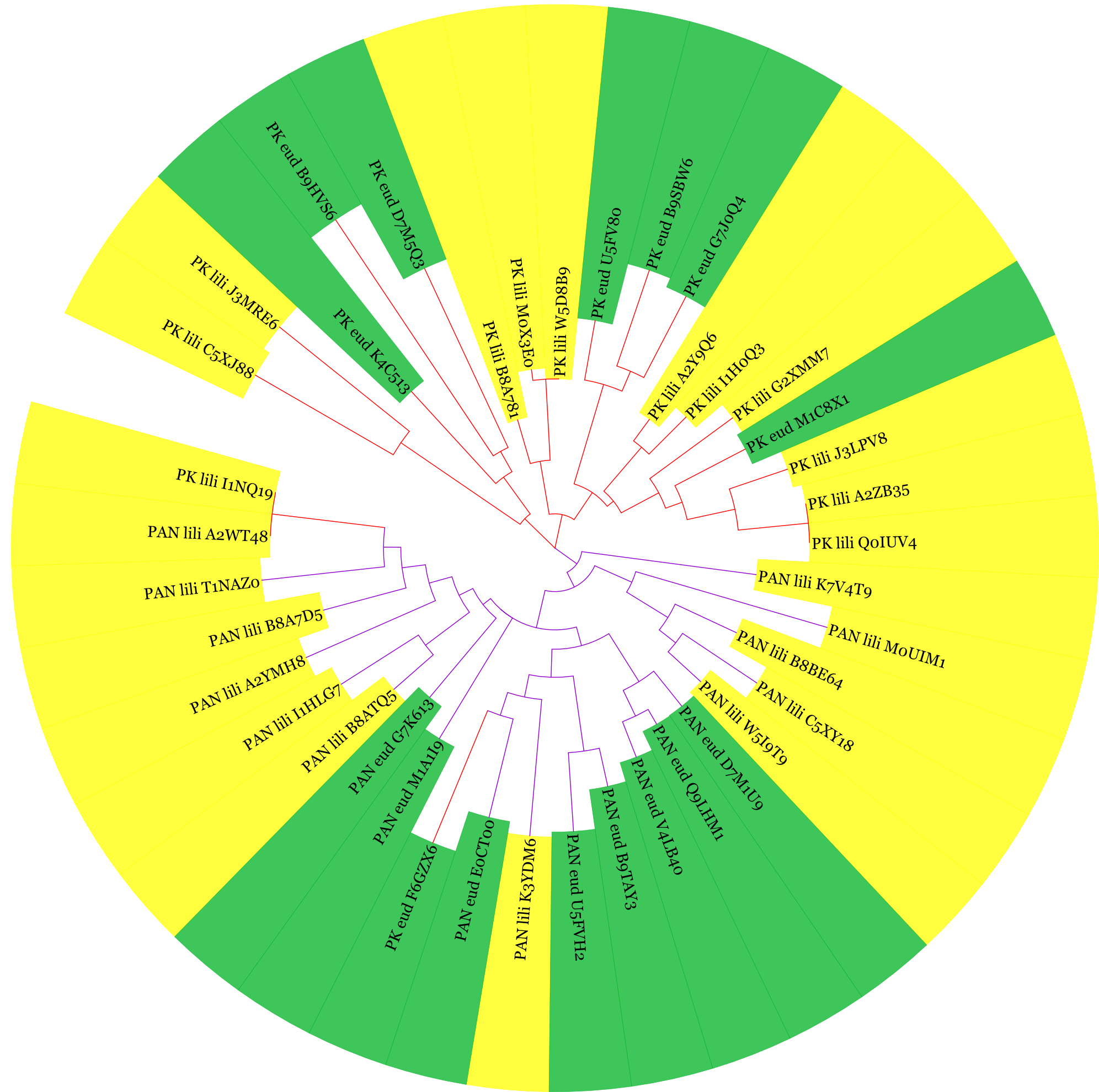
Изображение укорененного дерева, полученное из выравнивания доменов B_lectin из белков с разной доменной архитектурой: PAN (фиолетовые ветви на дереве) - архитектура из рисунка 2, PK (красные ветви) - архитектура из рисунка 1. Желтым отмечены последовательности из таксона Liliopsida, зеленым - из таксона Eudicotyledons. Изображение получено с помощью Itol.
По рисункам видно, что большинство белков, имеющих разную доменную архитектуру, принадлежат к разным кладам на дереве. Скорее всего, это связано не с ошибкой при построении выравнивания, так как вырванивание и внутри архитектур, и всех последовательностей в целом достаточно хорошее, последовательности гомологичны, а плохо выровненные последовательности были удалены. Возможно, был общий предок для этих двух доменных архитектур (например, с двумя доменами, которые для них являются общими), а затем некоторые белки приобрели домен с протеинкиназной активностью, а другие - домен из семейства PAN. Таким образом, белки функционально разошлись. При этом с точки зрения таксономии распределение архитектур в таксонах Liliopsida и Eudicotyledons примерно одинаковое и слабо выражено на дереве. Вероятно, эти белки являются консервативными для всех групп покрытосеменных растений. Стоит отметить, что есть 2 последовательности из первой (PK) архитектуры, которые оказались более близкими к белкам со второй (PAN) архитектурой, чем к белкам с PK-архитектурой. Это PK_eud_F6GZX6 и PK_lili_I1NQ19 (на рисунках - красные ветви среди фиолетовых). Это может быть связано с тем, что данные белки оказались взяты из того же организма или из очень близкого его родственника, или же они разделились на очень позднем этапе, поэтому их общие домены не успели сильно разойтись при независимой эволюции.
Для построения профиля было выбрано подсемейство, отмеченное на рисунке ниже. Затем с помощью программы HMMER был построен и откалиброван профиль по выравниванию отобранных последовательностей. Для этого использовались программы hmm2build и hmm2calibrate.
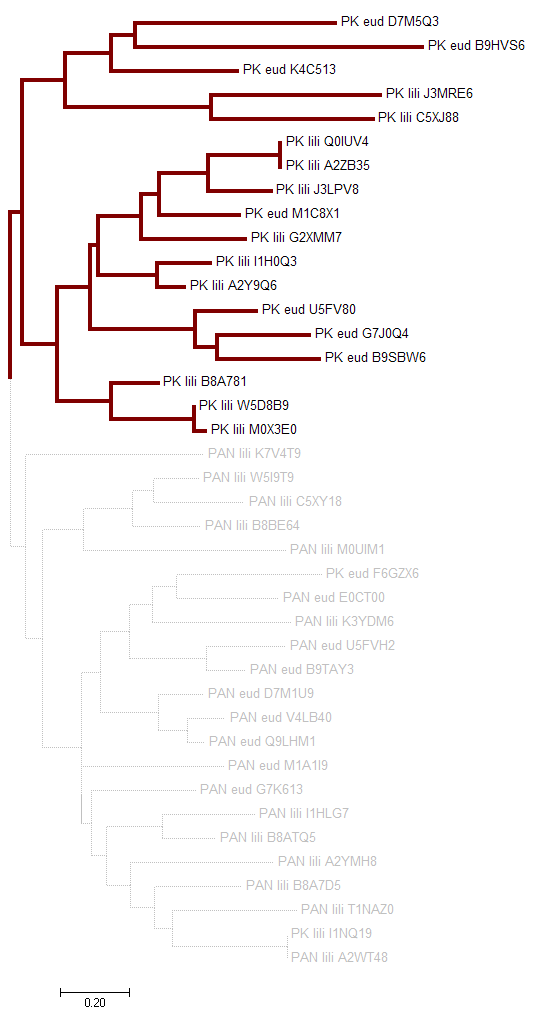
Филогенетическое дерево доменных архитектур из предыдущего задания. Бордовым отмечено подсемейство, выбранное для построения профиля.
С помощью программы hmm2search с использованием полученного профиля был проведен поиск по всем белкам UniProt, содержащим домен B_lectin. В результате был получен следующий файл.
Данные о всех находках и их E-value были перенесены в таблицу Excel (ссылка). Они представлены на листе "profile". Находки были отсортированы по увеличению E-value. Далее был создан столбец "profile", в котором отмечены последовательности (среди всех находок), участвовавшие в построении профиля (ИСТИНА), а также все остальные находки (ЛОЖЬ). На основании полученных данных была построена ROC-кривая (рисунок 6). Ее изображение и этапы построения представлены на листе "ROC".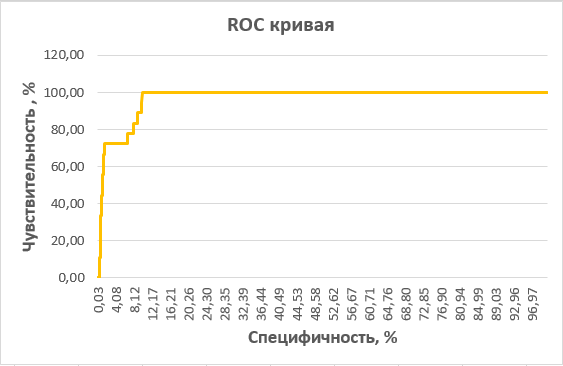
ROC-кривая, построенная по результатам поиска по профилю. По оси X - специфичность, по оси Y - чувствительность профиля (в %).
С помощью ROC-кривой был выбран порог E-value, по которому можно судить о принадлежности последовательности подсемейству. Пороговое значение E-value оказалось равным 7,6e-43. Значения при выбранном пороге представлены в таблице ниже.
| Таблица 1. Число последовательностей, найденных по профилю при пороге E-value 7,6e-43. | |||
| последовательность | принадлежит подсемейству | не принадлежит подсемейству | сумма |
| выше порога по профилю | 13 | 37 | 50 |
| ниже порога по профилю | 5 | 3300 | 3305 |
| сумма | 18 | 3337 | 3355 |