Для построения дерева были взяты последовательности 16s рРНК перечисленных в таблице 1 бактерий. Они были получены из базы полных геномов NCBI. Выбранные последовательности представлены в файле16s.fasta.
С помощью Muscle было построено выравнивание полученных последовательностей. Выравнивание в fasta-формате: align.fasta .С помощью программы MEGA методом Neighbor-Joining было построено филогенетическое дерево последовательностей по выравниванию.
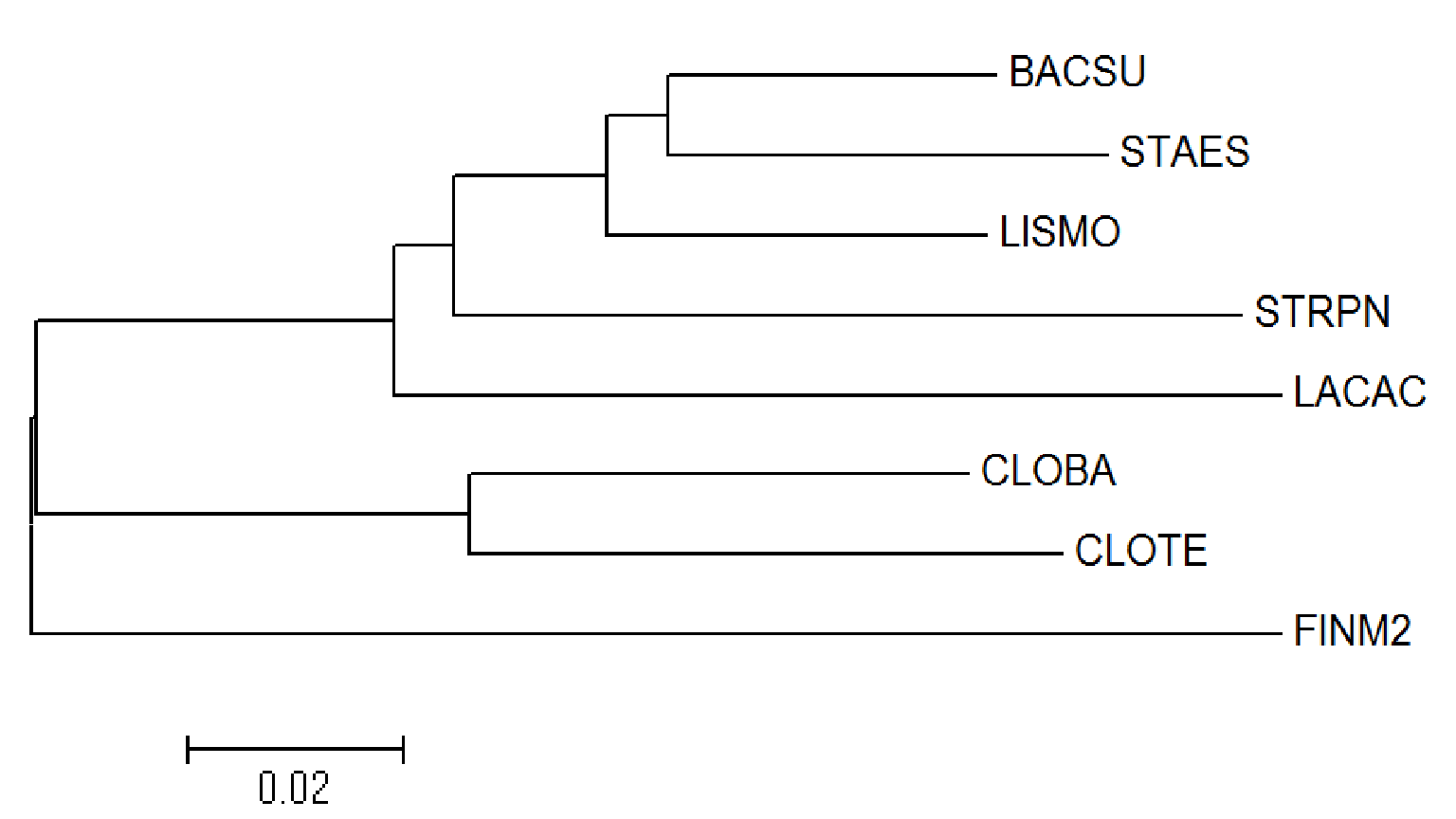
дерево построенное по последовательностям 16s рРНК алгоритмом Neighbor-Joining
В целом, существенных различий в качестве построения деревьев по белковым или по нуклеотидным последовательностям не наблюдалось.В данном задании необходимо реконструировать дерево по достоверным гомологам белка "ATP-dependent Clp protease ATP-binding subunit ClpX OS=Bacillus subtilis". В Таблице 1 представлены выбранные гомологичные белки и сопоставленные им мнемоники, поиск гомологичных белков осуществлялся с помощью blastp на сайте NCBI с ограничениям по нужным организмам. Все находки с e-value значительно ниже 0,001
Далее все белки были выравнены программой Musle в Mega - выравнивание
Потом было построено дерево методом Neighbor-Joining.
Таблица 1
| Обозначение |
Белок |
|---|---|
| Clpx | ATP-dependent protease ATP-binding subunit ClpX |
| hsiu | ATP-dependent protease ATP-binding subunit HslU |
| AAA | ATPase AAA |
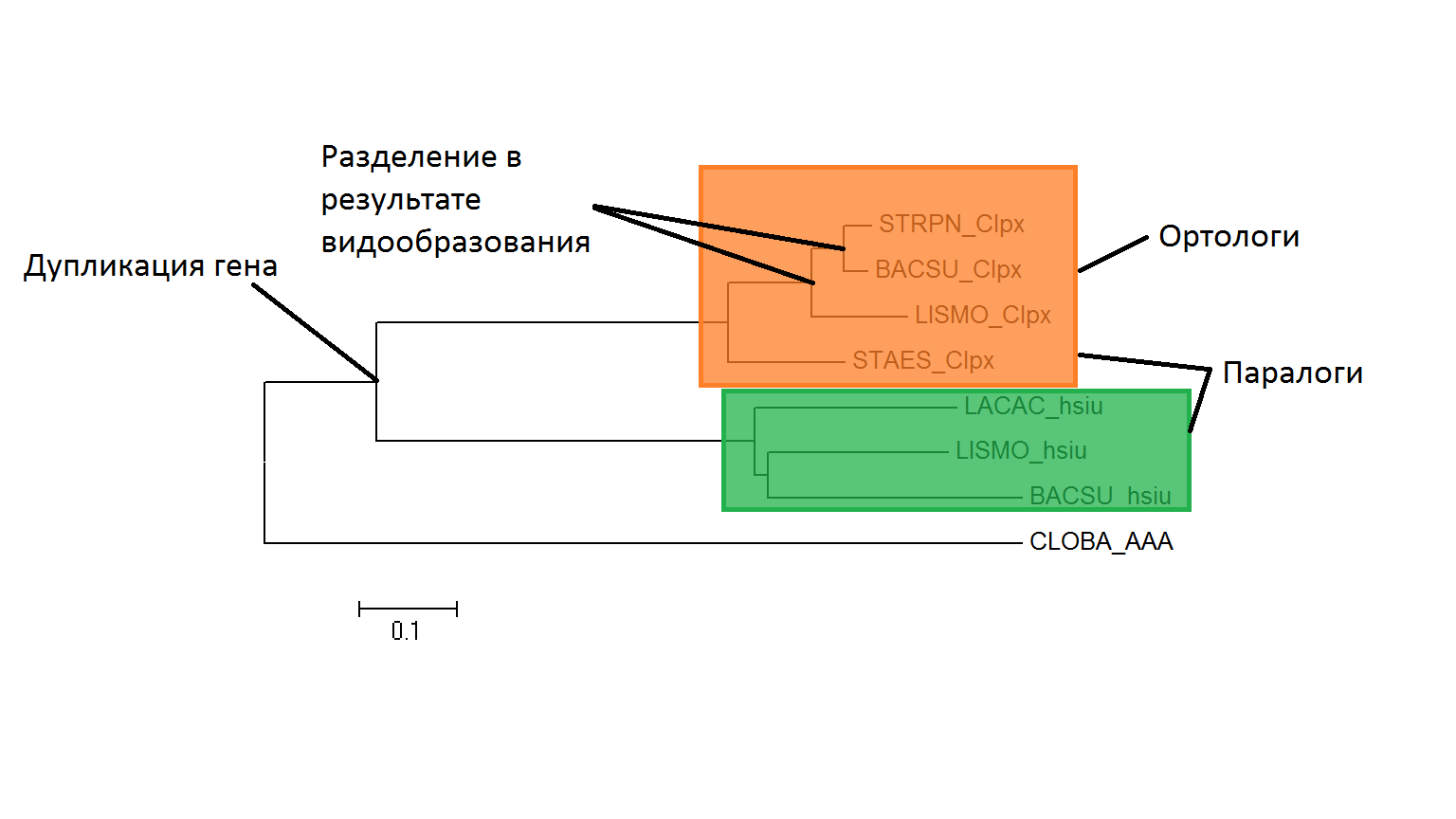
Дерево гомологичных белков
Term 4 Main page