1. Проверка гомологичности белков, найденных поиском по сходству
Для выполнения этого задания я использовала белок ANE87960.1, репарирующий алкилированные основания из генома Bacillus cereus. Поиск велся с помощью программы Blast в базе reference proteins, алгоритм blastp, размер слов составлял 6, матрица сравнения BLOSUM62, штраф за открытие гэпа: 10, за продоление гэпа: 1. Для того, чтобы найти последовательности с большим E-value, поиск велся с исключением представителей своего семейства, отдела и даже домена.| ID/AC | Coverage, % | E-value | Identity,% | Число инделей | Заключение о гомологичности |
| YP_009153656.1 | 16 | 39 | 3,1 | 4 | нет |
| WP_048169902.1 | 84 | 0,024 | 22 | 3 | да |
| WP_015264859.1 | 94 | 1e-58 | 48 | 6 | да |
| WP_013767680.1 | 94 | 5e-54 | 42 | 6 | да |
| WP_070109472.1 | 88 | 1e-50 | 46 | 8 | да |
| WP_049737686.1 | 84 | 3e-65 | 55 | 7 | да |
| WP_064200979.1 | 91 | 5e-70 | 51 | 7 | да |
| WP_071734573.1 | 100 | 9e-150 | 95 | 5 | да |
| WP_017150200.1 | 97 | 5e-126 | 80 | 6 | да |
На рисунке 1 показано множественное выравнивание для предположительно гомологичных участков упомянутых выше белков. Чтобы судить о гомологичности, я выделила несколько консервативных блоков (участки с высоким содержанием консервативных позиций для всех белков, входящих в блок, и не содержащих гэпы). Ссылка на выравнивание. Как видно из рисунка 1, белок YP_009153656.1 входит только в 1 такой блок, да к тому же принадлежит бактериофагу и является субъединицей эндонуклеазы, поэтому я считаю, что его нельзя назвать гомологичным моему белку.
WP_048169902.1 этот белок, репарирующий алкилирвоанные основания у метаногенной археи. Его последовательность входит в 3 блока множественного выравнивания (в 1-ый я его не включаю, тк мало консервативных позиций в этом участке). И несмотря на то, что данный белок идентичен по отношению к моему на 22 %, в нем есть высококонсервативные блоки, которые позволяют судить о гомологичности данных белков. Остальные белки я могу назвать гомологичными, по отношению к своему, тк они образуют несколько консервативных блоков и имеют большой (от 50 до 90) процент идентичных позиций. Кроме того они все выполняют функцию реапарции алкилированных оснований.

2. Описание крупных перестроек между парами белков, имещих гомологичные домены
Для выполнения этого задания выбрала семейство белков, содержащих KH-домен [1]. Он состоит из примерно 70 аминокислот и присутствует в широком спектре белков, связывающихся с нуклеиновыми кислотами, в частности, с РНК. Одним из самых известных белков, содержащих данные домены, является KSRP, регулирующий вреся жизни мРНК путем связывания с ее 3`нетранслируемыми регионами [2]. Я выбрала белки X6DCS0_9RHIZ, нуклеотид трансферазу и U9TJE9_RHIID, белок, без четко выявленной функции. Оба этих белка найдены у бактерий, образующих микоризы. Для поиска использовалась длина слова 2 и пороговое E-value =1.0E-6. Карта выравнивания представлена на рисунке 2.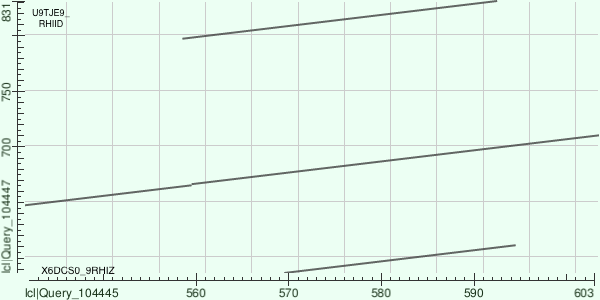
1. (587-611) Identity: 35%, Similarity: 60%, Gap:1%
2. (648-707) Identity: 38%, Similarity: 67%, Gap:0%
3. (798-831) Identity: 44%, Similarity: 64%, Gap:0%
Иллюстрация первых двух участков представлена на рисунке 3.
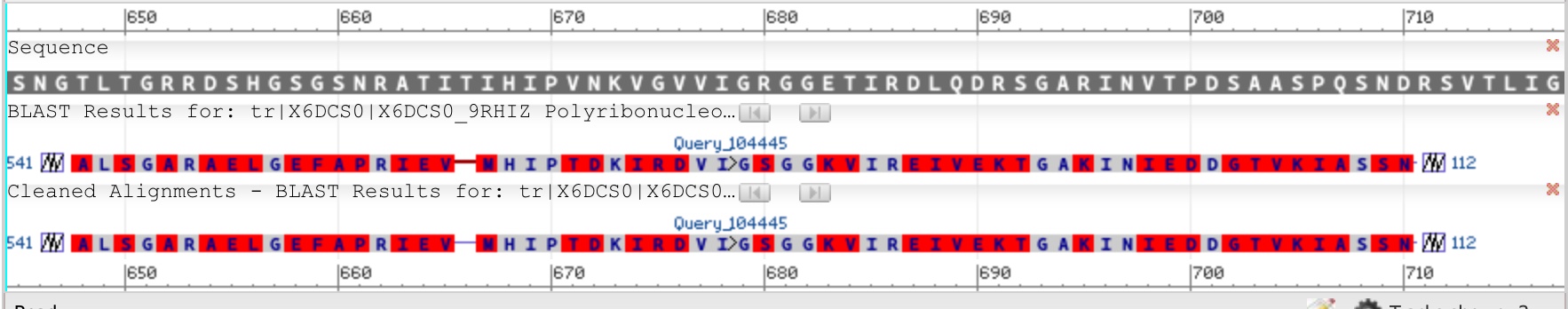
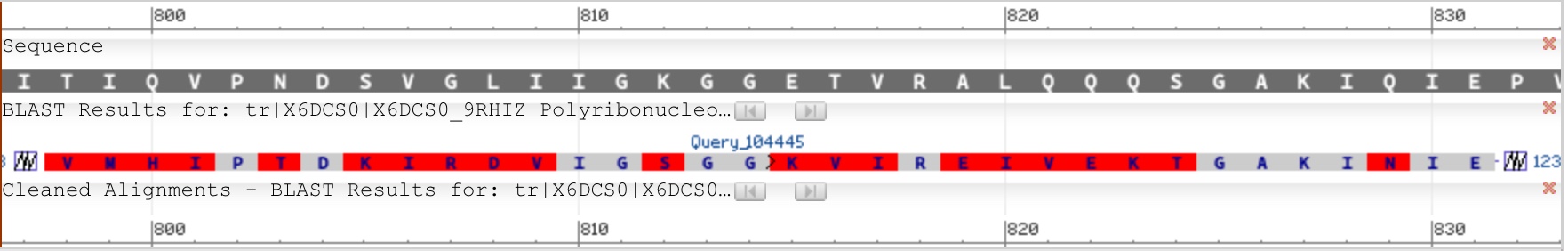
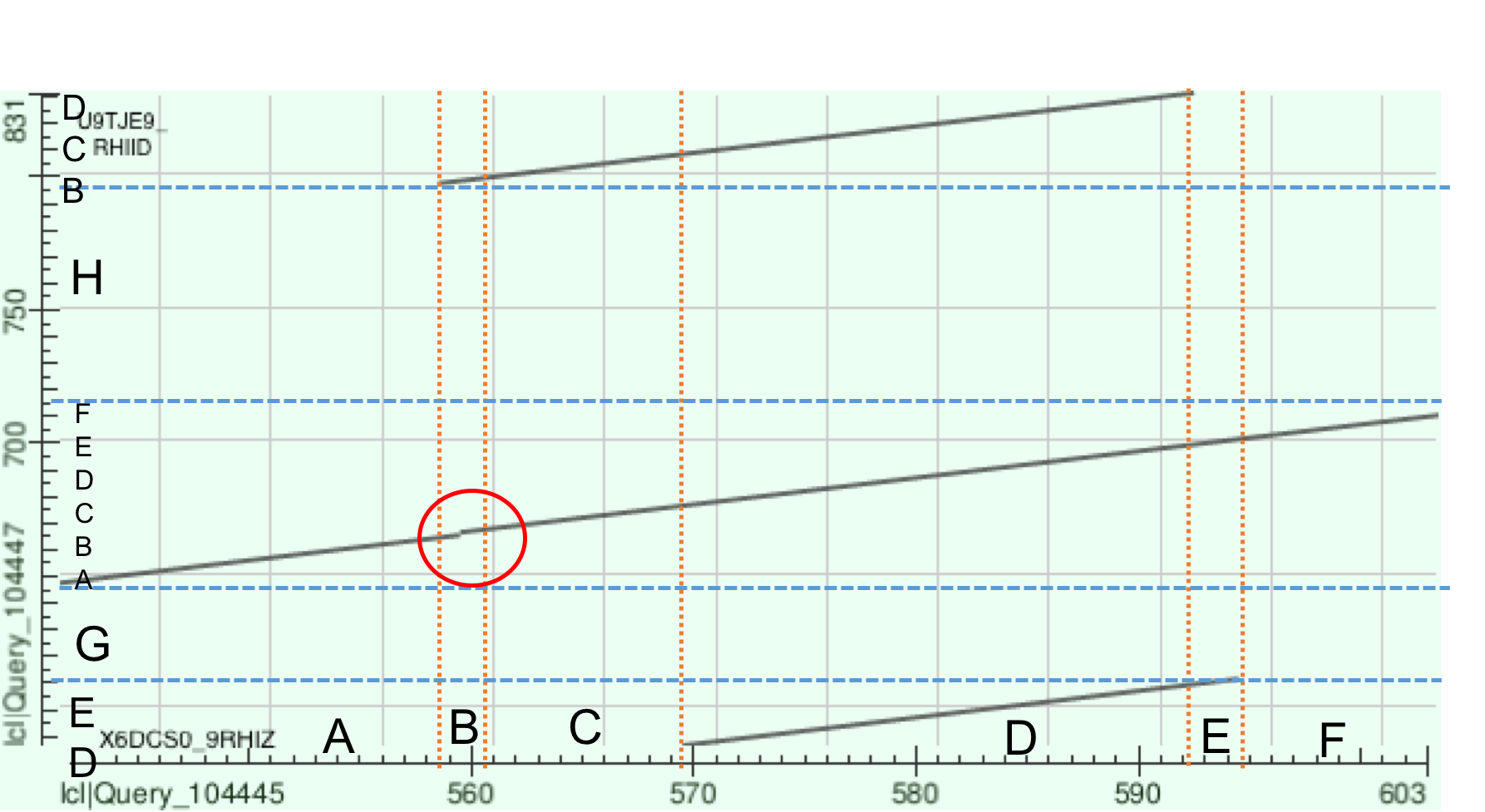
Затем мной был произведен более подробный анализ карты выравнивания X6DCS0_9RHIZ и U9TJE9_RHIID. По горизонтальной оси отложен X6DCS0_9RHIZ , по вертикальной - U9TJE9_RHIID. Для удобства я разметила линиями основные участки и обозначила различными латинскими буквами (одинаковыми буквами-одинаковые участки). В итоге получилась такая картина:
X6DCS0_9RHIZ: ABCDEF
U9TJE9_RHIID: DEGABCDFEFHBCD.
Кроме того можно заметить небольшую делецию, произошедшую в U9TJE9_RHIID ( выделена красным кругом).

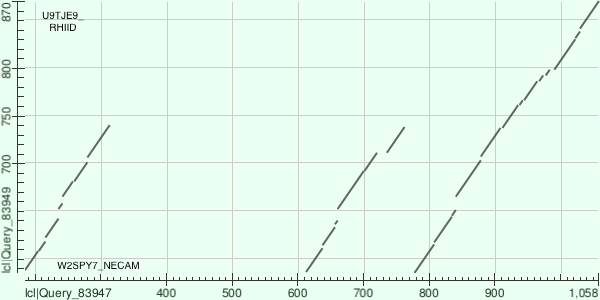
Потом мне стало интересно сравнить два белка, имеющих различное количество таких доменов. Поэтому я сравнила U9TJE9_RHIID с W2SPY7_NECAM. Параметры для выравнивания остались такими же. Результат представлен на рисунке 5. Как можно видеть, то в белке W2SPY7_NECAM нашлось 3 домен, гомологичных участку 1 U9TJE9_RHIID, 3 домена, гомологичных участку 2, и 1 домен, гомологичный участку 3. Из этого можно сделать вывод, что функция каждого из утроенного домена изменяется, каждый из них эволюционирует по-своему, приобретает новое свойство, которое закрепляется в процессе эволюции.
3. Список литературы
[1] http://pfam.xfam.org/family/PF00013#tabview=tab0
[2] https://www.ncbi.nlm.nih.gov/pubmed/19458619