1. Выберите три генома бактерий или архей одного вида
Из предложенного списка бактерий я выбрала 3-х бактерий : Bacillus cereus strain FORC_005 (NZ_CP009686), Bacillus cereus 172560W (NZ_CM000717), Bacillus cereus 95/8201 (NZ_CM000727). Основными критериями выбора организмов были представленность генома одной хромосомой, принадлежность их к одному виду, наличие крупных геномных перестроек, большая суммарная длина гомологичных участков.2. identity % на гомологичных участках геномов и покрытие геномов гомологичными участками
Для выполнения этого задания я воспользовлась пакетом NPG-explorer, установленным на Kodomo (параметры MIN_IDENTITY = Decimal('0.8').1) Для определения покрытия геномов гомологичными участками я воспользовалась выдачей файла blocks.gbi. Данные колонки"s_blocks_length" для блоков g (состоят из последовательно идущих во всех геномах s-блоков, перемежающихся блоками других типов) были просуммированы в excel и получена длина гомологичных участков для всех 3 бактерий =4118795. Затем длина гомологичных участков была поделена на длину генома каждой бактерии. Полученные результаты представлены в Таблице 1.
| Организм | Длина генома, п.о. | Процент покрытия |
| Bacillus cereus strain FORC_005 | 5 349 617 | 76,99% |
| Bacillus cereus 172560W | 5 699 545 | 72,27% |
| Bacillus cereus 95/8201 | 5 584 055 | 73,76% |
2) Для определения сходства на гомологичных участках я воспользовалась выдачей файла pangenome.info. В абзаце с описанием stem blocks (блоков, представленных во всех геномах) указано значение Identity: 0.911097.
3. Исследуйте крупные перестройки
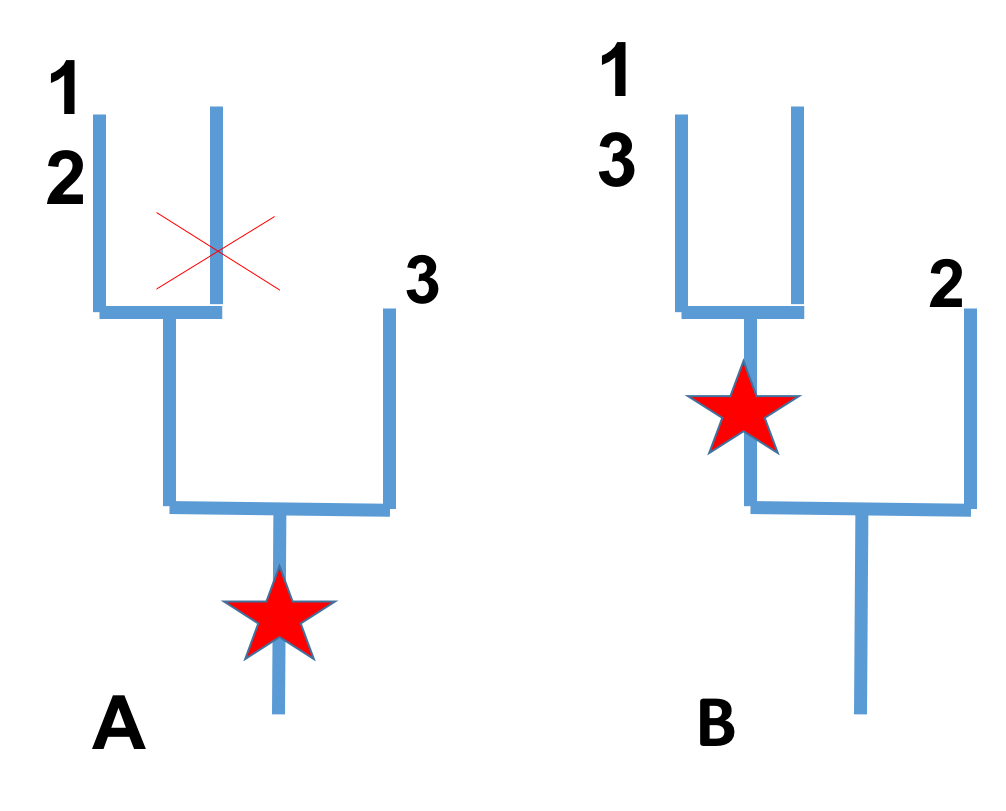
1) Крупные делеции/вставки. Чтобы найти крупную делецию, а анализировала файл pangenome.bi блоки h, показывающие участки, содержащиеся в нескольких (не во всех) геномах. В последних колонках этого файла, указано, в каких организмах присутствуют данные блоки. Для описания примера гена, имеющегося у двух бактерий и не имеющегося у третьей я взяла блок h2x1000 (информацию получила из файла pangenome.bi, который содержится в 1 и 3 бактерии (нумерация в том порядке, в каком они упомянуты выше). В этом блоке содержится белок, принадлежащией к TetR/AcrR семейству транскрипционных регуляторов, protein_id="WP_000074940.1" (4873918..4874466) у 1 бактерии и protein_id="WP_000238605.1"(4973875..4974504) у 2 бактерии. Оба белка закодированы на обратной цепочке ДНК. Чтобы понимать, произошла ли это вставка у предка 1 и 3 или делеция у 2, необходимо знать филогенетические взаимоотношения между организмами. Варианты развития событий представлены на рисунке 1.
| Bacillus cereus strain FORC_005 | Bacillus cereus 172560W | Bacillus cereus 95/8201 | |
| Bacillus cereus strain FORC_005 | Query cover 93%, Identity 99% | Query cover 86%, Identity 94% | |
| Bacillus cereus 172560W | Query cover 81%, Identity 94% |
2) Участки, имеющиеся в одном геноме и отсуствующие в двух других
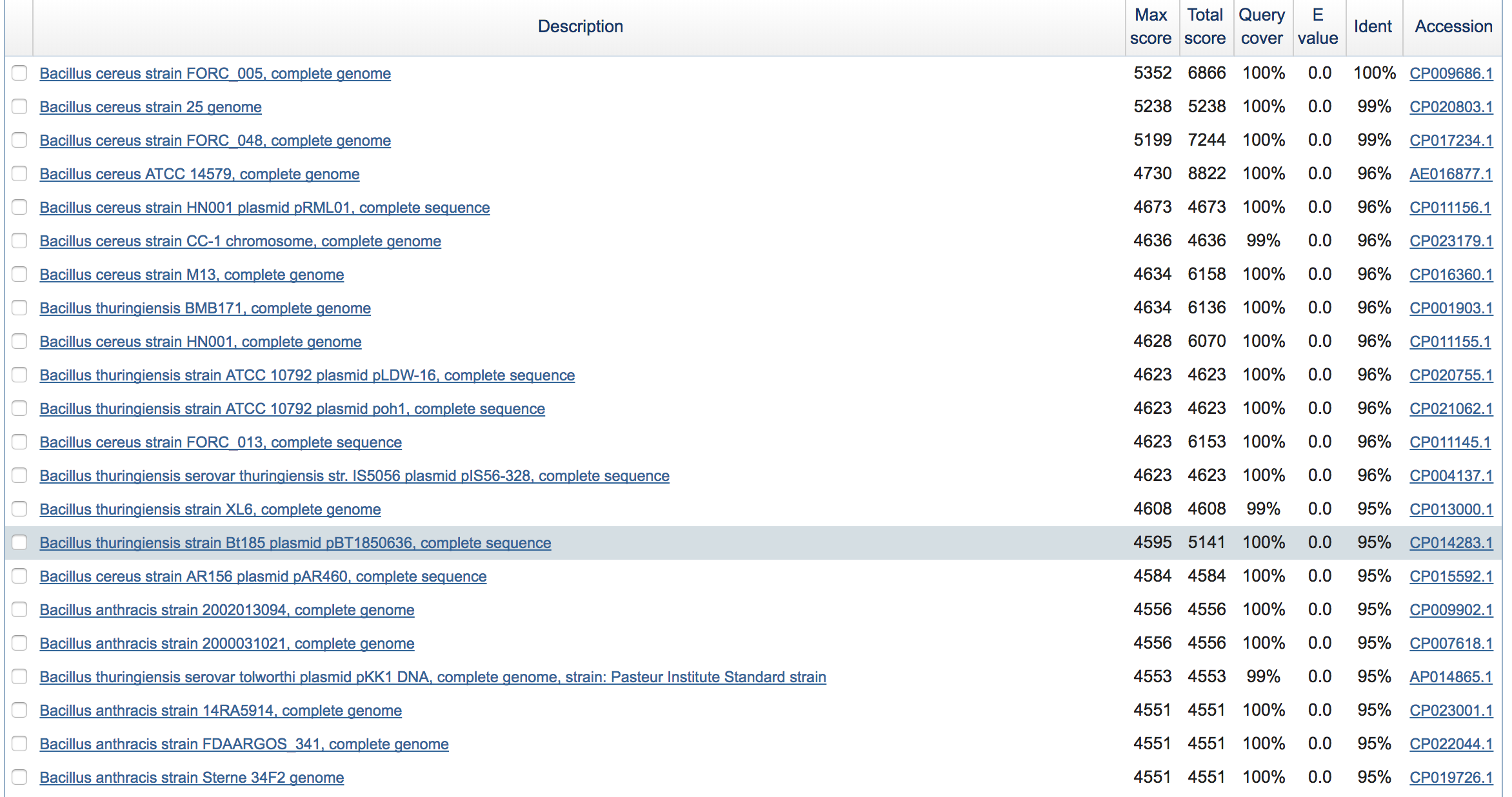
Для описания такого примера я взяла блок u1x892, уникальный для бактерии 1. В этом блоке содержится последовательность гена NP_832902.1, кодирующего коллагеназу (collagenase) (координаты 2450419 до 2453316). Так как мы исходим из предположения, что бактерии 1 и 2 ближе друг к другу, чем 3, то более вероятно что наблюдаемое событие - вставка в геном бактерии 1, так как для этого требуется 1 эволюционное преобразование, а не делеция этого гена у 2 и 3 (так как этот ген закодирвоан в основной хромосоме, а не в плазмиде, потерять его довольно сложно). Чтобы понять, откуда появился этот ген (например, с помощью горизонтального переноса), я попыталась найти гомологичную последовательность выбранной последовательности с помощью megablast (параметры, отличные от стандартных -evalue=0.001). Результат поиска представлен на рисунке 2.
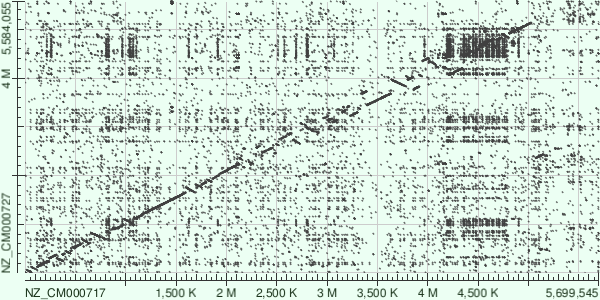
3)Инверсии. Чтобы ответить на этот вопрос я сравнила геномы 1 и 3 бактерий с помощью blast2seq. Карта локального сходства представлена на рисунке 3.
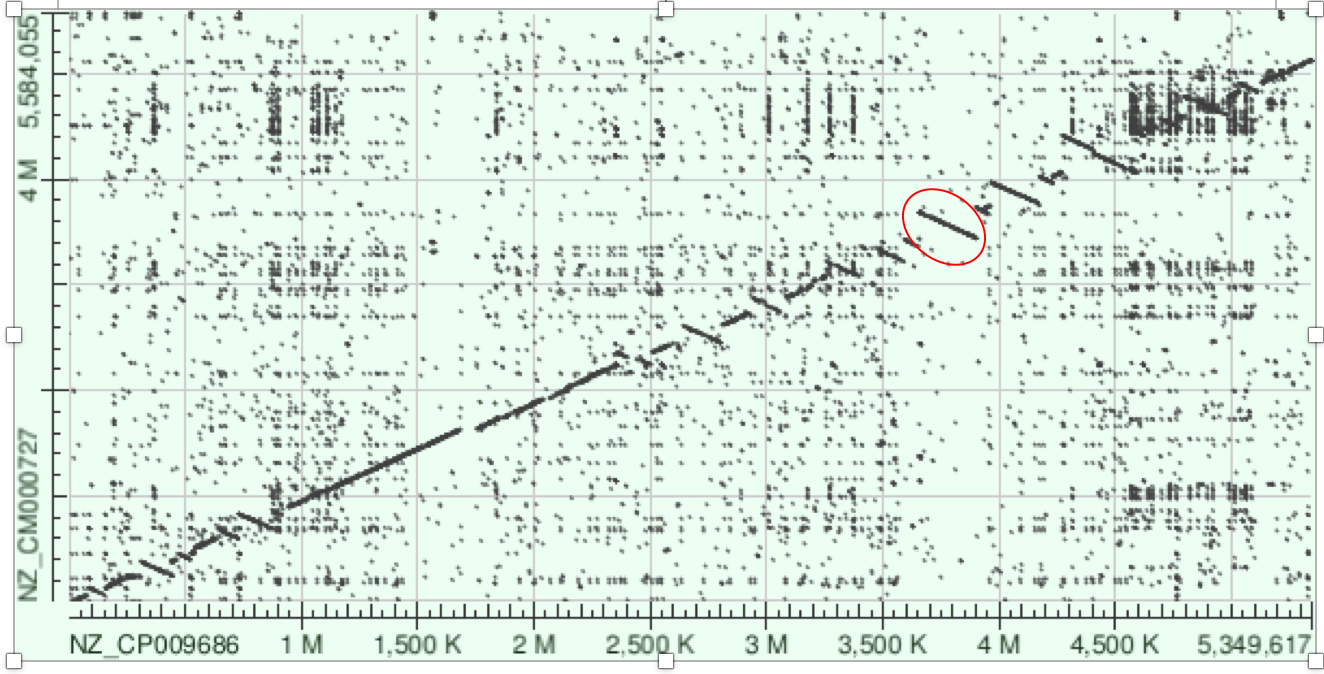
4)Синтетические участки . Чтобы ответить на этот вопрос я сравнила геномы 1 и 2 бактерий с помощью blast2seq. Карта локального сходства представлена на рисунке 4.
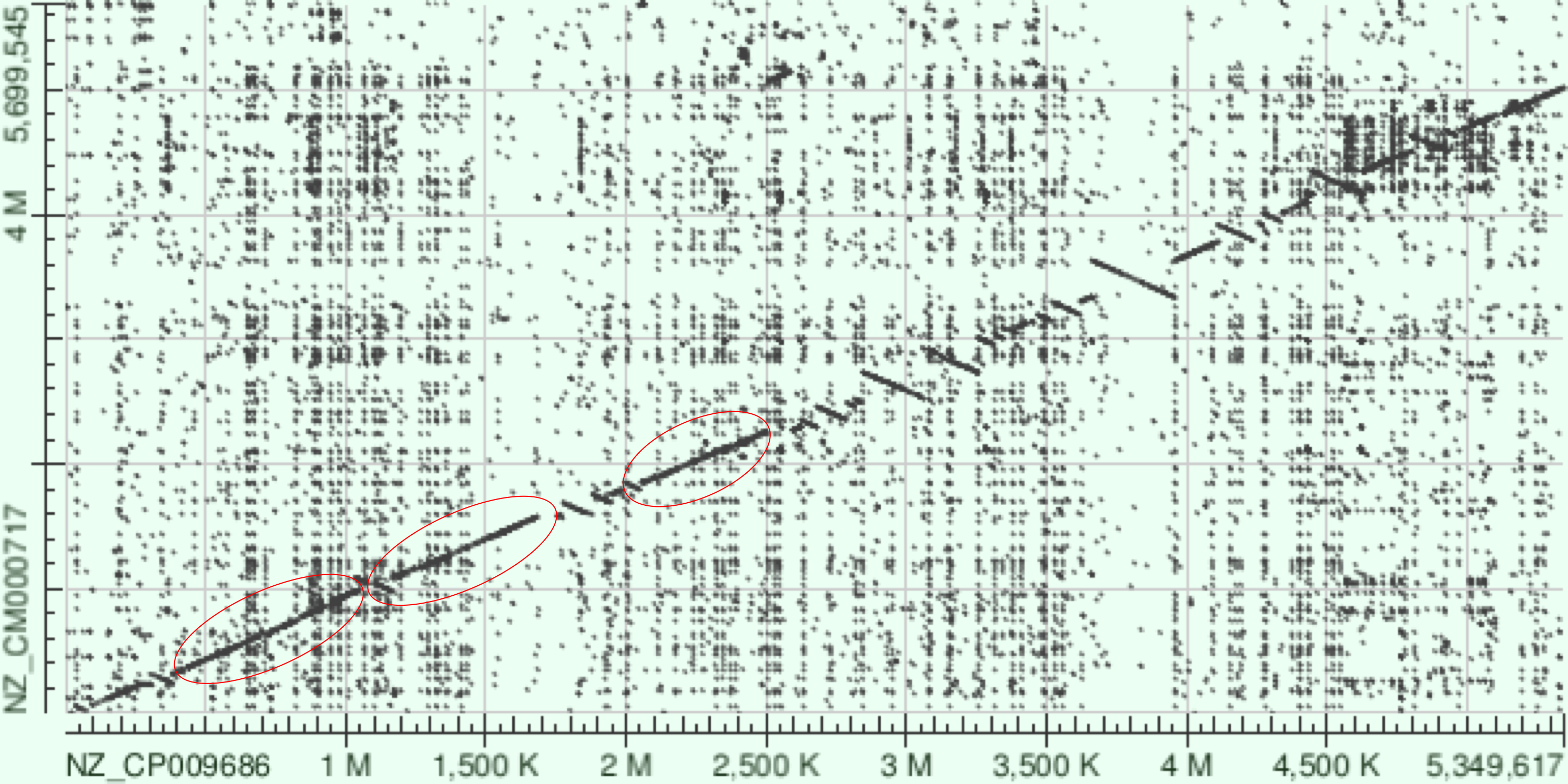
Если сравнить карты на рисунках 3 и 4, будет видно, что у 3-ей бактерии много инверсий в синтеничном участоке а).
На рисунке 5 приведена карта локального сходства NZ_CM000717 и NZ_CM000727 (2 и 3 бактерии). Видно, что карты локального сходства 1и 3 и 2 и 3 бактерий схожи бОльше и содержат бОльшее число инверсий, чем 1 и 2. Это подтверждает гипотезу того, что 1 и 2 бактерии близки к друг другу больше, чем к 3.