1. База данных OPM
В качестве примера белка, содержащего в трансмембраннной части α-спираль был выбран рецептор жирных кислот (Free fatty acid receptor 1, 1.1.01.02., PDB ID: 4PHU). На рисунке 1 представлено изображение этого белка. В таблице 1 представлена характеристика данного белка.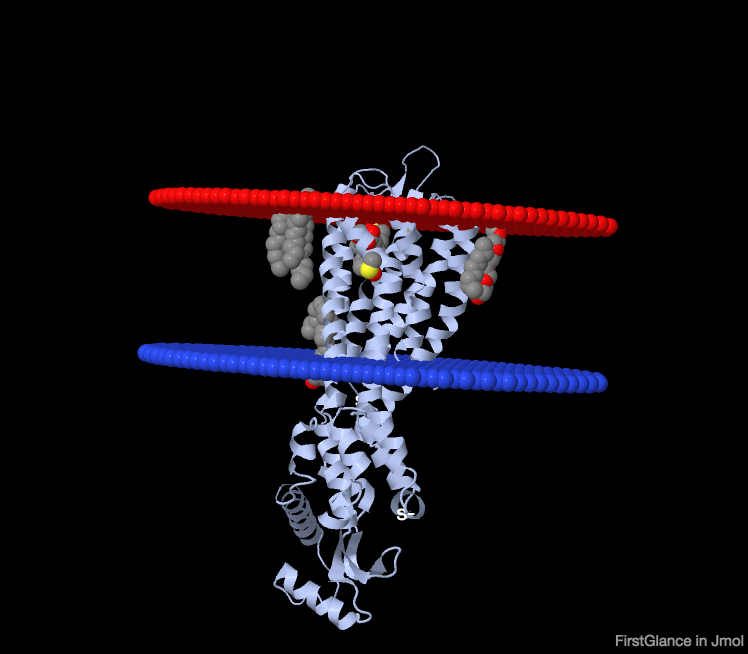
| Толщина
гидрофобной части белка в мембране |
31.8 ± 1.2 A |
| Координаты
трансмембранных спиралей |
1( 5- 28), 2(
43- 67), 3( 76- 102), 4( 123- 144), 5( 179- 204), 6(2223-2249), 7(2257-2276) |
| Среднее
количество остатков в одной трансмембранной спирали |
24 |
| Локализация | Трансмембранный |
| Толщина
гидрофобной части белка в мембране |
23.4 ± 1.0 A |
| Координаты
трансмембранных спиралей |
1( 205- 210),
2( 218- 225), 3( 233- 238), 4( 246- 254), 5( 261- 268), 6( 287- 296), 7( 303- 310), 8( 332- 340), 9( 344- 353), 10( 366- 376), 11( 382- 391), 12( 405- 416), 13( 423- 432), 14( 458- 467), 15( 481- 490), 16( 528- 538), 17( 548- 557), 18( 580- 587), 19( 593- 600), 20( 610- 618), 21( 624- 631), 22( 655- 664), 23( 671- 676), 24( 685- 693), 25( 700- 707), 26( 724- 731) |
| Среднее
количество остатков в одной трансмембранной спирали |
9 |
| Локализация | Трансмембранный
(внешняя мембрана грамтотрицательных бактерий) |
2. Анализ предсказания трансмембранных спиралей
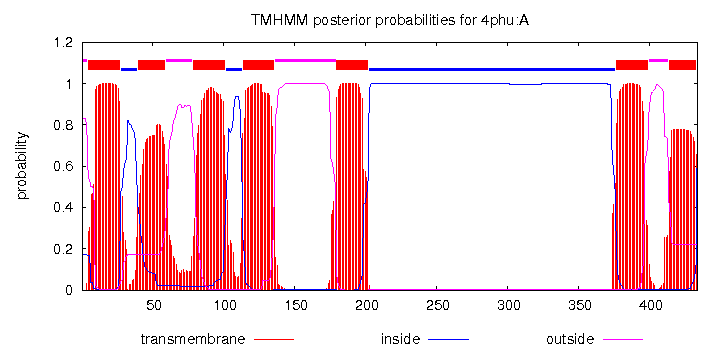
Далее был проведен сравнительный анализ точности предсказания трансмембранных спиралей у известного белка 4PHU, используемого в первом задании. Для сравнения использовались сервисы TMHMM и Phobius. На вход оба сервиса запрашивают аминокислотную последовательность белка. Текстовый и графический результаты работы программ представлены ниже.TMHMM
# 4phu:A Length: 434 # 4phu:A Number of predicted TMHs: 7 # 4phu:A Exp number of AAs in TMHs: 144.01047 # 4phu:A Exp number, first 60 AAs: 36.66187 # 4phu:A Total prob of N-in: 0.17113 # 4phu:A POSSIBLE N-term signal sequence 4phu:A TMHMM2.0 outside 1 4 4phu:A TMHMM2.0 TMhelix 5 27 4phu:A TMHMM2.0 inside 28 39 4phu:A TMHMM2.0 TMhelix 40 59 4phu:A TMHMM2.0 outside 60 78 4phu:A TMHMM2.0 TMhelix 79 101 4phu:A TMHMM2.0 inside 102 113 4phu:A TMHMM2.0 TMhelix 114 136 4phu:A TMHMM2.0 outside 137 179 4phu:A TMHMM2.0 TMhelix 180 202 4phu:A TMHMM2.0 inside 203 376 4phu:A TMHMM2.0 TMhelix 377 399 4phu:A TMHMM2.0 outside 400 413 4phu:A TMHMM2.0 TMhelix 414 433 4phu:A TMHMM2.0 inside 434 434
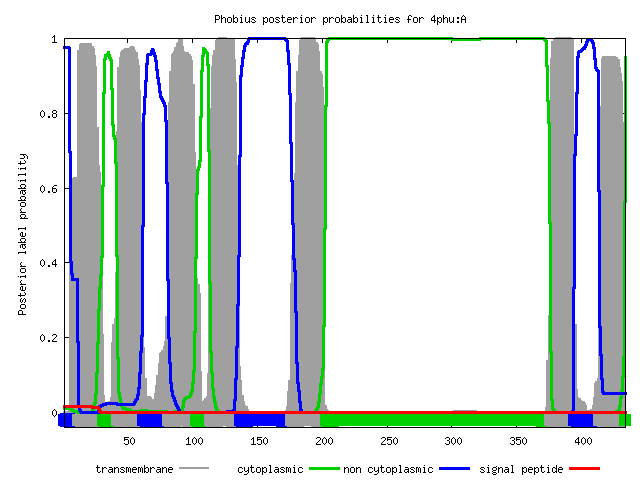
Phobius
ID 4phu:A FT TOPO_DOM 1 11 NON CYTOPLASMIC. FT TRANSMEM 12 30 FT TOPO_DOM 31 41 CYTOPLASMIC. FT TRANSMEM 42 61 FT TOPO_DOM 62 80 NON CYTOPLASMIC. FT TRANSMEM 81 102 FT TOPO_DOM 103 113 CYTOPLASMIC. FT TRANSMEM 114 136 FT TOPO_DOM 137 175 NON CYTOPLASMIC. FT TRANSMEM 176 202 FT TOPO_DOM 203 375 CYTOPLASMIC. FT TRANSMEM 376 394 FT TOPO_DOM 395 413 NON CYTOPLASMIC. FT TRANSMEM 414 433 FT TOPO_DOM 434 434 CYTOPLASMIC. //
| Номер спирали | Структур | TMHMM | Phobius | Лучшее предсказание |
| 1 | 5- 28 | 5- 27 | 12- 30 | TMHMM |
| 2 | 43- 67 | 40- 59 | 42- 61 | Phobius |
| 3 | 76- 102 | 79- 101 | 81- 102 | TMHMM |
| 4 | 123- 144 | 114- 136 | 114- 136 | - |
| 5 | 179- 204 | 180- 202 | 176- 202 | TMHMM |
| 6 | 2223-22 | 377- 399 | 376- 394 | ? |
| 7 | 2257-22 | 414- 433 | 414- 433 | ? |
Оба алгоритма включают в себя построение скрытых марковских моделей, разница состоит в том, что Phobius комбинирует алгоритм предсказания топологии трансмембранного белка (TMHMM) и предсказания сигнального белка. Путем проработки предсказания изначально выбирать одну из двух подмоделей, Phobius позволяет лучше различать, является ли начальный участок белка сигнальным пептидом или трансмембранным регионом (меньше ложноположительных результатов для трансмембранных спиралей) [3]. На рисунке 5 представлена модель алгоритма Phobius (разделенного на две подмодели для участков белка, расположенных с наружней стороны мембраны).
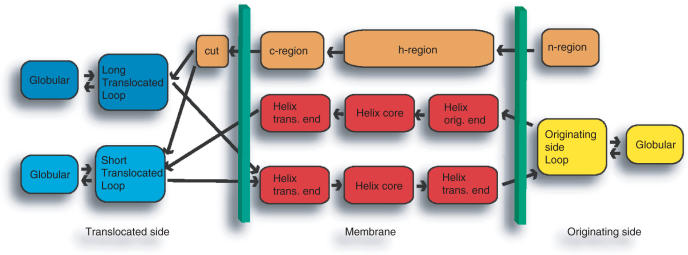
3. База данных TCDB
1. Белок 4phu относится к семейству, имееющему идентификатор в базе TCDB 9.A.14, который соответствует рецепторам, сопряженным с G-белками. Первая цифра идентификатора соответсвует классу транспортера (9 в данном случае означает недостаточно охарактеризованное семейство), второй символ - это буква, обозначающая принадлежность к подклассу транспортера, определяется тем, какой ресурс энергии используется транспортером для работы. Буква А в конкретном случае означает то, что биохимический механизм транспорта неизвестен. Все транспортеры с невыявленным механизмом транспорта относятся к этому подклассу и переносятся в другие после выяснения механихмов их работы. Следующий номер соответствует семейству (суперсемейству) транспортеров. В случае белка 4phu это семейство рецепторов, сопряженных с G-белками. Если белок хорошо охарактеризован, то его TCDB идентификатор содержит еще 2 цифры, обозначающие подсемейство и транспортируемый субстрат.Этот рецептор наиболее вероятно активируется эйкоза-3-еновой кислотой (C20:3) и обеспечивает стимуляцию инсулиновой секреции. Семейство этих рецепторов рассматривается как важный компонент, обоспечивающий регуляцию метаболизма организма (в частности, рецеция и анализ поступающей пищи), поэтому они могут быть потенциальной мишенью для лечения метаболических расстройтв, таких как диабет второго типа и ожирение [5]. В базах GO и KEGG не удалось найти этого белка.
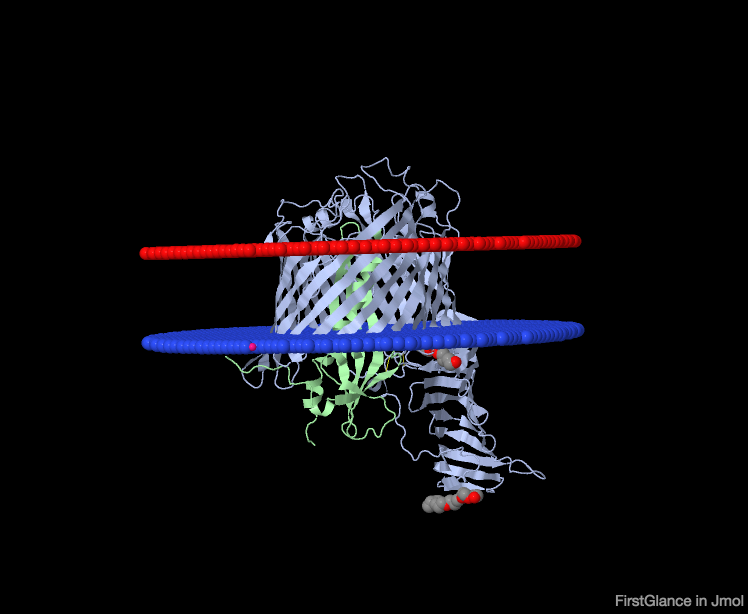
2. Белок 5IV9 относится к семейству, имееющему идентификатор в базе TCDB 1.B.42., который соответствует семейству, обеспечивающих сборку ЛПС (липополисахарид) на внешней стороне мембраны грамотрицательных бактерий. Структуные части ЛПС синтезируются по-отдельности на внутренней мембране, затем транспортируются наружу и собираются в большой полимер. Цифра 1 означает, что белок принадлежит к супер семейству каналов/пор. Буква B - то, что трансмембранный участок представлен бета-бочонком. Цифра 42 - отнесение к конкретному семейству.
4. Источники
[1] http://opm.phar.umich.edu/protein.php?pdbid=4phu
[2] http://opm.phar.umich.edu/protein.php?pdbid=5iv9
[3] ftp://ftp.sanger.ac.uk/pub/resources/theses/dogruel/chapter5.pdf
[4] Käll L, Krogh A, Sonnhammer ELL. Advantages of combined transmembrane topology and signal peptide prediction—the Phobius web server. Nucleic Acids Research. 2007;35(Web Server issue):W429-W432. doi:10.1093/nar/gkm256.
[5] https://www.ncbi.nlm.nih.gov/Structure/cdd/cddsrv.cgi?ascbin=8&maxaln=10&seltype=2&uid=cd15169