1. Выравнивание последовательностей
Для реконструкции дерева, содержащего паралоги, было предложено найти гомологи белка CLPX_ECOLI в выбранных ранее организмах . Для этого протеомы этих организмов были объединены в 1 файл. Для этого командой bash myscropt.sh был запущен скрипт ( скачать и список). Полученный файл был использован для создания базы данных ( команда: makeblastdb -in allproteoms_2.fasta -dbtype prot) и для дальнейшего поиска с помощью blastp (команда: blastp -query CLPX_ECOLI.fasta -db allproteoms_2.fasta -out gomo -outfmt 7 -evalue 0.001 -num_alignments 50). Результат поиска представлен в файле gomo. Всего было найдено 24 гомологичных белка. Для построения дальнейшего дерева были выбраны белки, длина выравнивнивания которых с референсным белком превышают половину их длины. Затем с помощью сервиса Uniprot был получен файл, содержащий аминокислотные последовательности указанных белков (список идентификаторов белков можно скачать здесь). Белковые последовательности были выровнены с помощью программы muscle (команда: muscle -in task4.fasta -out task4_aligned.fasta).
2. Построение филогенетического дерева
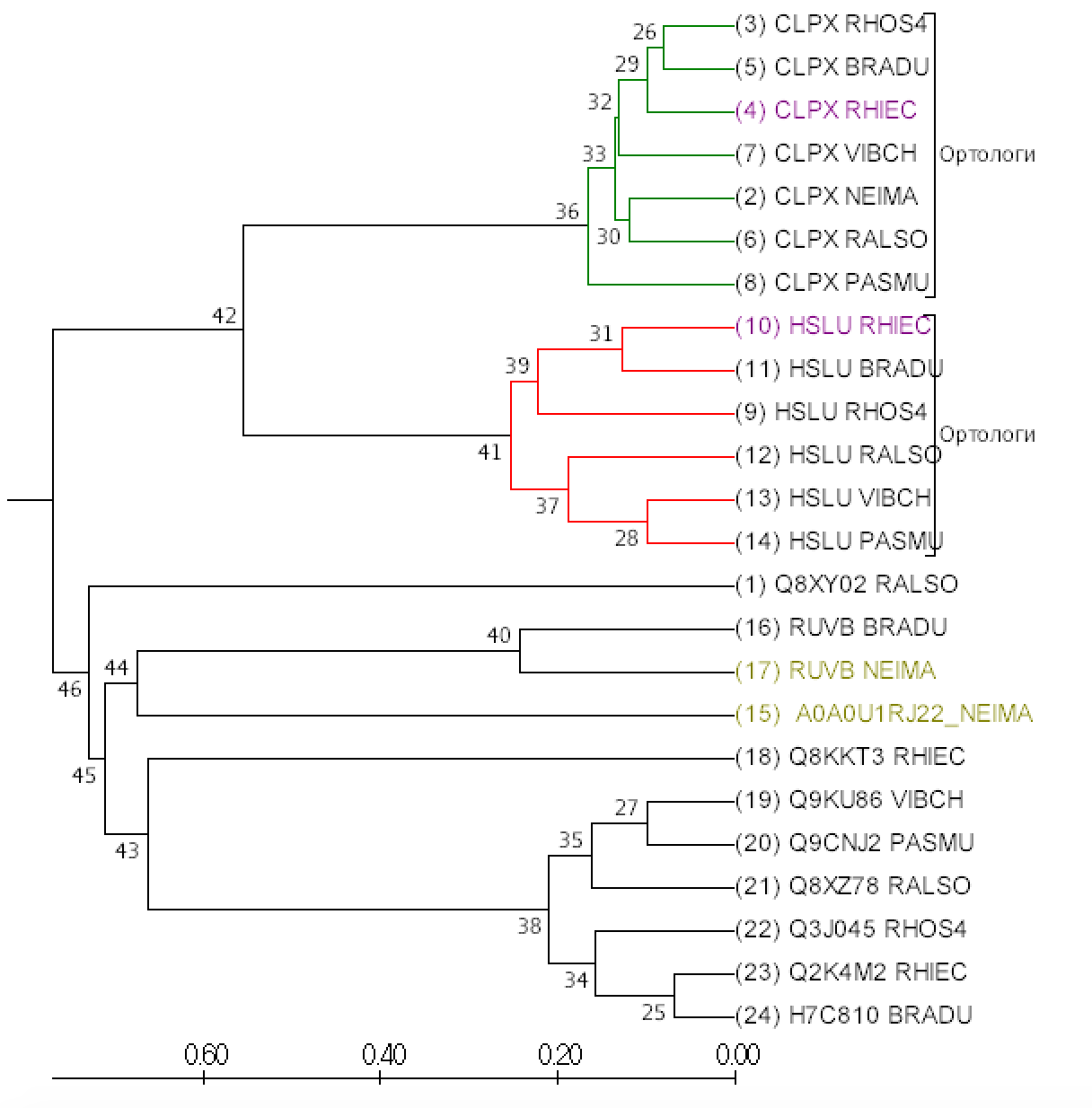
С помощью выровненных последовательностей было построено филогенетическое дерево с помощью программы Mega [1] методом UPGMA. Результат представлен на рисунке 1.
На полученном дереве можно увидеть паралоги (гомологичные белки из одного организма): белок 4 и 10 (показаны фиолетовым) и бело 17 и 15 (покрашены оливковым). Узел под номером 36 содержит ветви ортологов (гомологичные белки из разных организмов, разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования), отмечено зеленым. Эта ветвь содержит белки, имеющие АТФ-зависимые специфичные домены Clp протеаз, направляющие протеазы к специфичным субстратам. Кроме того они могут выполнять функцию шаперонов в отсутсвии Clp протеаз [2]. Узел под номером 41 также содежит ветви белков-ортологов (покрашены красным цветом). Эта ветвь содержит белки представляют собой АТФ-азную субъединицу протеасом-подобного комплекса для деградации. Эта субъединица также обладает активностью шапернов [3].
Анализируя полученное дерево, можно утверждать, что у предка организмов, представленных на этом дереве (NEIMA, RALSO, VIBCH, PASMU, RHOS4, RHIEC, BRADU), произошла дупликация гена (возмозно, в узле 42), одна его копия стала CLPX, а другая - HSLU. У NEIMA в узле 44 произошла дупликация гена RUVB, что привело к появлению белка A0A0U1RJ22 у этого же организма.
Примерами разделения путей эволюции белков в результате видообразования могут быть уже указанные ранее ветви, выходящие из узлов 41 и 36. Интересно, что восстановление филогенетических взаимоотношений между указанными видами по белку CLPX и по белку HSLU приводит к разным результатам. Например, в узле 29 RHOS4 и BRADU ближе дуг к другу, чем к RHIEC, а в узле 39 RHIEC и BRADU ближе друг к другу, чем к RHOS4.
3. Источники
[1] http://www.megasoftware.net
[2] http://www.uniprot.org/uniprot/Q9KQS7
[3] http://www.uniprot.org/uniprot/Q8Y3D8