1. Отбор данных
Я работала с организмом Escherichia coli (strain K12) (мнемоническое название: ECOLI, идентификатор таксона: 83333). По ключевому слову "Purine biosynthesis [KW-0658]" для данного штамма нашлось 17 аннотированных записей. Для дальнейшей работы были выбраны 9 белков, информация о которых представлена в таблице 1. Затем в базе данных EMBL была найдена запись полного генома данного штамма (идентификатор: AP009048.1). С помощью команд extractfeat и extractseq были получены нуклеотидные последовательности upstream элементов выбранных генов длиной 100 нуклеотидов (cсылка на скрипт). Затем полученные последовательности командой seqret @prot_list.txt upstream_ecoli.fasta были записаны в один общий файл.| Идентификатор белка | Мнемоника белка | Название белка | Название гена | Координаты |
| P0ADG7 | IMDH_ECOLI | Inosine-5'-monophosphate dehydrogenase (IMP dehydrogenase) (IMPD) (IMPDH) (EC 1.1.1.205) | guaB | complement(2631260..2632726) |
| P04079 | GUAA_ECOLI | GMP synthase [glutamine-hydrolyzing] (EC 6.3.5.2) (GMP synthetase) (GMPS) (Glutamine amidotransferase) | guaA | complement(2629614..2631191) |
| P0AB89 | PUR8_ECOLI | Adenylosuccinate lyase (ASL) (EC 4.3.2.2) (Adenylosuccinase) (ASase) | purB | complement(1192193..1193563) |
| P0ACP7 | PURR_ECOLI | HTH-type transcriptional repressor PurR (Pur regulon repressor) (Purine nucleotide synthesis repressor) | purR | 1739558..1740583 |
| P15254 | PUR4_ECOLI | Phosphoribosylformylglycinamidine synthase (FGAM synthase) (FGAMS) (EC 6.3.5.3) (Formylglycinamide ribonucleotide amidotransferase) (FGAR amidotransferase) (FGAR-AT) | purL | complement(2690312..2694199) |
| P0AG16 | PUR1_ECOLI | Amidophosphoribosyltransferase (ATase) (EC 2.4.2.14) (Glutamine phosphoribosylpyrophosphate amidotransferase) (GPATase) | purF | complement(2434167..2435684) |
| P08179 | PUR3_ECOLI | Phosphoribosylglycinamide formyltransferase (EC 2.1.2.2) (5'-phosphoribosylglycinamide transformylase) (GAR transformylase) (GART) | purN | 2620890..2621528 |
| P0A7D4 | PURA_ECOLI | Adenylosuccinate synthetase (AMPSase) (AdSS) (EC 6.3.4.4) (IMP--aspartate ligase) | purA | 4409367..4410665 |
| P33221 | PURT_ECOLI | Formate-dependent phosphoribosylglycinamide formyltransferase (5'-phosphoribosylglycinamide transformylase 2) (Formate-dependent GAR transformylase) (EC 2.1.2.-) (GAR transformylase 2) (GART 2) (Non-folate glycinamide ribonucleotide transformylase) (Phosphoribosylglycinamide formyltransferase 2) | purT | 1932595..1933773 |
2. Поиск мотивов с помощью программы МЕМЕ
Мотив - это описание сигнала (того, что узнает белок) человеком. Для поиска мотивов использовалась программа MEME [1], которая установлена на сервере kodomo (команда ememe). Для того, чтобы поиск велся на прямой и на обратной цепи ДНК нужно поставить флаг -revcomp, флаг -nmotifs позволяет задавать число различных мотивов, которые будет искать программа. На вход программе подается файл с последовательностями, в качетсве -output указывается имя директории, которая будет содержать файлы с результатами работы. Итоговая команда: ememe -dataset upstream_ecoli_.fasta -outdir motiv_ecoli -revcomp -nmotifs 3. Результат работы программы здесь. Ссылка на результаты в формате html.Мотив 1.
Регулярное выражение: A[CT]GCAAACG[GT]T[TG][AT], длина 13 нуклеотидов, E-value = 3.2e-002. Мотив найден в purL, purA, purT, purR, guaB, purN (E-value порядка e-07).
Мотив 2.
Регулярное выражение [GT][CA]A[AT][CA]A[GA][AG][TG][GT][CTA]T[TA]T, длина 14 нуклеотидов, E-value = 1.4e+001. Мотив найден в purL, purN , purF, purB, guaB, guaA, purA, purT, purR (E-value порядка e-06).
Мотив 3.
Регулярное выражение [CG][TA]GG[TG]C[GA][AG]G[ACG][GT][AC][TC]TT длина 15 нуклеотидов, E-value = 2.0e+003. Мотив найден в purN, guaB, guaA, purF, purB, purL (E-value порядка e-06).
Таким образом мотивом, который можно назвать "достоверно хорошим" не нашлось. Для поиска "хорошего мотива" я проведа аналогичные операции для всех 17 находок uniprot, Ссылка на excel файл с описанием последовательностей. Ссылка на результат работы MEME.
По итогам этого поиска был найден 1 хорший мотив. Регулярное выражение [AT]CGCAA[AT]CG[GT]T[TG][ATG]C[GTC]TT, длина 17 нуклеотидов, E-value = 4.8e-005. Было найдено 8 сайтов в purT, purM, purL, purE, purR, guaB, purA, purN . На рисунке 1 показана топология расположения домена по находкам. Видно, что в мотив расположен на разном расстоянии от старт - кодона у разных генов.
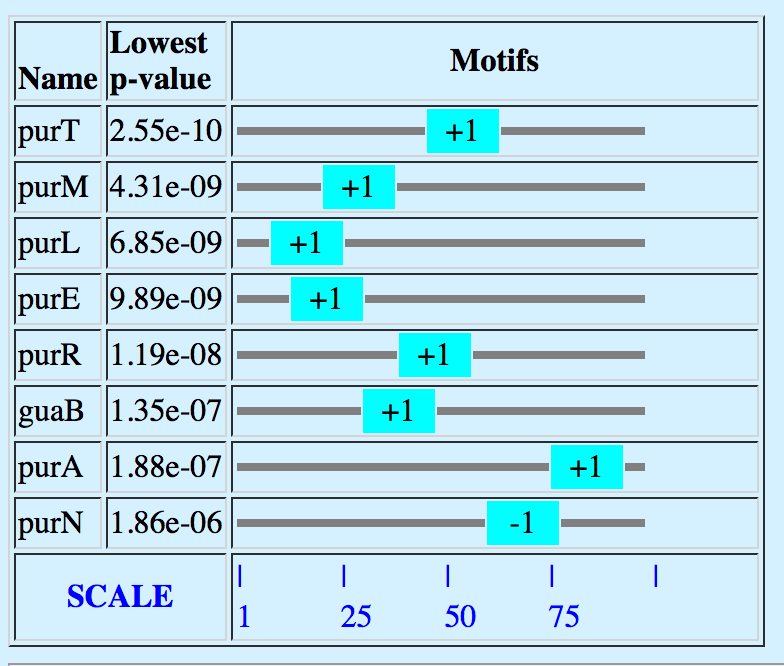
3. Сравнение найденного мотива с известным сайтом связывания пуринового репрессора
На рисунке 2 представлены лого лучшего мотива, полученного в предыдущем пункте (выше) и лого сайта связывания пуринового репрессора E.coli (ниже) [2]. По оси Х представлены координаты нуклеотида, а по оси Y - его информационное содержание в битах. Как видно из рисунка позиции 1, (3-12), 14 совпадают, однако на лого из статьи меньше стандартное отклонение значений информационного содержания. Полученная информация позволяет сделать вывод о том, что гены, в upstream области которых находится данный мотив, могут регулироваться пуриновым репрессором. И действительно, 7 из 8 находок отвечают за синтез пуринов в организме de novo, а продукт ген guaB участвует в синтезе конкретно гуанина (который является пурином).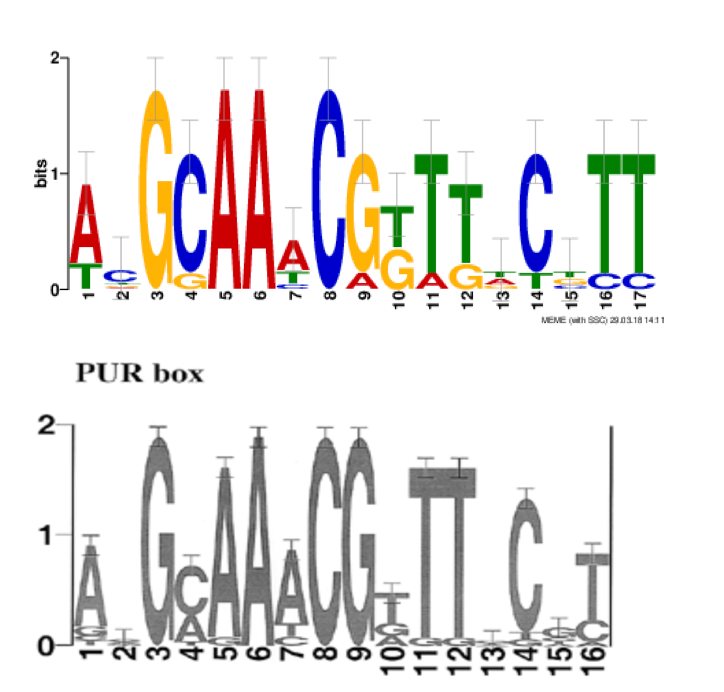
4. Поиск найденного мотива в геноме с помощью программы mast
C помощью программы mast был проведен поиск лучшего мотива в геноме e_coli. (команда mast motiv_ecoli_2/meme.txt), в качестве базы данных был указан фаста-файл, содержащий геном e_coli (AP009048.1). Результат работы программы. Так как программа не выдала точных координат находок в геноме проаннотировать последовательности не удалось.4. Источники
[1] http://meme-suite.org[2] Mironov A.A. и др. Computer analysis of transcription regulatory patterns in completely sequenced bacterial genomes // Nucleic Acids Res. 1999. Т. 27. № 14. С. 2981–2989.