1. PSI-BLAST
Для выполнения данного задания был выбран белок B2V8C0.1 экстремофильной бактерии Sulfurihydrogenibium sp. (strain YO3AOP1). Возможной функцией этого белка является определение местоположения септы (MinC). Этот белок ингибирует образование Z - кольца дестабилизируя FtsZ филаменты, полимеризация которых необходима для созревания кольца. MinC и другие белки этого семейства расположены на полюсах клетки, поэтому Z-кольцо созревает только посередине клетки (рис. 1).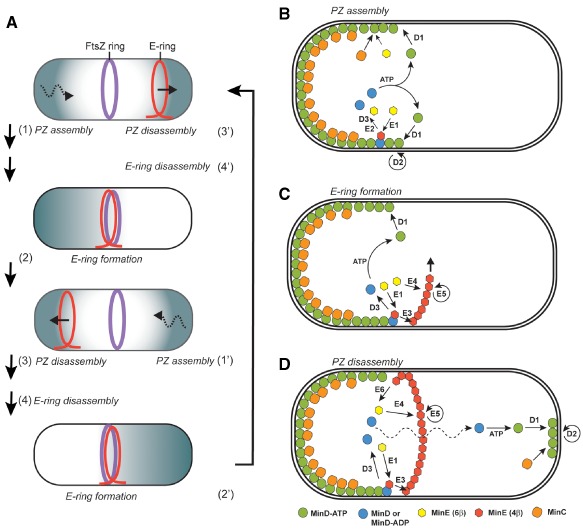
Затем был проведен поиск сервисом PSI-BLAST (Position-Specific Iterated BLAST) в базе Swiss-Prot. Список итераций приведен в таблице 1. Результат удалось стабилизировать уже после второй итерации. Как видно из таблицы, количество находок и идентификаторы лучшей и худшей находок совпадают.
| Номер итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value этой находки | Идентификатор лучшей находки ниже порога | E-value этой находки |
| 1 | 163 | Q88M41.2 | 0,004 | Q4US07.1 | 0,006 |
| 2 | 188 | Q9ZM51.1 | 6,00E-07 | A7H8E6.1 | 0,036 |
| 3 | 188 | Q9ZM51.1 | 2,00E-11 | A7H8E6.1 | 0,025 |
2. Мотивы и паттерны
Для поиска паттернов в семействе белков TIG я нашла мотив белка P0A850 (TIG_ECOLI) в банке Prosite [2]. Для этого белка нашелся 1 паттерн длиной 89 нуклеотидов, местоположение 161 - 249, последовательность:EDRVTIDFTGSVD-GEEFEGGKASD--FVLAMGQGRMIPGFEDGIKGHKAGEEFTIDVTF PEEYHAENLKgkaAKFAINLKKVEERELPELT. |
Этот паттерн предположительно является FKBP пептидил-пролил цис/транс изомеразным доменом (PPIase).
Затем этот паттерн был найден в выравнивании белков (ссылка на выравнивание получено программой muscle) и отредактирован.
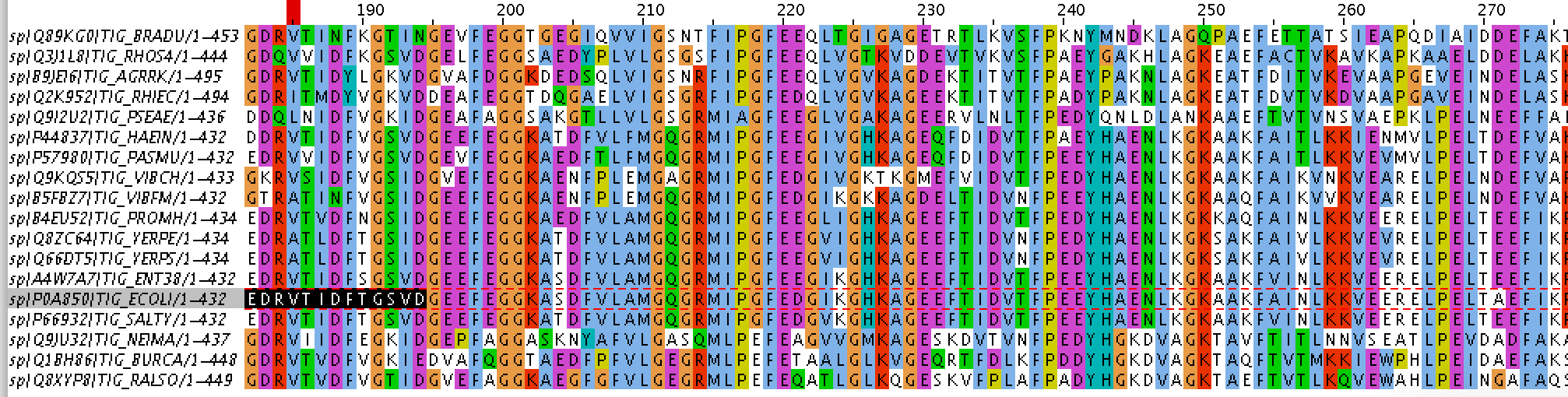
1)[EGD]-[DKT]-[RQ]-[VILA]-[TVI]-[IMV]-[DN]-[FY]-x-G[KTS]-[VI]-[DNE]-[GD]-[EV]-[EA]-F-[EAD]-GG-x-[AD]-x-[DN]-F-V-L-[AEV]-[MIL]-G-[QS]-[GN]-[RTS]-[MF]-I-P-G-F-E-[DEAT]-[QAG]-I-[KVTL]-G-[HML]-[KG]-A-[GD]
2)[EGD]-[DKT]-[RQ]-[VILA]-[TVI]-[IMV]-[DN]-[FY]-x-G[KTS]-[VI]-[DNE]-[GD]-[EV]-[EA]-F-[EAD]-GG-x-[AD]-x-[DN]-F-V-L-[AEV]-[MIL]-G-[QS]-[GN]-[RTS]-[MF]-I-P-G-F-E
3)[EGD]-[DKT]-[RQ]-[VILA]-[TVI]-[IMV]-[DN]-[FY]-x-G[KTS]-[VI]-[DNE]-[GD]-[EV]-[EA]-F-[EAD]-GG-x-[AD]-x-[DN]-F-V-L-x(2)-G-[QS]-[GN]-[RTS]-[MF]-I-P-G-F-E-x(2)
4)G-[KTS]-[VI]-[DNE]-[GD]-[EV]-[EA]-F-[EAD]-G-G-x-[AD]-x-[DN]-F-V-L
Для каждого паттерна было посчитано число истинных находок (True positives, TP), то есть размер пересечения списков, число ложных находок (False positives, FP), то есть число тех белков, которые нашлись паттерном, но не входят в правильный список, и число ненайденных (False negatives, FN). В качестве рефересного списка использовались идентификаторы аннотированных белков семейства Proteobacteria (TIG_*). Результаты представлены в таблице 2, ссылка на excel файл с находками. Как видно из таблицы все находки являются истинными, ложных не найдено, однако довольно много ненайденных находок, причем с уменьшением длины паттерна их длина уменьшается. Можно сделать вывод, что данный паттерн чувствительный, но не специфичный.
| TP | FP | FN | |
| паттерн 1 | 34 | 0 | 381 |
| паттерн 2 | 52 | 0 | 363 |
| паттерн 3 | 57 | 0 | 358 |
| паттерн 4 | 67 | 0 | 358 |
3. Psiblast командная строка
1) Input query options
-query
-query_loc
2) Общие настройки поиска
-db
-out
-evalue
-word_size
-gapopen
-gapextend
-matrix
-threshold
-outfmt - формат выдачи, самый популярный -7, табулированная таблица с шапкой.
-num_alignments
Кроме того с помощью флага -html можно получить выдачу в html формате
-num_iteration задает количество итераций
-phi_pattern - имя входного файла, содержащего паттерн для поиска
3) Построение матрицы PSSM
-in_msa
-msa_master_idx
-ignore_msa_master Игнорирование главной последовательности для создания PSSM
-in_pssm
-pseudocount
Команда для запуска: psiblast -query 1.fasta -db swissprot -remote -evalue 0.005 -pseudocount 1
Выдает ошибку "Critical: Secure socket layer (GNUTLS) has not been properly initialized in the NCBI toolkit. Have you forgotten to call SOCK_SetupSSL()?"
4. Источники
[1] Shih Y.-L., Zheng M. Spatial control of the cell division site by the Min system in Escherichia coli // Environ. Microbiol. 2013. Т. 15. № 12. С. 3229–3239.
[2] https://prosite.expasy.org/cgi-bin/prosite/ScanView.cgi?scanfile=9555154161605.scan.gz