1. Выбор семейства доменов
Для выполнения данного задания было выброно семейство доменов PF01582, Toll-Interleukin receptor (TIR). Это семейство представлено в 6950 последовательностях белков. Этот внутриклеточный сигнальный домен найден в белках MyD88, интерелейкине-1, Толл-подобных рецепторах (TLR). Он содержит 3 высококонсервативных региона, которые опосредуют белок-белковое взаимодействие между Толл-подобным рецептором и сигнальным компонентом. Кроме того он найден в некоторых растительных белках и предполагается, что он вовлечен в механизмы устойчивости к забоелваниям. После активации на TLR, TIR превлекает цитоплазматический адапторный белок MyD88 (UniProt Q99836), который запускает различные сигнальные каскады (в том числе и MAP киназные) [1].Выравнивание доменов этого семейства было скачено с pfam, визуализировано с помощью программы JalView. Кроме того была добавлена 3D структура белка MYD88_HUMAN (PDB ID: 2js7). Все выравнивание раскрашено ClustalX, порог консервативности - 10. Cсылка на проект.
2. Выбор архитектур
Ссылка на список доменных архитектур, содержащих домены выбранного семейства. Для получение списка доменных архитектур был запущен скрипт python swisspfam_to_xls.py -i /srv/databases/pfam/swisspfam.gz -z -p PF01582 -o task2Дл дальнейшей работы были выбраны две архитектуры: PF00047.24 Immunoglobulin domainи PF00560.32 Leucine Rich Repeat. На рисунке 1 представлены схемы выбранных архитектур. Лейцин-богатый домен образует гидрофобное ядро и участвует в белок-белковых взаимодействиях. Иммуноглобулиновый дмен содержит двухслойный сэндвич из 7-9 антипараллельных бета-слоев, организованных в Греческий ключ [2].

Результат работы: скачать. Лист all data содержит информацию о всех белках, содержащих домен TIR, на листе selected есть информация о белках, содержащих выбранные архитектуры с данными об их таксономической пренадлежности (434 штуки).
3. Выбор таксонов и представителей архитектур
В качестве таксона были выбраны Vertebrata, 2 подтаксона: Archelosauria (клада, объединяющая черепах, крокодилов и птиц) и Mammalia (Млекопитающие). Буквенные коды соответственно: А и M. Затем были отобраны прдествители, примерно по 30 для каждой архитектуры и таксона ( в поле selection на листе selected architecture напротив отобранных последовательностей стоит +. Кроме того к этому списку добавлен белок TLR6_HUMAN, так как для него известна 3 D структура). python filter-alignment.py -i big_align.fasta -m to_align_new.txt -o small_align_new -a "/"Полученное выравнивание было обработано вручную с помощью программы JalView:
- удалены пустые и неинформативные столбцы
- удалены гэпы на месте спиралей и тяжей, составляющих ядро домена
- удалены N и С концы последовательностей
- удалены последовательности, которые плохо выравнивались (программа могла найти в них домен по ошибке)
- выравнивание разбито на две группы по архитектуре, покрашены ClustalX. Скачать проект можно здесь.
4. Построение филогении
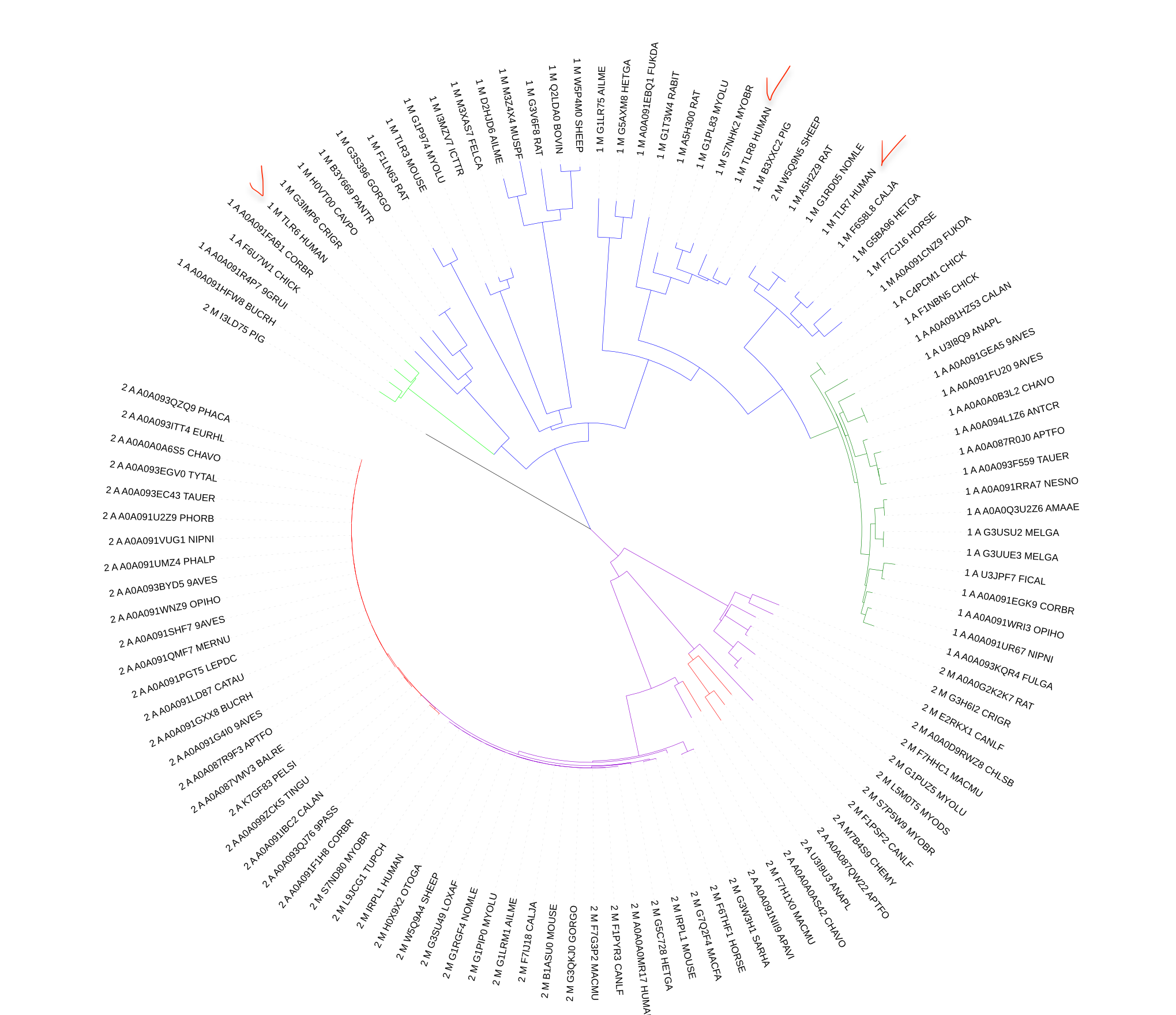
Для построения филогении были закодированы доменные архитектуры (1- Leucine Rich Repeat, 2- Immunoglobulin) и подтаксоны (Archelosauria - A, Mammalia - М). Для переименовывания последовательностей был написан скрипт на питоне (ссылка на скрипт, список идентификаторов млекопитающих, список идентификаторов лейциновых доменов). Полученный файл с переименнованными идентификаторами - ссылка.Итоговый файл был обработан программой Mega. Филогенетическое дерево было построено методом минимальной эволюции (ME). Для оценки достоверности ветвей был применен метод бутстрэп реплик (100 штук). На рисунке 2 представлено дерево , в котором толщина веток пропорциональная бутстрэп-поддержке.

Интересно заметить, что на дереве есть 3 белка (TLR6, 7 и 8 человеческие, белки, принадлежащие к семейству толл-подобных рецепторов, отмечены красным галочками), которые не образуют монофилетическую группу (как я предполагала), а образуют кластеры с другими организмами. Затем я решила подробнее рассмотреть ветвь, в которую входит TLR7 и оказалось, что белки других млекопитающх, входящие в эту кладу, относятся к TLR7 (просто почему-то называются не "говорящими именами", а набором цифр). Поэтому я решила узнать, что за белки входят в эти клады. Большая зеленая ветка, относящаяся к пресмыкающимся (и птицам), расположенная рядом с TLR7_HUMAN содержит в себе белки TLR7, что позволяет сделать вывод о том, что ген TLR7 был у их общего предка и наследовалася без сильных изменений и млекопитающим и пресмыкающимся. То, что домены TIR TLR7_HUMAN и TLR8_HUMAN ближе друг к другу, чем к TLR6, позволяет сделать предположение, что они проихошли в результате дупликации 1 гена у предка.
При более подробном рассмотрении маленькой клады зеленого цвета, соседствующей с кладой, содержащей TLR6 человеческий, выяснилось, что эти белки пресмыкающихся также относятся к TLR, но все к разным номерам (куриный -15 (предсказан, не охарактеризован), кобры - 2, 2 у птицы 9GRUI и 2 у птицы Малайский калао (BUCRH)). То есть можно сделать вывод о том, что TLR2 пресмыкающихся близок к TLR6 млекопитающих. Возможно, эти белки образовались в результате дупликации гена у древнего позвоночного.
При обращении к архитектуре, содержащей иммуноглобулиновый домен (фиолетовые и красные ветви), можно также наблюдать дупликации генов (большая ветвь, содержащая красную и фиолетовые ветки). Кроме того, инетересна ветка, содержащая 3 красных ветви интерлекиновых рецепторов пресмыкающихся и 1 фиолетовую веть неохарактеризованного белка собаки. Возможно здесь мы также наблюдаем дупликацию генов. Можно заметить, что гораздо более разнообразна эволюция доменов у млекопитающих. Я думаю это связано с тем, что гораздо больше проаннотированных и отсеквенированных геномов млекопитающих, чем птиц и черепах, находится в базах даннных, поэтому случайная выборка охватывает бОльшее разнообразие белков. Кроме того, эволюция домена TIR ассоциирована с эволюцией иммунной системы, которая гораздо больше развита у млекопитающих и содержит более разнообразный спектр различных белков. Ссылка на файл, содержащий скобочную формулу дерева. Рисунки деревьев отредактированы с помощью сервера ITOL.
5. Источники
[1] http://pfam.xfam.org/family/PF01582
[2] http://pfam.xfam.org/family/PF00047#cite_note-pmid7932691-1