Выбор объекта
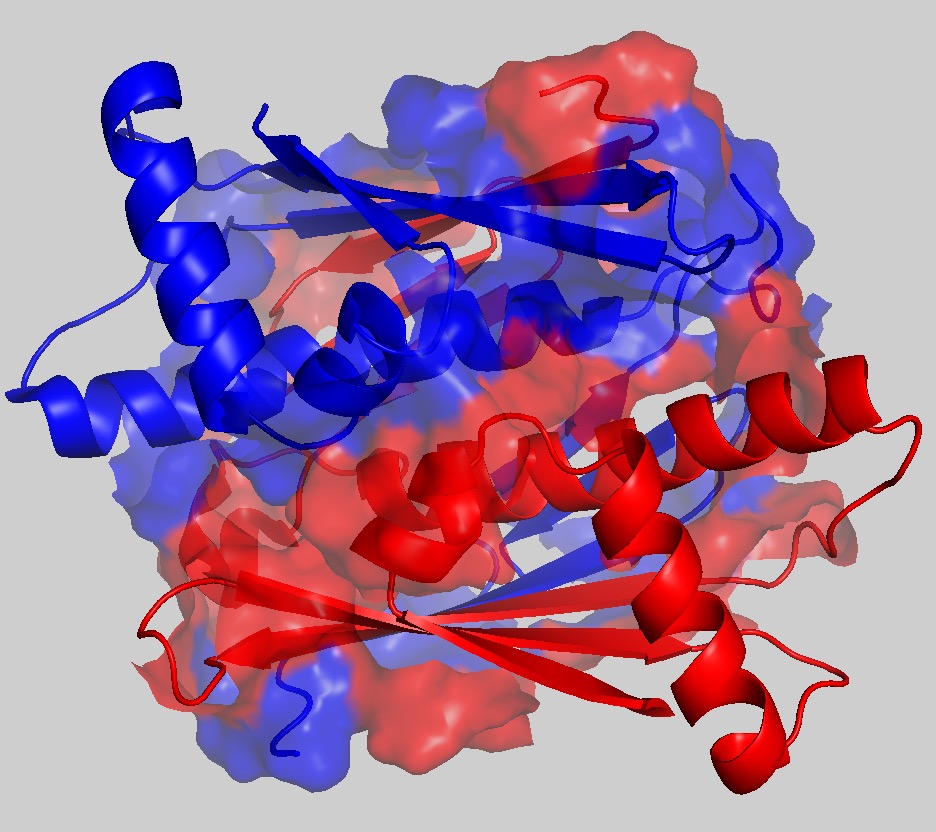
Для выполнения задания была выбрана структура с PDB ID 6uhw. Она получена методом ЯМР-анализа, содержит 20 моделей и 2 цепи. На рисунке 1 представлено изображение молекулярной поверхности для полной структуры биомолекулы на фоне cartoon модели биомолекулы.
Расчет поверхностей
Суммарная модель была разделена на 20 моделей, для каждой из которых была определена молекулярная поверхность (MS) и поверхность, доступная для растворителя (SAS). Для этого в PyMOL были выполнены следующие команды:set all_states, on
split_states 6uhw
set dot_solvent, off #MS
get_area 6uhw_000i #(i = [1, 20])
set dot_solvent, on #SAS
get_area 6uhw_000i #(i = [1, 20])
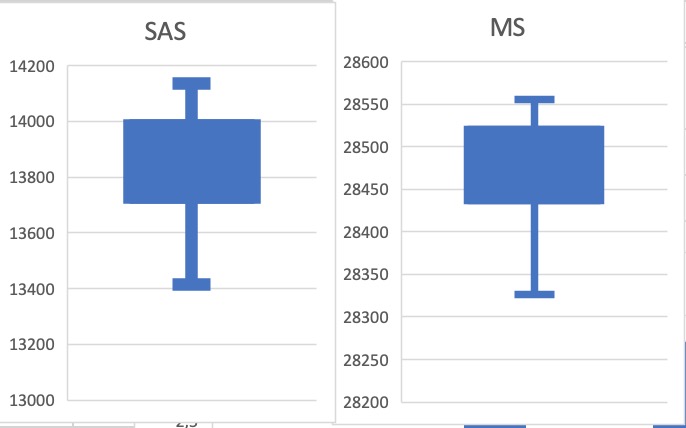
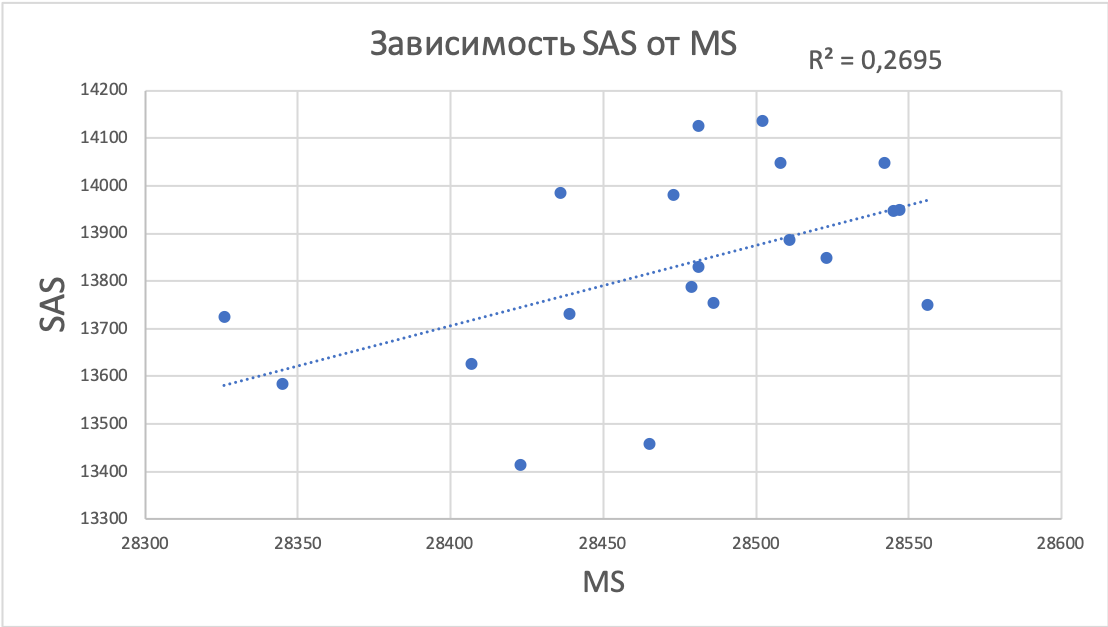
Полученные результаты представлены в файле. Получено, что распределение MS более "компактное", чем SAS (Рис.2), которое имеет бОльшую дисперсию. MS больше SAS примерно в два раза. Также было получено, что эти параметры не зависят линейно друг от друга (Рис.3).
Построение поверхности контактов
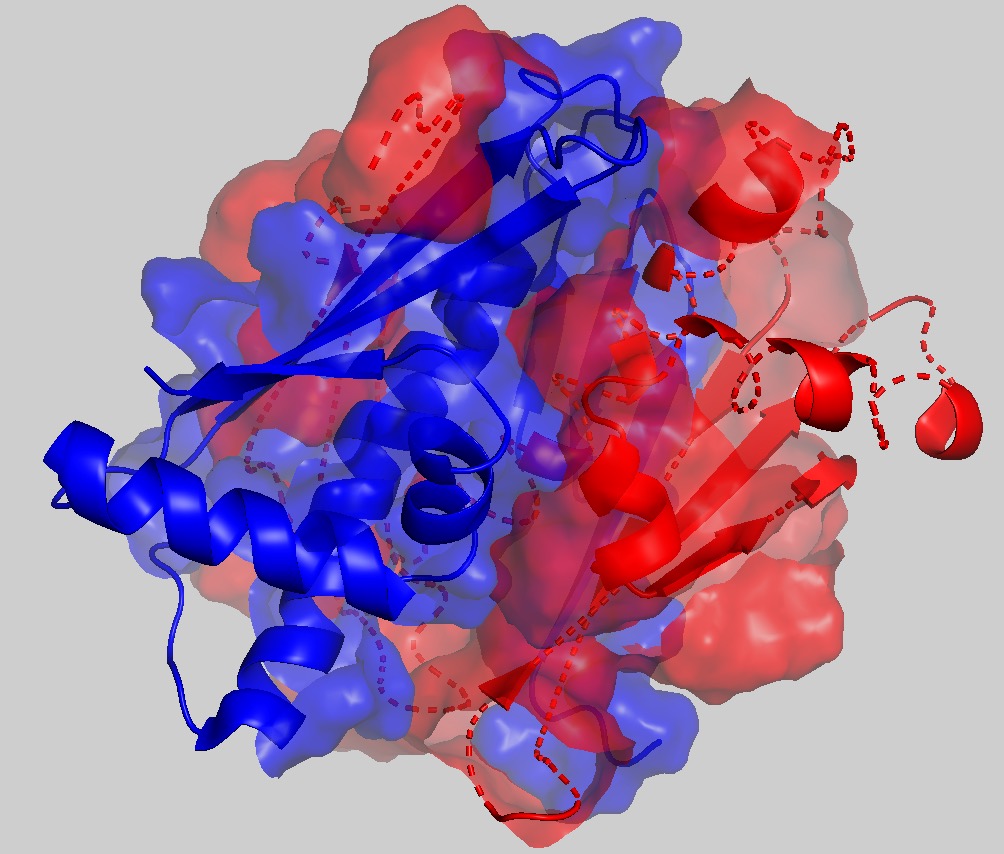
Далее была построена поверхность контактов двух цепей (в пределах 3,5 ангстрем). Результат представлен на рисунке 4. Интересно, что поверхность контакта представлена в основном контактами альфа-спиралей. Затем была получена поверхность контактов для цепей, разделенных командой extract (рис.5). Видно, что поверхности во втором случае разделены "четче", в то время как в первом цвета как бы "перетекают" один в другой.