Выравнивание последовательностей
Глобальное парное выравнивание гомологичных белков
Для выполнения задания я взял список идентификаторов Swiss-Prot с заданной мнемоникой организма (*_ecoli и *_bacsu) на сайте Uniprot.org.
Далее я применил глобальное выравнивание для мнемоничных белков.
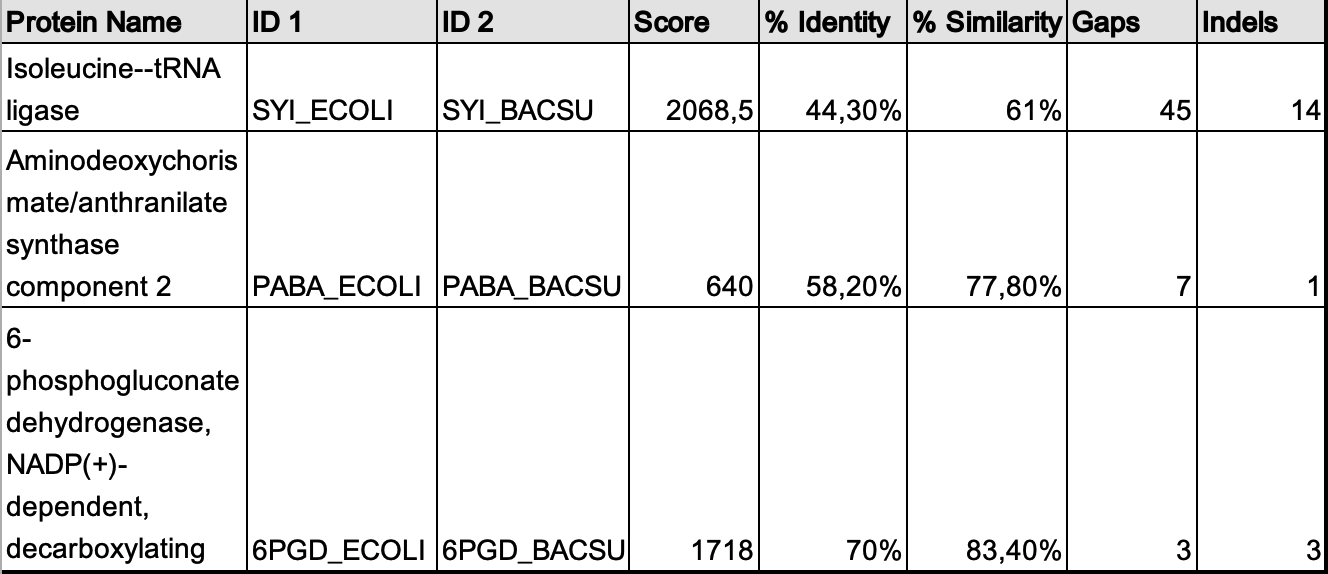
Команда для глобального парного выравнивания:
needle sw:*_ecoli sw:*_bacsu *.needle -auto
Локальное парное выравнивание гомологичных белков
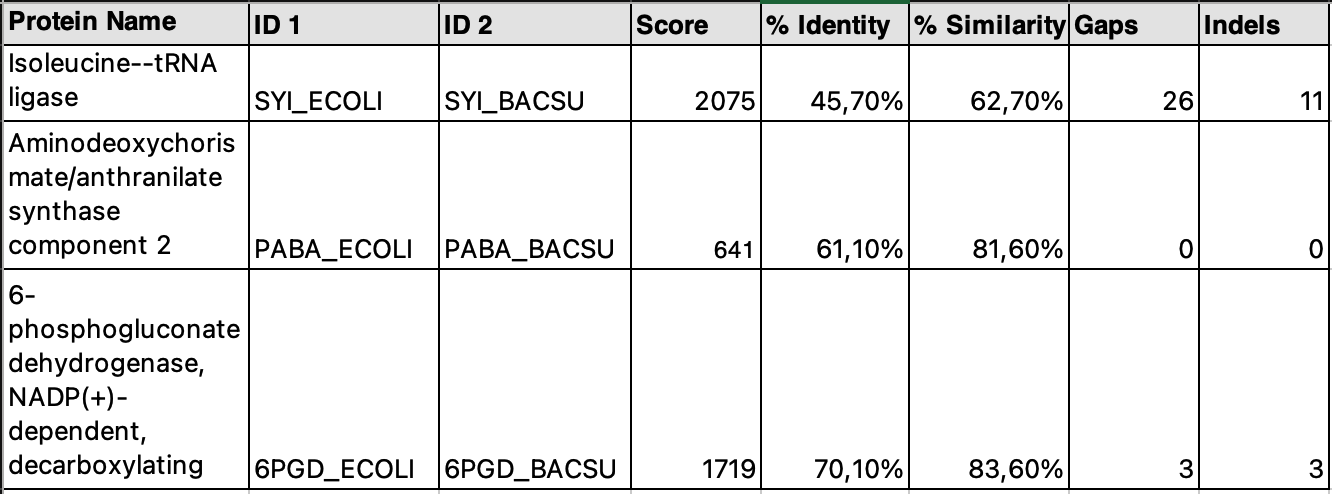
Команда для локального парного выравнивания:
water sw:*_ecoli sw:*_bacsu *.water -auto
Isoleucine--tRNA ligase: coverage1 - 95.6%, coverage2 - 94.3%
Aminodeoxychoris mate/anthranilate synthase component 2: coverage1 - 95.4%, coverage2 - 95.4%
6-phosphogluconate dehydrogenase, NADP(+)- dependent, decarboxylating: coverage1 - 99.3%, coverage2 - 99.5%
Результат применения программ выравнивания к неродственным белкам
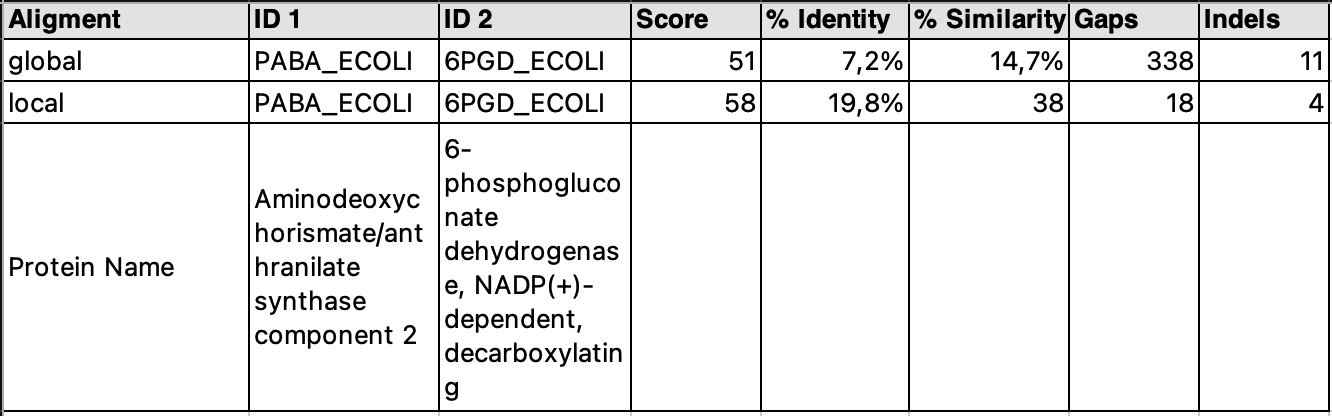
Второй ID: 6PGD_BACSU
Для выполнения этого задания я выбрал уже использовавшиеся белки.
Сравнение доказало, что эти белки не гомологичны (схожесть меньше 25%).
Показатели локального выравнивания выше 25%, но это не позволяет нам сделать вывод, что белки гомологичны. Возможно, они имеют схожие функции.
local: coverage1 - 22.9%, coverage2 - 22.1%
Множественное выравнивание белков и импорт в Jalview
Для выполнения этого задания я использовал белки с мнемоникой SYQ_* (Glutamine--tRNA ligase). Поиск осуществлялся по базе данных UniprotKB. По результатам поиска было найдено 154 белков.
Случайным образом было выбрано 5 белков:
SYQ_HUMAN - Homo sapiens (Human)
SYQ_YERPY - Yersinia pseudotuberculosis serotype O:3 (strain YPIII)
SYQ_RAT - Rattus norvegicus (Rat)
SYQ_SALEP - Salmonella enteritidis PT4 (strain P125109)
SYQ_ECOLI - Escherichia coli (strain K12).
Выравнивание было произведено в программе Jalview с использованием метода T-coffee with Defaults.
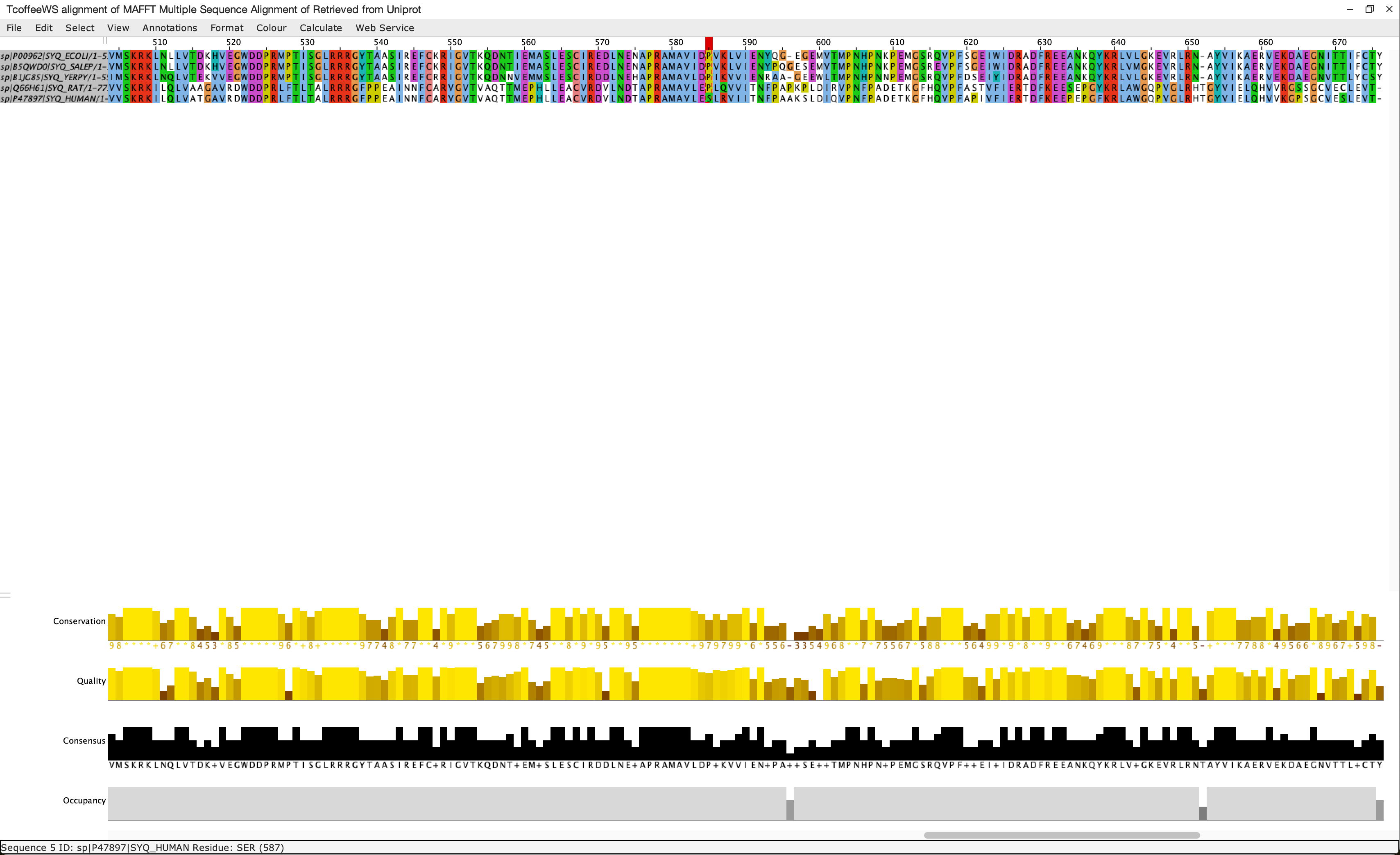
Исходя из результатов выравнивания, можно сделать вывод, что все белки гомологичны. Наиболее консервативные области находятся на интервале 260-675. Наименее консервативный участок располагается на интервале 16-24 и 44-49.
Файл выравнивания в формате Jalview