Поиск гипотетических гомологов белка PDUO_BACSU в разных банках данных
Результаты поиска гипотетических гомологов белка PDUO_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (в принципе должна соответствовать заданному белку) |
|||
| Accession | O34899 | 1RTY_A | ZP_03593108 |
| E-value | 6e-138 | 6e-136 | 2e-136 |
| Вес (в битах) | 390 | 382 | 390 |
| Процент идентичности | 100% | 99% | 100% |
2. Сколько хороших кандидатов в гомологи найдено? (число находок в списке описаний с E-value < 1e-10) |
|||
| 7 | 17 | 1707 | |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 9 | 17 | 1714Описание сравнений заканчивается на белке под номером 1000, и, поскольку он - последний, про которого есть какие-то данные, делее речь пойдет о нем |
| Accession | A4SGA5 | 1I4J_A | YP_955108.1 |
| E-value | 0.92 | 5e-28 | 1e-31 |
| Вес (в битах) | 33.5 | 105 | 122 |
| % идентичности | 43% | 40% | 40% |
| % сходства | 64% | 59% | 57% |
| Длина выравнивания | 45 | 168 | 188 |
| Координаты выравнивания в запросе | 85-129 | 1-167 | 2-182 |
| Координаты выравнивания в находке | 439-484 | 1-160 | 7-188 |
| Число гэпов | 3(6%) | 9(5%) | 13(7%) |
- В swissprot я нашел лишь 7 похожих последовательностей, в PDB - 17, Оба числа мелнькие, это объясняется жестким курированием в swissprot, и там просто не хватает лююдей на все тысячи и тысячи белков, которые можно описать, а для того чтобы добавить белок в PDB, необходимо провести его рентгеноструктурный анализ, что также сильно ограничивает число находок а вот в nr похожих белков оказалось 1714, оно и понятно, ведь это сборная солянка из многих баз данных, содержащая много повторов и белков, секвенированных у родственных видов
- во всех трех случаях число находок было ограничено числом E-value(10, по умолчанию), правда, в случае с nr лимит выдачи равнялся 10000, а белков с подходящим E-value-только 1734
Поиск гомологов из других таксонов
гипотетический гомолог (с E-value > 0.001)
используемая БД - swissprot
| Таксон | 'Eukaryota' (другое царство) |
| Accession | Q9D273.1 |
| E-value | 1e-35 |
| Вес (в битах) | 121 |
| % идентичности | 42% |
| % сходства | 53% |
| Длина выравнивания | 186 |
| Координаты выравнивания в запросе | 2-180 |
| Координаты выравнивания в находке | 51-234 |
| Число гэпов | 9(5%) |
сравнение весов выравниваний
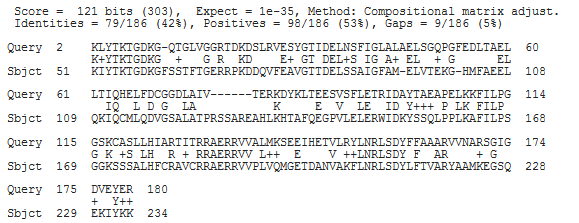
Это эвристическоеЭврестическое ли? Я ничего не напутал? выравнивание белков PDUO_BACSU и MMAB_MOUSE программой blastp, матрица весов - BLOSSUM62, штраф за открытие гэпа - 11, за продолжение - 1, вес выравнивания, как видно, 303

Это глобальное выравнивание белков PDUO_BACSU и MMAB_MOUSE программой stretcher, матрица весов - BLOSSUM62, штраф за открытие гэпа - 11, за продолжение - 1, вес выравнивания - 152
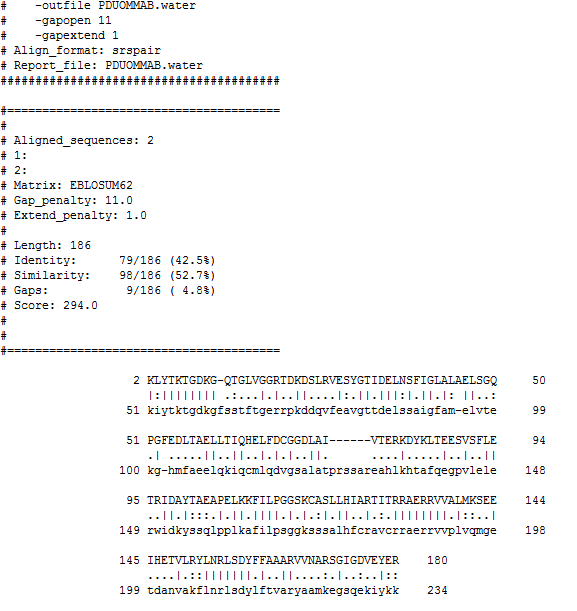
Это локальное выравнивание белков PDUO_BACSU и MMAB_MOUSE программой water, матрица весов - BLOSSUM62, штраф за открытие гэпа - 11, за продолжение - 1, вес выравнивания - 294
Локальное(программой water) и эврестическое(программой BLASTP) выравнивания в общем несильно отличаются от глобального(программой stretcher) и между собой, разве что они начинаются со второго остатка белка PDUO_BACSU, так-как в начале у его гомолога есть довольно длинная последовательность, которой нет у бактериального белка, а в конце у бактериального белка есть хвост, которого нет у мышиного белка, но в рамках тех аминокислот, которые есть в серединах обоих белков(и в частности в их активных центрах) есть только одно отличие, примерно в середине последовательности PDUO_BACSU есть длинный гэп, но в эврестическом выравнивании он располагается между 77 и 78 остатками, в локальном - между 76 и 77, а в глобальном - между 80 и 81 остатками аминокислот. Во всех остальных местах все три программы показали идентичные результаты.
| программа выравнивания | BLASTp | stretcher | water |
| Вес выравнивания | 303 | 220 | 294 |
| Процент идентичности | 79(42.5%) | 82(32.7) | 79(42.5%) |
| Процент сходства | 98(52.7%) | 102(40.6) | 98(52.7%) |
| Число гэпов | 9(5%) | 72(28.7%) | 9(5%) |
| Длина выравнивания | 186 | 251 | 186 |
| Координаты выравнивания в PDUO_BACSU | 2-180 | 1-193 | 2-180 |
| координаты выравнивания в MMAB_MOUSE | 51-234 | 1-237 | 51-234 |
Дополнительно
Поиск с другими параметрами blast
Я задал BLAST нестандартные параметры, а именно - понизил штраф за открытие гэпа до девяти, а за продление - увеличил до двух, заменил параметр word size с трех до двух и включил фильтр для борьбы с последовательностями малой сложности, в результате выбрав nr как базу данных я получил 1688 последовательностей вместо 1714, при word size 3 - 1694 последовательностей, а при отсутствии фильтра борьбы с последовательностями малой сложности - 1742
Находки с E-value > 1
Так как при E-value больше единицы вероятность того что последовательности не гомологичны становится весьма существенной, следует смотреть на другие параметры белков, а именно - на их функцию, и, при множественном выравнивании на аминокислоты в консервативных позициях, а также на то, насколько далеки в таксономическом плане белки, ведь понятно, что белки, выполняющие одну и ту же функцию у некой бактерии и у человека, даже если они гомологичны, за миллиарды лет раздельной эволюции стали до неузнаваемости разными. Если то что обсуждалось выше - неправда, можно смело утверждать, что белки негомологичны, если же это справедливо, то можно робко утверждать о гомологии.
Интерфейсы BLAST на разных платформах
Я попробовал повторить некоторые запросы из предыдущих заданий в BLAST с другими интерфейсам на сайтах EBI и ExPASy
-На сайте NCBI просто и удобно можно получить подсказки ко всем полям, на ExPASy тоже есть подсказки, но для просмотра их придется открыть новую вкладку в браузере, на EBI - же, чтобы найти хелп, мне пришлось изрядно его поискать.
-По функциональности интерфейсы на сайтах EBI и NCBI примерно равны, все на виду, все расположено удобно и логично, интерфейс же ExPASy мне показался пегруженным и разнородным, и в количестве настроек он явно уступает своим аналогам. Отдельно скажу про библиотеки, наиболее полно и широко они представлены на сайте EBI, что на мой взгляд является преимуществом перед другими интерфейсами.
-Наиболее удобным мне представляется интерфейс выдачи результатов на EBI, реализованные там кнопки show/hide annotations и show/hide alignments позволяют регулировать количество подаваемой информации по желанию, на NCBI и ExPASy интерфейсы в общем похожи, но ExPASy опять же на мой взгляд ярок и перегружен.