структуры ДНК A B и Z форм, полученные программой X3DNA
Структуры А и В форм содержат последовательность GTCA, повторенную 4 раза, в то время как Z форма состоит только из пар GC
| A-форма | B-форма | Z-форма |
Все нуклеотиды |
||
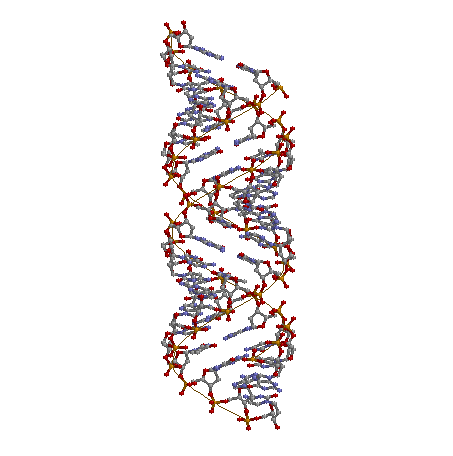 |
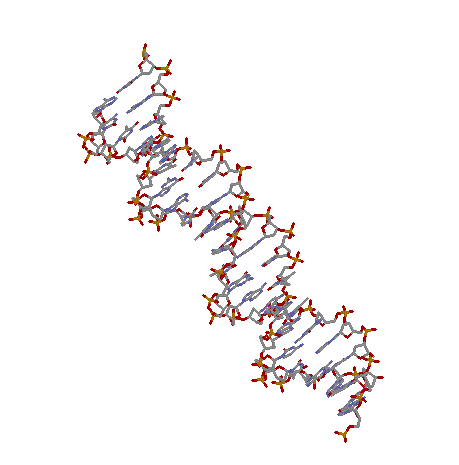 |
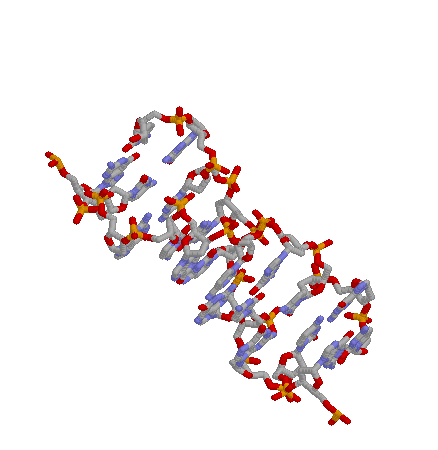 |
Остов цепей |
||
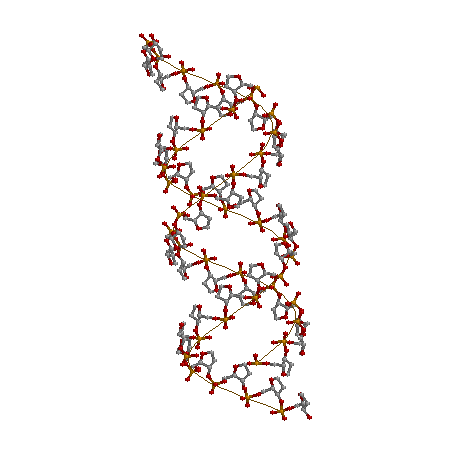 |
 |
 |
Все аденины |
||
 |
 |
 |
Гуанины с выделенными N7 атомами |
||
 |
 |
 |
Исследования на разрыв в цепях тРНК и ДНК
| комплекс ДНК-белок | комплекс тРНК-белок |
Изображения с PDB |
|
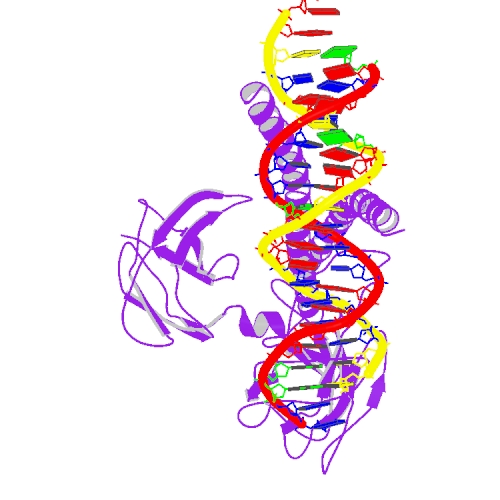 |
 |
Проволочные модели нуклеиновыз кислоткак видно, разрывов нет |
|
 |
 |
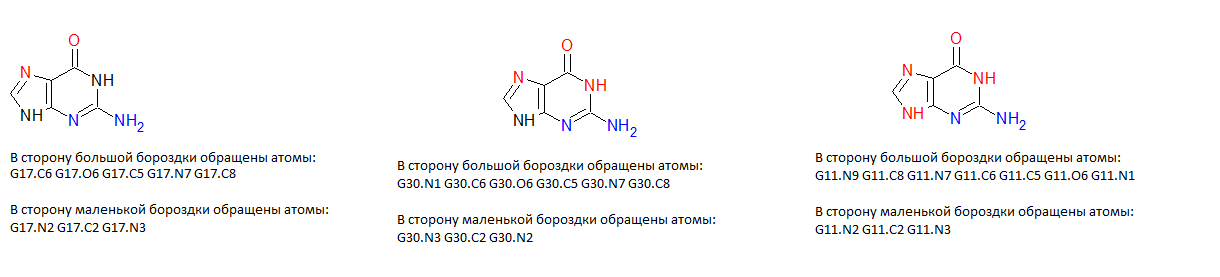
атомы гуанина, обращенные в сторону большой и малой бороздки в A B и Z формах ДНК
спиральные параметры разных форм ДНК
| A-форма | B-форма | Z-форма | |
| Тип спирали (правая или левая) | правая | правая | левая |
| Шаг спирали (Å) | 28.3 | 33.2 | 44.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Шширина большой бороздки | 16.6(от фосфата G16) | 17.4 (от фосфата G12) | 9.9 (от фосфата G10) |
| Ширина малой бороздки | 7.8 (от фосфата A6) | 12.0 (от фосфата C14) | 18.30 (от фосфата C4) |
значения торсионных углов Гуанина
| α | β | γ | δ | ε | ζ | χ | |
| А-форма (моя) | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| А-форма (презентация) | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-форма (моя) | -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| B-форма (презентация) | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Далее все приведенные данные были получены с помощью программ base pair и analyze
Торсионные углы нуклеотидов, измеренные в заданной структуре ДНК
я взял pdb файл заданной ДНК, с помощью программ find pair и analyze получил файл, в котором были отмечены все торсионные углы всех нуклеотидов,
после чего, убрав значения концевых нуклеотидов, преобразовал данные в OpenOffice Calc(аналог Excel)в таблицу в формате ods(ссылка на таблицу),
где посчитал средние значения торсионных углов
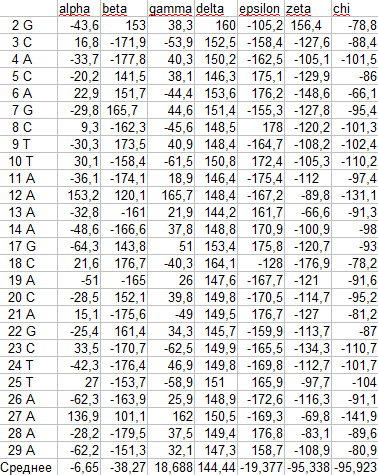
как видно из таблицы, наиболее по углу α, а также и по другим довольно сильно от других отличаются 12 и 27 Аденины,
по углу β нуклеотиды разделились на 2 группы, около 160° и около -160° выделяются опять же А12 и А2.
по углу γ опять же все нуклеотиды имеют значения около 30° или -30°, в то время как А12 и А27 - около 165°
по углам δ и ε все нуклеотиды идут довольно ровно, а по остальным нет четкого строя, из которого можно было бы выделить особо отличающиеся углы
Структура водородных связей
Акцепторный стебель образуют нуклеотиды 501-507 и комплементарные им 566-572
D-стебель 510-513 и 522-525
T-стебель 561-565 и 549-553
антикодоновый стебель 538-544 и 526-532
Всего 8 не Уотсон-Криковских пар, а именно: G502-U571, U555-G518, A538-C532, A544-G526, U513-G522, G501-C572, U555-G518, A538-C532
После удаления из рассмотрения всех водородных связей, образующих стебли, осталось довольно внушительное количество пар, и я просто выделил их в отдельный файл (ссылка на файл)
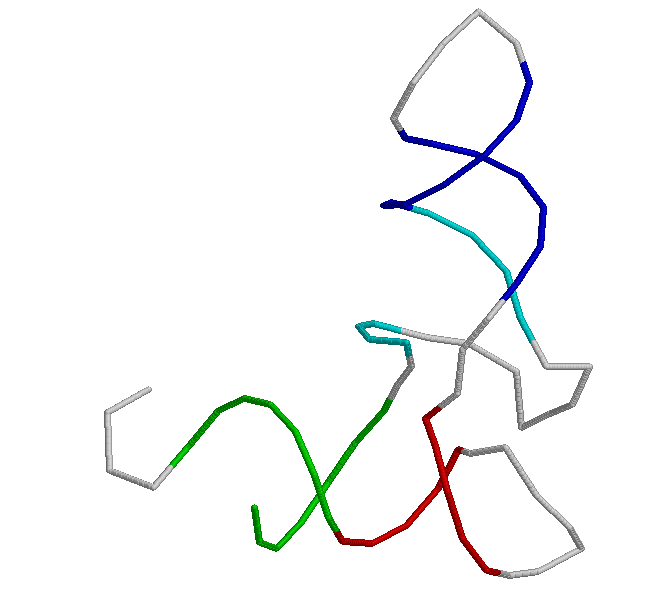
Третичная структура
На изображении - 3-х мерная структура остова тРНК, где акцепторный стебель обозначен зеленым цветом, D-стебель - циановым, Т-стебель - красным, а антикодоновый стебель - синим
Все петли - серые
Стэкинг взаимодействия
В файле 1N77.out есть данные о площади перекрывания пар нуклеотидов, самое большое - 13.40Å2, самое маленькое ненулевое - 0.13Å2
соответственно изображения
| наибольшее перекрывание | наименьшее перекрывание |
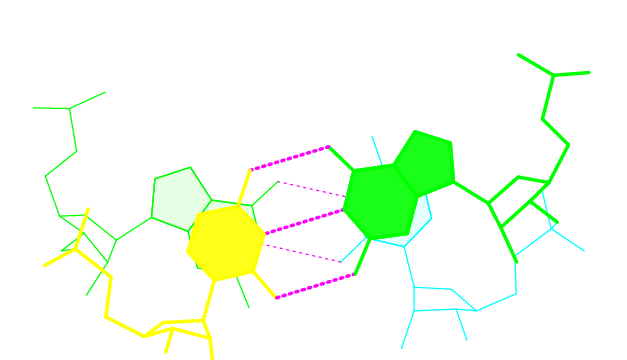 |
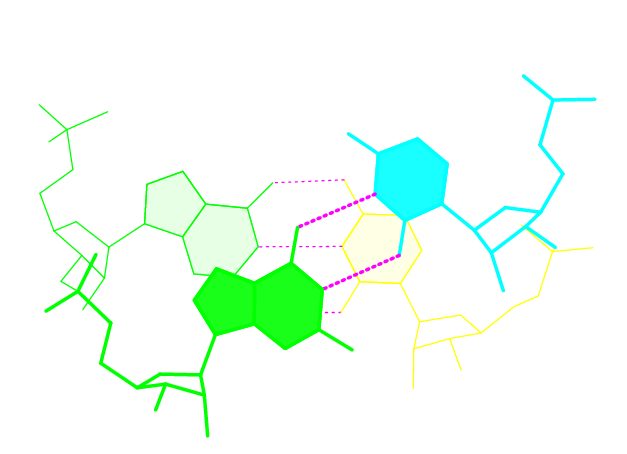 |