вступительное слово
Вообще у геномных браузеров, и у ансамбля в частности - нагруженный и тяжелый для понимания интерфейс, что лично для меня, как для новичка, оказалось большой тудностью для понимания происходящего на экране.1 задание
Сначала с помощью программы BLAST/BLAT с постым относительно других подобных сервисов я, введя данные из записи гена еmbl, получил страницу результатов, на которой оказалось 3 информативных блока: первый - положения выравниваний кариотипа (в данном случае человеческого) с выделением в рамочку самого близкого(99.48% сходства) результата к запросу
второй - визуальное отображение расположения самого близкого выравнивания на участке хромосомы, а так же выравниваний, находящихся на прилежащих участках(как видно, есть очень большое количество короткмих (примерно по 200-300bp) выравниваний, совпадающих с лучшим, видимо, это контиги, правда, почему они собрались все в одном месте - непонятно)
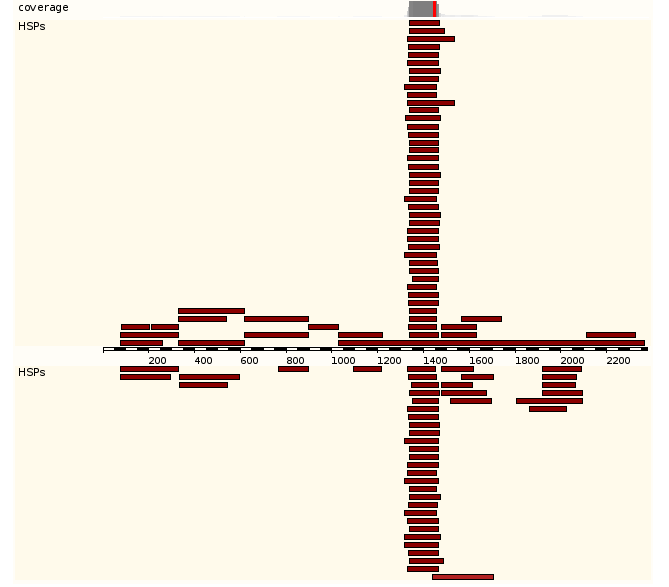
Третий - это уже знакомые результаты программы BLAT (по аналогии с BLAST), где приводятся длины, веса выравниваний, e-value, а так же информация о положении в геноме и наличии ori(вот этого я совсем не понял, ведь ori - это origin of replication, а это чисто бактериальная фича)
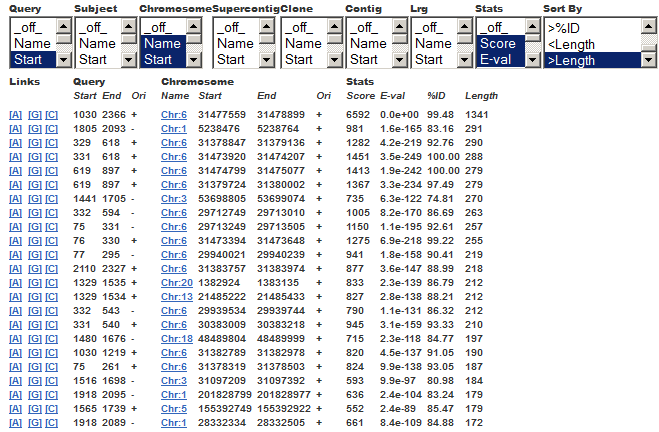
Также рядом с результатами выравнивания есть 3 ссылки - на само выравнивание, на участок в геноме и на контиги, перейдем по ссылке на контиги
контиги
По ссылке также открываются 3 информационных блока. Они очень удобны, так, можно увеличить/уменьшить размер отображаемого фрагмента хромосомы, сохранить блок в виде картинки или перейти к любому аннотированному участку с помощью двух кликов. Перый - хромосома с выделенным в рамочку участком, рассмотренным на нижних блоках
Второй - "Region in detail" - регион хромосомы, содержащий искомый запрос, в увеличенном виде с отмеченными аннотироваными участками, которые можно изучить, кликнув на них

Третий - участок хромосомы в высоком разрешении, на котором видны варианты альтернативного сплайсинга гена(если он есть на участке), непосредственно участки, выравненные программой BLAST/BLAT, а также, если рассматривается геном человека или мыши, (а рассматривается геном человека), то и строку CCDS set,(кстати, я так и не смог до конца разобраться в обозначениях CCDS) представляющую из себя исследованние данного участка различными методами и их объединенного результата и прочую информацию.
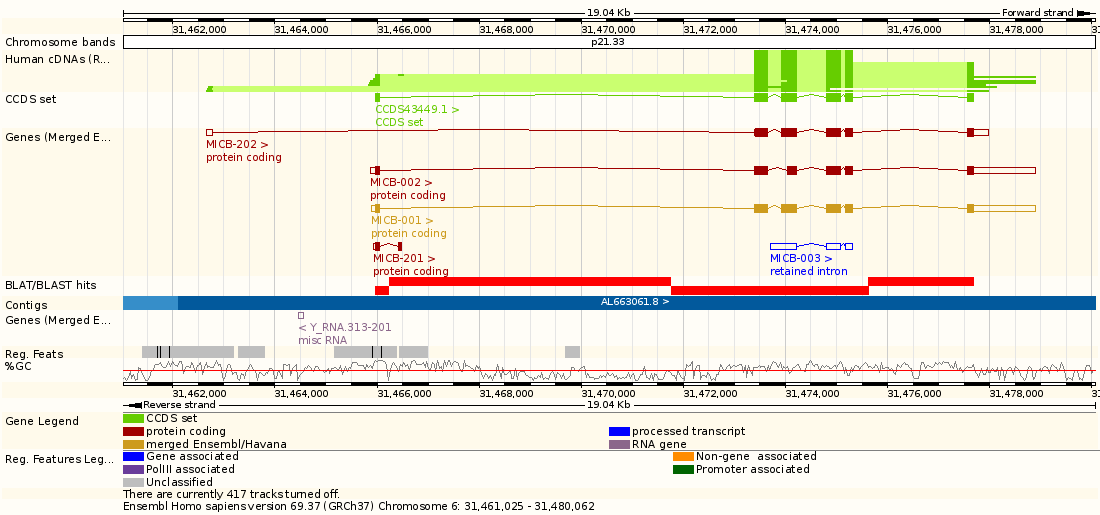
Далее посмотрим на меню справа сверху, там можно найти вариации нуклеотидного кода у разных инднивидуумов(у человека - это "типичный" для человека геном, геном Уотсона и Вентера)
Также интересно оказалось узнать источники информации, ссылки на которые есть у всех генов, так, мой белок MICB был аннотирован в рамках проекта Havana, созданного для браузера Vega, который поставил задачу вручную аннотировать и анализировать геномы позвоночныхэ и человека.
Также в этом меню есть ссылка на сравнение данного участка генома с гомологичными ему. Вот, например, выравнивание моего гена у человека и аналогичного у кошки (Felis catus)

Дополнительно
Теперь попробуем найти ген по его названию, возьмем MICB, заданный мне в предыдущих занятиях, страничка результатов поиска содержит 2 таблицы, одна отсортирована по типам данных, которые содержат в своем названии MICB, а вторая - по организмам, в которых есть данные с этим названием. Возьмем, например, гены человека с данным названием. По ссылке получаем табличку, где для каждой записи есть 4 типа данных, это описание, ID записи, ссылка на место в геноме и источник, для генов также есть ссылка на страничку с альтернативным сплацсингом. Также неожиданно наткнулся на страницу с роликами, помогающими освоить браузердругие браузеры
Геномный браузер NCBI мне понравился меньше, чем ансамбль. Во-первых дизайн, он не такой удобный как в ансамбле, и вообще очень минималистичный, нету тех всплывающих подсказок, объясняющих, что чего значит, во-вторы когда я попал на страницу результатов, я увидел вертикальный дизайн участка генома, и без подсказок я так и не смог с ним разобраться. В браузере UCSC дизайн - вообще адовый, Огромное количество параметров,в которых трудно разобраться, однако, в этом браузере мне очень понравился сервис VisiGene, позволяющий увидеть экспрессию гена по тканям, вот, неапример, экспрессия гена nkx2-2 у 10 дневного эмбриона мыши.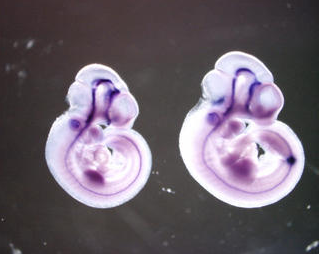 Браузер VEGA имеет такой же дизайн, как и ансамбль, но имеет меньшую фунциональность, так что описывать его не стоит.
Браузер VEGA имеет такой же дизайн, как и ансамбль, но имеет меньшую фунциональность, так что описывать его не стоит.
Я нашел в интернете еще один геномный браузер, им оказался браузер 1000 genomes, и специализируется он только на геноме человека. Он довольно простой в применении и с удобным интерфейсом, однако, имеет функциональность намного ниже того же ансамбля, но там собрано много секвенированных геномов человека, что может, например, определить эволюцию человека, или проследить генетические отклонения и особенности определенных народов.