Таксономия выбранных бактерий
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Bacillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Staphylococcus epidermidis | STAES | Firmicutes | Bacilli | Bacillales | Staphylococcaceae |
| Lactobacillus acidophilus | LACAC | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Lactobacillus delbrueckii | LACDA | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Enterococcus faecalis | ENTFA | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| Clostridium botulinum | CLOB1 | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Finegoldia magna | FINM2 | Firmicutes | Clostridia | Clostridiales | Clostridiales Family XI. Incertae Sedis |
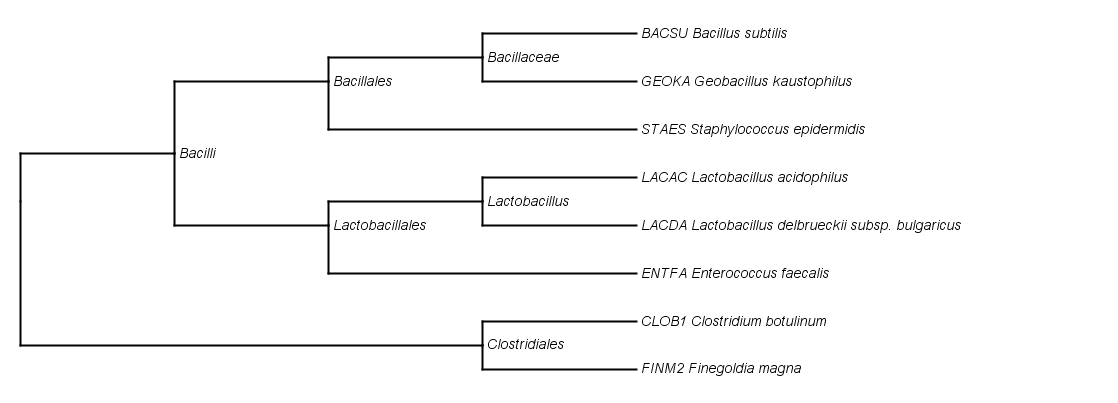
Я строил филогенетическое дерево в программе archaeopterix, которая умеет автоматически связывать мнемонические названия с таксономией, но как базу данных для таксономии она использует не NCBI taxonomy, а другой сервис, UniProt taxonomy Впрочем, никакой разицы в таксономии выбранных мной бактерий я не заметил, судя по всему, эти сервисы имеют одинаковую классификацию.
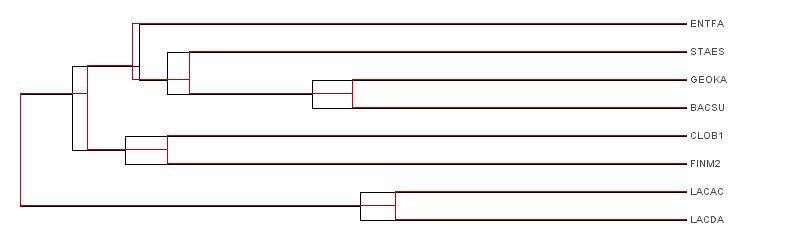
Нетривиальные ветви - следующие:
{ENTFA, STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC}
{ENTFA, STAES, GEOKA, BACSU, LACDA, LACAC}vs{CLOB1, FINM2}
{STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC, ENTFA}
{STAES, GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA}
{GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA, STAES}
Реконструкция по белку
Для реконструкции дерева я выбрал семейство белков Пептидил-тРНК гидролаза(PTH)Воспользовавшись расширеным поиском на сайте UniProt я нашел в БД SwissProt белки данного семейства для всех 8 бактерий, сервис UniProt предлагает выравнить их сразу же на сайте и выдает выравнивание в Fasta формате и дерево, очень удобно, заодно сравню то, что получилось на сайте uniprot с требованием в задании.
JalView выдал следующие выравнивание:
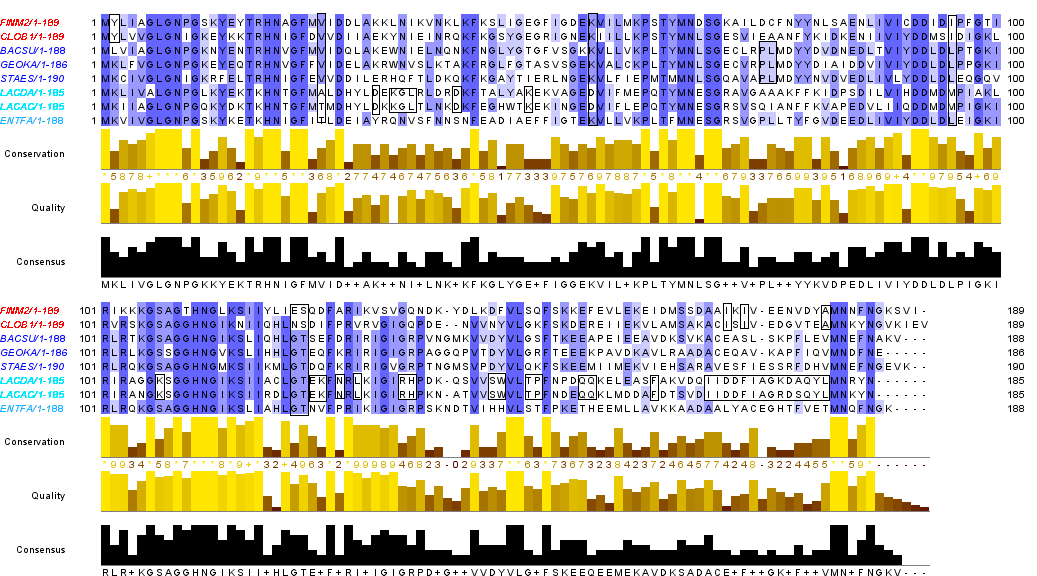 выравнивание JalView
выравнивание JalView
Поиск диагностических позиций
Попытаемся найти диагностические позиции выравнивания, для этого я раскрасил названия организмов по отрядам(и легко отличить окраску между классами). Их оказалось очень мало, многие аминокислоты характерные для клостридий, могут встретиться в одной из бацилл, да, и среди бацилл строго диагностических позиций почти нет. Скорее всего имел место горизонтальный перенос и, возможно гомоплозии, однако,очень интересны выделенные мной столбцы 2, где у клостридий встречается только Y, 93, где у всех бацилл, кроме лактобактерий - L и 122-123 c GT у всех бацилл , участок 167-180, а также выделенные мной участки поменьше по всему выравниванию очень характерны для лактобактерий и совершенно не похожи на таковой у всех остальных(хотя, возможно, это - просто 2 очень близких вида внутри лактобактерий и никаких диагностических позиций тут нет)Различные варианты реконструкции
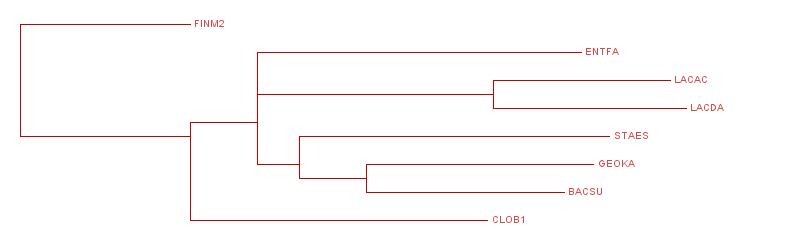
Реконструируем деревья всеми способами, доступными в JalView:Average distance using % identity
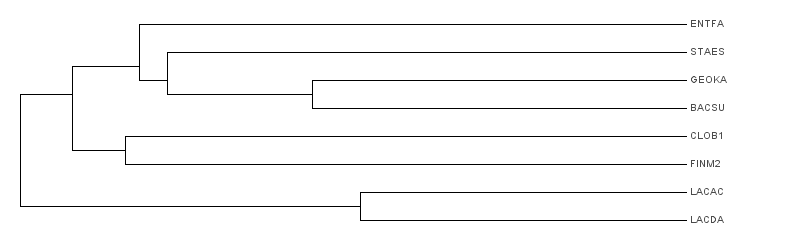
Average distance using BLOSUM62
Деревья очень похожи, вот - скомпанованное изображение этих двух деревьев, видно, что не только все ветви одинаковы, но и расстояния отличаются незначительно
На дереве - следующие нетривиальные ветви:
{ENTFA, STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC}
{ENTFA, STAES, GEOKA, BACSU, LACDA, LACAC}vs{CLOB1, FINM2}
{ENTFA, STAES, GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2}
{STAES, GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA}
{GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA, STAES}
Совпадают 4 из 5 разбиений(подчеркнуты)
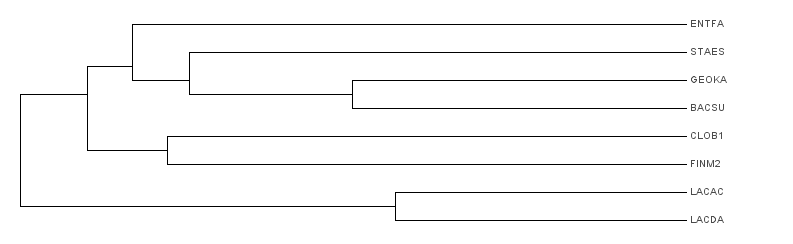
Neighbour joining using % identity
Neighbour joining using BLOSUM62
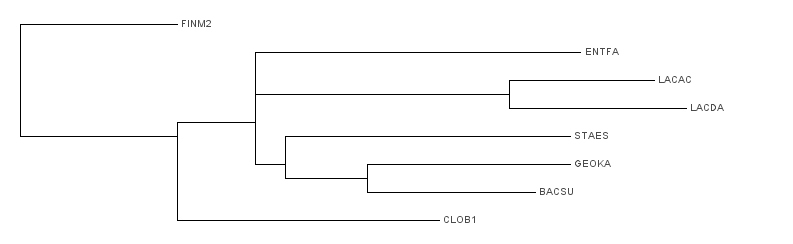
Деревья также очень похожи, вот - скомпанованное изображение этих двух деревьев
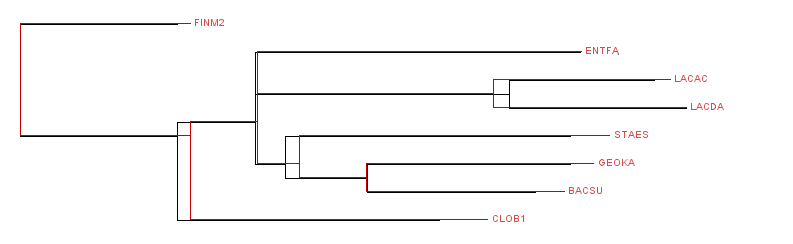
{ENTFA, STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC}
{ENTFA, STAES, GEOKA, BACSU, LACDA, LACAC}vs{CLOB1, FINM2}
{GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA, STAES}
{STAES, GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA}
{STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC, ENTFA}
Дерево получилось небинарным,(как я потом уточнил, все-таки бинарным, но ветвь, противопоставляющая ENTFA, LACDA и LACAC остальным организмам имеет длину 0) и тогда все ветви реконструированы правильно, хотя и на грани.
Дерево построенное методом Maximum Parsimony
Программа MEGA, используя выравнивание в формате fasta построила дерево по методу Maximum Parsimony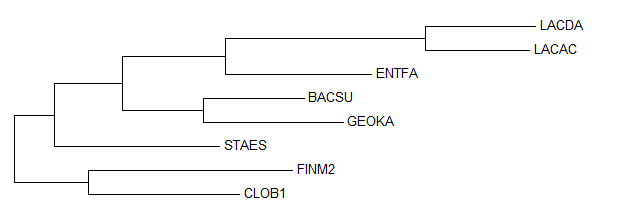
Удобней, наверное, будет выровнять все ветви по длине
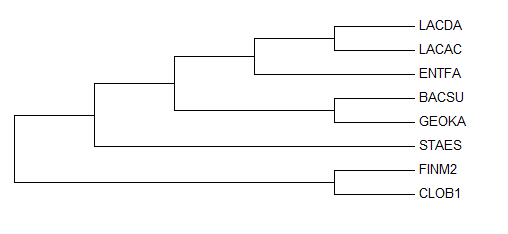
Нетривиальные ветви("правильные" разбиения подчеркнуты):
{ENTFA, STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC}
{ENTFA, STAES, GEOKA, BACSU, LACDA, LACAC}vs{CLOB1, FINM2}
{STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC, ENTFA}
{GEOKA, BACSU, LACDA, LACAC, ENTFA}vs{CLOB1, FINM2, STAES}
{GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA, STAES}