1 - Построение дерева по нуклеотидным последовательностям
С помощью EMBL я, проанализировав(глядя в экран на аннотации к геному и ища тэги 16s rRNA) геномы исследуемых бактерий нашел в каждом по 16s рРНК и вырезал программой seqret.| Название бактерии | Мнемоника | AC записи EMBL | Начало | Конец | цепь |
| Bacillus subtilis | BACSU | AL009126 | 30279 | 31832 | + |
| Enterococcus faecalis | ENTFA | AE016830 | 248466 | 249987 | + |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421 | 11973 | + |
| Lactobacillus delbrueckii | LACDA | CR954253 | 45160 | 46720 | + |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255 | 60826 | + |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006 | 1599559 | - |
| Clostridium botulinum | CLOB1 | CP000726 | 9282 | 10783 | + |
| Finegoldia magna | FINM2 | AP008971 | 197837 | 199361 | + |
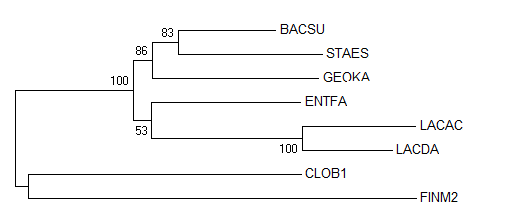
А вот, что выдал бутстреп в виде консенсусного дерева(топология получилась точно такая же)
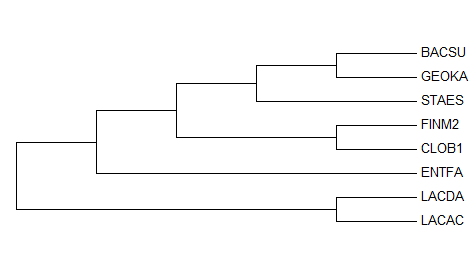
Топология дерева правильная(правильные ветви, как всегда, подчеркнуты):
{ENTFA, STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC}
{ENTFA, STAES, GEOKA, BACSU, LACDA, LACAC}vs{CLOB1, FINM2}
{STAES, GEOKA, BACSU, CLOB1, FINM2}vs{LACDA, LACAC, ENTFA}
{STAES, GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA}
{GEOKA, BACSU}vs{LACDA, LACAC, CLOB1, FINM2, ENTFA, STAES}
2 - построение и анализ дерева, содержащего паралоги
Итак, взяв в свисспроте последовательность белка CLPX_BACSU, и (преобразовав в базу данных функцией makeblastdb) использовав в качестве базы данных proteo.fasta я нашел гомологи (с помощью функции blastp) этого белка в своих организмах, затем оттуда взял id белков и с помощью сервиса retrieve на юнипроте вытащил последовательности белков, и там же на сайте с помощью алгоритма Clustalo их выравнял и отправил в программу MEGA, где построил дерево методом Minimal Evolution
Итак, найдем несколько пар ортологов(их тут очень много, но выберем 3, бросающиеся в глаза):
CLPX_BACSU и CLPX_GEOKA
HSLU_GEOKA и HSLU_STAES
HSLU_LACDA и HSLU_LACAC
И несколько пар паралогов(которых тоже очень много):
B0S3J0_FINM2 и B0S3X9_FINM2
CLPX_BACSU и CLPY_BACSU (туда же CLPC_BACSU и CLPE_BACSU)
Q5L436_GEOKA и CLPX_GEOKA