Определение подсистемы и расшифровка записи GO
идентификатор GO у моего белка только один - GO:0005524, означающий, что это ATP-связывающий белок, но это и не удивительно, Evidence code моего белка - лишь IEA, так что про системы, к которым принадлежит этот белок сказать ничего невозможно. Ближайшая находка, найденная бластом по SwissProt с E-value 2.0*10-51 - белок с Accession Q9R2W4 прекрасно аннотирован в GO, будем смотреть на него и по гомологии говорить о данном мне белке.Таблица расшифровки идентификаторов GO белка Q9R2W4
| GO ID | тип | значение | описание |
| GO:0006810 | P | transport | белок, облегчающий транспорт |
| GO:0008152 | P | metabolic process | процесс, приводящий к росту клеток |
| GO:0009405 | P | pathogenesis | цитотоксичная акивность |
| GO:0000166 | F | nucleotide binding | связывание нуклеотидов |
| GO:0005524 | F | ATP binding | связывание АТФ |
| GO:0017111 | F | nucleotide-triphosphatase activity | нуклеотидтрифосфатазная активность |
| GO:0005886 | C | plasma membrane | локализуется в плазматической мембране |
| GO:0016020 | C | membrane | мембранный белок |
Роль в подсистеме
Этот белок относится к секреторной системе типа IV, которая представляет собой иглу на поверхности бактерии, пронизывающую клетку-мишень и секретируемые вещества прямо из цитоплазмы атакующей клетки. Используется для секреции ДНК, белков и комплексов ДНК-белок. Система состоит примерно из 20 белков, не у всех из них известны функции. (источник - статьи, данные в задании)Согласно пункту General annotation в описании SwissProt белок может гидролизовать АТФ для сборки секреторной системы и быть транспортом для субстрата.
Карта окрестностей ортологов
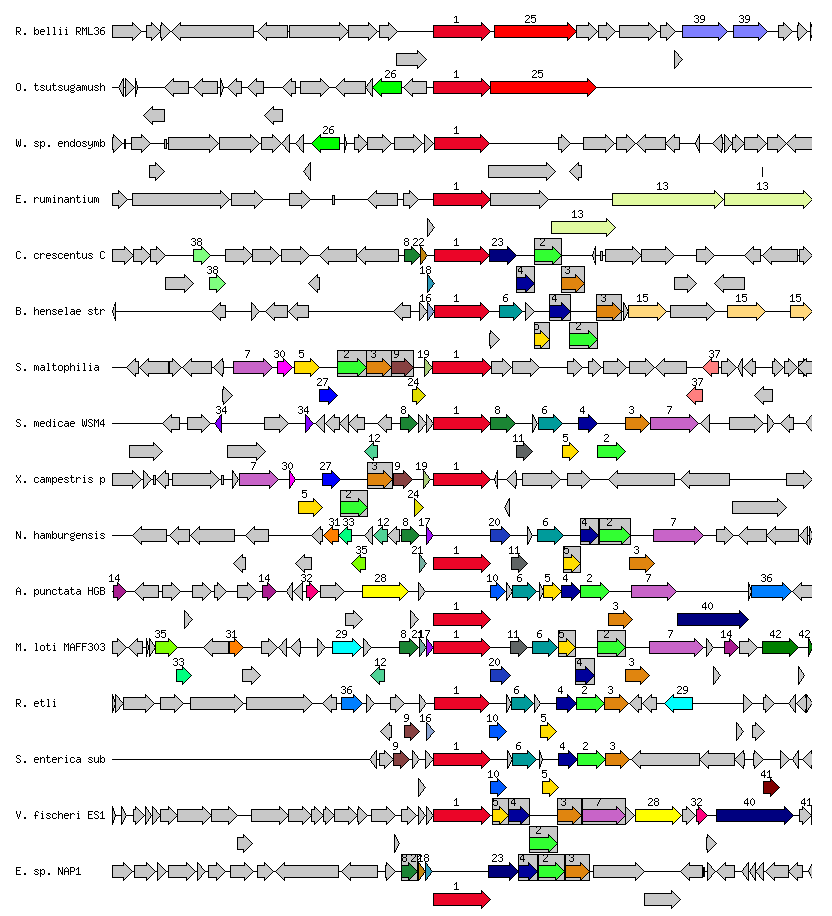
Карта получена на сайте The Seed Viewer со следующими значениями:
Region size - 30000 bp
Number of regions - 18(с последующим отбраковыванием слишком похожих геномов)
Collapse close genomes
E-value cut off = 1e-15
Выравнивание ортологов в JalView
Таблица в формате ods
у меня таблица читается неверно, если открыть ее непосредственно отсюда, но корректно, если скачать с сервера с данными из БД seed(с дополнительным столбцом, где отмечены гены из исследуемой подсистемы) и сводной таблицей с группами ортологов во втором листе
Выводы
a - В существенной части организмов в выдаче присутствуют похожие белки(особенно много белков из групп 2-8), расположенные в похожей последовательности(особенно характерно, например, для белков из групп 8, 1 10, 6, 5, 2, 3 и 7) и в одной ориентации, но причина этого становится понятна, когда несмотря на то что в поле function написано, что они относятся к секреторной системе IV типа, подсистема определяется, как pVir plasmid of Campylobacter, то есть плазмида.b - Во-первых часто гены, колокализованные с исследуемым действительно относятся к той же подсистеме, но даже, когда соответствующей отметки в столбце SS нет, часто(слишком часто, чтобы вставлять сюда примеры) в поле function можно найти упоминание о системе секреции IV типа (группы 2-7, 11, 20, 21 и пр.) или схожие функции(группа 8 - прекурсор белка, лизирующего муреин, много транспортных белков)
c - В основном колокализация распространяется на небольшие области, что хорошо видно из изображения с фрагментами геномов, при области в 30000 пн на краях колокализованных генов довольно мало, так что значение по умолчанию в поле размера 16000 пн кажется довольно разумным.
d - особого ничего еще сказать не могу, разве что, огромное число генов, относящихся к изучаемой подсистеме не показаны, как относящиеся к ней, что говорит о плохой аннотации. Вообще, у многих белков подсистемы не указаны вообще, а у еще большего количества не указана даже функция, что говорит о плохой аннотации и в некотором роде сырости БД seed
e - Когда речь идет о колокализации, у меня перед глазами встают хрестоматийные примеры Lac-оперона и Trp-оперона. У бактерий довольно часто в одной синтезируемой мРНК содержатся несколько генов, что, безусловно, упрощает регуляцию экспрессии, а еще у бактерий есть плазмиды, которые несут гены одной подсистемы и, безусловно, эти гены оказываются колоколизованными.